+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Poliovirus type 2 (strain MEF-1) virus-like particle in complex with capsid binder compound 17 | |||||||||
 マップデータ マップデータ | Local resolution scaled final map from RELION. Sharpened with an ad-hoc B-factor of -15A^2. Contour level determined in UCSF ChimeraX. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Capsid protein / VIRUS LIKE PARTICLE / inhibitor / complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / receptor-mediated endocytosis of virus by host cell / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / ribonucleoside triphosphate phosphatase activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / receptor-mediated endocytosis of virus by host cell / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / ribonucleoside triphosphate phosphatase activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / DNA replication / RNA helicase activity / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / host cell nucleus / structural molecule activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human poliovirus 2 (ポリオウイルス) Human poliovirus 2 (ポリオウイルス) | |||||||||
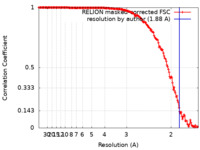
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 1.88 Å | |||||||||
 データ登録者 データ登録者 | Bahar MW / Fry EE / Stuart DI | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2022 ジャーナル: Commun Biol / 年: 2022タイトル: A conserved glutathione binding site in poliovirus is a target for antivirals and vaccine stabilisation. 著者: Mohammad W Bahar / Veronica Nasta / Helen Fox / Lee Sherry / Keith Grehan / Claudine Porta / Andrew J Macadam / Nicola J Stonehouse / David J Rowlands / Elizabeth E Fry / David I Stuart /   要旨: Strategies to prevent the recurrence of poliovirus (PV) after eradication may utilise non-infectious, recombinant virus-like particle (VLP) vaccines. Despite clear advantages over inactivated or ...Strategies to prevent the recurrence of poliovirus (PV) after eradication may utilise non-infectious, recombinant virus-like particle (VLP) vaccines. Despite clear advantages over inactivated or attenuated virus vaccines, instability of VLPs can compromise their immunogenicity. Glutathione (GSH), an important cellular reducing agent, is a crucial co-factor for the morphogenesis of enteroviruses, including PV. We report cryo-EM structures of GSH bound to PV serotype 3 VLPs showing that it can enhance particle stability. GSH binds the positively charged pocket at the interprotomer interface shown recently to bind GSH in enterovirus F3 and putative antiviral benzene sulphonamide compounds in other enteroviruses. We show, using high-resolution cryo-EM, the binding of a benzene sulphonamide compound with a PV serotype 2 VLP, consistent with antiviral activity through over-stabilizing the interprotomer pocket, preventing the capsid rearrangements necessary for viral infection. Collectively, these results suggest GSH or an analogous tight-binding antiviral offers the potential for stabilizing VLP vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15727.map.gz emd_15727.map.gz | 210 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15727-v30.xml emd-15727-v30.xml emd-15727.xml emd-15727.xml | 25.9 KB 25.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15727_fsc.xml emd_15727_fsc.xml | 15.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15727.png emd_15727.png | 98.8 KB | ||
| Filedesc metadata |  emd-15727.cif.gz emd-15727.cif.gz | 8.1 KB | ||
| その他 |  emd_15727_half_map_1.map.gz emd_15727_half_map_1.map.gz emd_15727_half_map_2.map.gz emd_15727_half_map_2.map.gz | 276.4 MB 276.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15727 http://ftp.pdbj.org/pub/emdb/structures/EMD-15727 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15727 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15727 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15727_validation.pdf.gz emd_15727_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15727_full_validation.pdf.gz emd_15727_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_15727_validation.xml.gz emd_15727_validation.xml.gz | 23.7 KB | 表示 | |
| CIF形式データ |  emd_15727_validation.cif.gz emd_15727_validation.cif.gz | 31.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15727 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15727 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15727 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15727 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15727.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15727.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution scaled final map from RELION. Sharpened with an ad-hoc B-factor of -15A^2. Contour level determined in UCSF ChimeraX. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.829 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map from final refinement in RELION. Contour...
| ファイル | emd_15727_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map from final refinement in RELION. Contour level determined in UCSF ChimeraX. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map from final refinement in RELION. Contour...
| ファイル | emd_15727_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map from final refinement in RELION. Contour level determined in UCSF ChimeraX. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human poliovirus 2
| 全体 | 名称:  Human poliovirus 2 (ポリオウイルス) Human poliovirus 2 (ポリオウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human poliovirus 2
| 超分子 | 名称: Human poliovirus 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Recombinantly expressed virus-like particle of PV2 (strain MEF-1) NCBI-ID: 12083 / 生物種: Human poliovirus 2 / Sci species strain: MEF-1 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 5.81 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Virus shell 1 / 直径: 310.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein, VP1
| 分子 | 名称: Capsid protein, VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human poliovirus 2 (ポリオウイルス) / 株: MEF-1 Human poliovirus 2 (ポリオウイルス) / 株: MEF-1 |
| 分子量 | 理論値: 33.111207 KDa |
| 組換発現 | 生物種:  Mesocricetus auratus (ネズミ) Mesocricetus auratus (ネズミ) |
| 配列 | 文字列: GLGDLIEGVV EGVTRNALTP LTPANNLPDT QSSGPAHSKE TPALTAVETG ATNPLVPSDT VQTRHVIQKR TRSESTVESF FARGACVAI IEVDNDAPTK RASKLFSVWK ITYKDTVQLR RKLEFFTYSR FDMEFTFVVT SNYTDANNGH ALNQVYQIMY I PPGAPIPG ...文字列: GLGDLIEGVV EGVTRNALTP LTPANNLPDT QSSGPAHSKE TPALTAVETG ATNPLVPSDT VQTRHVIQKR TRSESTVESF FARGACVAI IEVDNDAPTK RASKLFSVWK ITYKDTVQLR RKLEFFTYSR FDMEFTFVVT SNYTDANNGH ALNQVYQIMY I PPGAPIPG KWNDYTWQTS SNPSVFYTYG APPARISVPY VGIANAYSHF YDGFAKVPLA GQASTEGDSL YGAASLNDFG SL AVRVVND HNPTKLTSKI RVYMKPKHVR VWCPRPPRAV PYYGPGVDYK DGLAPLPEKG LTTY UniProtKB: Genome polyprotein |
-分子 #2: Capsid protein, VP0
| 分子 | 名称: Capsid protein, VP0 / タイプ: protein_or_peptide / ID: 2 / 詳細: Sequence is given for the VP0 polypeptide. / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human poliovirus 2 (ポリオウイルス) / 株: MEF-1 Human poliovirus 2 (ポリオウイルス) / 株: MEF-1 |
| 分子量 | 理論値: 37.456855 KDa |
| 組換発現 | 生物種:  Mesocricetus auratus (ネズミ) Mesocricetus auratus (ネズミ) |
| 配列 | 文字列: MGAQVSSQKV GAHENSNRAY GGSTINYTTI NYYRDSASNA ASKQDFAQDP SKFTEPIKDV LIKTAPTLNS PNIEACGYSD RVMQLTLGN STITTQEAAN SVVAYGRWPE YIKDSEANPV DQPTEPDVAA CRFYTLDTVT WRKESRGWWW KLPDALKDMG L FGQNMFYH ...文字列: MGAQVSSQKV GAHENSNRAY GGSTINYTTI NYYRDSASNA ASKQDFAQDP SKFTEPIKDV LIKTAPTLNS PNIEACGYSD RVMQLTLGN STITTQEAAN SVVAYGRWPE YIKDSEANPV DQPTEPDVAA CRFYTLDTVT WRKESRGWWW KLPDALKDMG L FGQNMFYH YLGRAGYTVH VQCNASKFHQ GALGVFAVPE MCLAGDSTTH MFTKYENANP GEKGGEFKGS FTLDTNATNP AR NFCPVDY LFGSGVLAGN AFVYPHQIIN LRTNNCATLV LPYVNSLSID SMTKHNNWGI AILPLAPLDF ATESSTEIPI TLT IAPMCC EFNGLRNITV PRTQ UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein, VP3
| 分子 | 名称: Capsid protein, VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human poliovirus 2 (ポリオウイルス) / 株: MEF-1 Human poliovirus 2 (ポリオウイルス) / 株: MEF-1 |
| 分子量 | 理論値: 26.472293 KDa |
| 組換発現 | 生物種:  Mesocricetus auratus (ネズミ) Mesocricetus auratus (ネズミ) |
| 配列 | 文字列: GLPVLNTPGS NQYLTADNYQ SPCAIPEFDV TPPIDIPGEV RNMMELAEID TMIPLNLTNQ RKNTMDMYRV ELNDAAHSDT PILCLSLSP ASDPRLAHTM LGEILNYYTH WAGSLKFTFL FCGSMMATGK LLVSYAPPGA EAPKSRKEAM LGTHVIWDIG L QSSCTMVV ...文字列: GLPVLNTPGS NQYLTADNYQ SPCAIPEFDV TPPIDIPGEV RNMMELAEID TMIPLNLTNQ RKNTMDMYRV ELNDAAHSDT PILCLSLSP ASDPRLAHTM LGEILNYYTH WAGSLKFTFL FCGSMMATGK LLVSYAPPGA EAPKSRKEAM LGTHVIWDIG L QSSCTMVV PWISNTTYRQ TINDSFTEGG YISMFYQTRV VVPLSTPRKM DILGFVSACN DFSVRLLRDT THISQEAMPQ UniProtKB: Genome polyprotein |
-分子 #4: SPHINGOSINE
| 分子 | 名称: SPHINGOSINE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: SPH |
|---|---|
| 分子量 | 理論値: 299.492 Da |
| Chemical component information |  ChemComp-SPH: |
-分子 #5: 4-[[4-[1,3-bis(oxidanylidene)isoindol-2-yl]phenyl]sulfonylamino]b...
| 分子 | 名称: 4-[[4-[1,3-bis(oxidanylidene)isoindol-2-yl]phenyl]sulfonylamino]benzoic acid タイプ: ligand / ID: 5 / コピー数: 1 / 式: FHK |
|---|---|
| 分子量 | 理論値: 422.411 Da |
| Chemical component information | 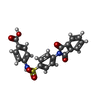 ChemComp-FHK: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 262 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.75 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 / 構成要素:
詳細: 1 x DPBS was made from tissue culture grade Dulbecco's Phosphate Buffered Saline (Sigma-Aldrich). 20 mM EDTA was prepared from fresh stocks of buffered EDTA. | ||||||
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR 詳細: The specific type of grid used was Ultra-thin carbon support film, 3nm - on lacey carbon AGS187-4 from Agar Scientific. | ||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Single blotting with 3.5ul of sample. Four second blot time and -17 blot force on the vitrobot.. | ||||||
| 詳細 | Recombinant VLP of PV2 mixed with capsid inhibitor compound 17 at a molar ratio of 1:2500. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 4750 / 平均露光時間: 1.7 sec. / 平均電子線量: 34.68 e/Å2 詳細: Images were collected in electron counting mode with a calibrated pixel size of 0.829 A/pixel. Super-resolution pixel size 0.4145 A/pixel. Movies were collected in 50 frames. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 60314 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Initial model was rigid body fitted using UCSF chimera and Coot. Global minimization and B-factor refinement was performed in real space using phenix_real.space.refine. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-8ayz: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)