+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of ASCC3 in complex with ASC1 | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | helicase / zinc finger / activator / DNA binding / DNA BINDING PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ALKBH3 mediated reversal of alkylation damage / activating signal cointegrator 1 complex / regulation of myoblast differentiation / DNA alkylation repair / ribosome disassembly / ribosome-associated ubiquitin-dependent protein catabolic process / DNA duplex unwinding / histone acetyltransferase binding / ubiquitin-like protein ligase binding / 3'-5' DNA helicase activity ...ALKBH3 mediated reversal of alkylation damage / activating signal cointegrator 1 complex / regulation of myoblast differentiation / DNA alkylation repair / ribosome disassembly / ribosome-associated ubiquitin-dependent protein catabolic process / DNA duplex unwinding / histone acetyltransferase binding / ubiquitin-like protein ligase binding / 3'-5' DNA helicase activity / estrogen receptor signaling pathway / rescue of stalled ribosome / nuclear estrogen receptor binding / nuclear receptor binding / neuromuscular junction / DNA helicase / protease binding / cell population proliferation / transcription coactivator activity / nuclear body / nuclear speck / centrosome / regulation of DNA-templated transcription / protein kinase binding / positive regulation of DNA-templated transcription / protein-containing complex / RNA binding / zinc ion binding / nucleoplasm / ATP binding / membrane / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
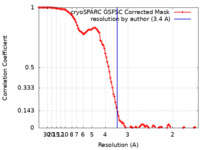
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Jia J / Hilal T / Loll B / Wahl MC | ||||||||||||||||||
| 資金援助 |  ドイツ, 5件 ドイツ, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structure of ASCC3 in complex with ASC1 著者: Jia J / Hilal T / Loll B / Wahl MC | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15521.map.gz emd_15521.map.gz | 22.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15521-v30.xml emd-15521-v30.xml emd-15521.xml emd-15521.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15521_fsc.xml emd_15521_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15521.png emd_15521.png | 56.8 KB | ||
| Filedesc metadata |  emd-15521.cif.gz emd-15521.cif.gz | 7.3 KB | ||
| その他 |  emd_15521_half_map_1.map.gz emd_15521_half_map_1.map.gz emd_15521_half_map_2.map.gz emd_15521_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15521 http://ftp.pdbj.org/pub/emdb/structures/EMD-15521 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15521 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15521 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15521_validation.pdf.gz emd_15521_validation.pdf.gz | 766 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15521_full_validation.pdf.gz emd_15521_full_validation.pdf.gz | 765.5 KB | 表示 | |
| XML形式データ |  emd_15521_validation.xml.gz emd_15521_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_15521_validation.cif.gz emd_15521_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15521 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15521 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15521 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15521 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8alzMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15521.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15521.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_15521_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15521_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ASCC3-ASC1
| 全体 | 名称: ASCC3-ASC1 |
|---|---|
| 要素 |
|
-超分子 #1: ASCC3-ASC1
| 超分子 | 名称: ASCC3-ASC1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Protein complex was produced in baculovirus-insect system. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 270 KDa |
-分子 #1: Activating signal cointegrator 1
| 分子 | 名称: Activating signal cointegrator 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 66.650656 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GAEFMAVAGA VSGEPLVHWC TQQLRKTFGL DVSEEIIQYV LSIESAEEIR EYVTDLLQGN EGKKGQFIEE LITKWQKNDQ ELISDPLQQ CFKKDEILDG QKSGDHLKRG RKKGRNRQEV PAFTEPDTTA EVKTPFDLAK AQENSNSVKK KTKFVNLYTR E GQDRLAVL ...文字列: GAEFMAVAGA VSGEPLVHWC TQQLRKTFGL DVSEEIIQYV LSIESAEEIR EYVTDLLQGN EGKKGQFIEE LITKWQKNDQ ELISDPLQQ CFKKDEILDG QKSGDHLKRG RKKGRNRQEV PAFTEPDTTA EVKTPFDLAK AQENSNSVKK KTKFVNLYTR E GQDRLAVL LPGRHPCDCL GQKHKLINNC LICGRIVCEQ EGSGPCLFCG TLVCTHEEQD ILQRDSNKSQ KLLKKLMSGV EN SGKVDIS TKDLLPHQEL RIKSGLEKAI KHKDKLLEFD RTSIRRTQVI DDESDYFASD SNQWLSKLER ETLQKREEEL REL RHASRL SKKVTIDFAG RKILEEENSL AEYHSRLDET IQAIANGTLN QPLTKLDRSS EEPLGVLVNP NMYQSPPQWV DHTG AASQK KAFRSSGFGL EFNSFQHQLR IQDQEFQEGF DGGWCLSVHQ PWASLLVRGI KRVEGRSWYT PHRGRLWIAA TAKKP SPQE VSELQATYRL LRGKDVEFPN DYPSGCLLGC VDLIDCLSQK QFKEQFPDIS QESDSPFVFI CKNPQEMVVK FPIKGN PKI WKLDSKIHQG AKKGLMKQNK AV UniProtKB: Activating signal cointegrator 1 |
-分子 #2: Activating signal cointegrator 1 complex subunit 3
| 分子 | 名称: Activating signal cointegrator 1 complex subunit 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 206.276094 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GAEFHYPHVY DSQAEAMKTS AFIAGAKMIL PEGIQRENNK LYEEVRIPYS EPMPLSFEEK PVYIQDLDEI GQLAFKGMKR LNRIQSIVF ETAYNTNENM LICAPTGAGK TNIAMLTVLH EIRQHFQQGV IKKNEFKIVY VAPMKALAAE MTDYFSRRLE P LGIIVKEL ...文字列: GAEFHYPHVY DSQAEAMKTS AFIAGAKMIL PEGIQRENNK LYEEVRIPYS EPMPLSFEEK PVYIQDLDEI GQLAFKGMKR LNRIQSIVF ETAYNTNENM LICAPTGAGK TNIAMLTVLH EIRQHFQQGV IKKNEFKIVY VAPMKALAAE MTDYFSRRLE P LGIIVKEL TGDMQLSKSE ILRTQMLVTT PEKWDVVTRK SVGDVALSQI VRLLILDEVH LLHEDRGPVL ESIVARTLRQ VE STQSMIR ILGLSATLPN YLDVATFLHV NPYIGLFFFD GRFRPVPLGQ TFLGIKCANK MQQLNNMDEV CYENVLKQVK AGH QVMVFV HARNATVRTA MSLIERAKNC GHIPFFFPTQ GHDYVLAEKQ VQRSRNKQVR ELFPDGFSIH HAGMLRQDRN LVEN LFSNG HIKVLVCTAT LAWGVNLPAH AVIIKGTQIY AAKRGSFVDL GILDVMQIFG RAGRPQFDKF GEGIIITTHD KLSHY LTLL TQRNPIESQF LESLADNLNA EIALGTVTNV EEAVKWISYT YLYVRMRANP LAYGISHKAY QIDPTLRKHR EQLVIE VGR KLDKAQMIRF EERTGYFSST DLGRTASHYY IKYNTIETFN ELFDAHKTEG DIFAIVSKAE EFDQIKVREE EIEELDT LL SNFCELSTPG GVENSYGKIN ILLQTYISRG EMDSFSLISD SAYVAQNAAR IVRALFEIAL RKRWPTMTYR LLNLSKVI D KRLWGWASPL RQFSILPPHI LTRLEEKKLT VDKLKDMRKD EIGHILHHVN IGLKVKQCVH QIPSVMMEAS IQPITRTVL RVTLSIYADF TWNDQVHGTV GEPWWIWVED PTNDHIYHSE YFLALKKQVI SKEAQLLVFT IPIFEPLPSQ YYIRAVSDRW LGAEAVCII NFQHLILPER HPPHTELLDL QPLPITALGC KAYEALYNFS HFNPVQTQIF HTLYHTDCNV LLGAPTGSGK T VAAELAIF RVFNKYPTSK AVYIAPLKAL VRERMDDWKV RIEEKLGKKV IELTGDVTPD MKSIAKADLI VTTPEKWDGV SR SWQNRNY VQQVTILIID EIHLLGEERG PVLEVIVSRT NFISSHTEKP VRIVGLSTAL ANARDLADWL NIKQMGLFNF RPS VRPVPL EVHIQGFPGQ HYCPRMASMN KPAFQAIRSH SPAKPVLIFV SSRRQTRLTA LELIAFLATE EDPKQWLNMD EREM ENIIA TVRDSNLKLT LAFGIGMHHA GLHERDRKTV EELFVNCKVQ VLIATSTLAW GVNFPAHLVI IKGTEYYDGK TRRYV DFPI TDVLQMMGRA GRPQFDDQGK AVILVHDIKK DFYKKFLYEP FPVESSLLGV LSDHLNAEIA GGTITSKQDA LDYITW TYF FRRLIMNPSY YNLGDVSHDS VNKFLSHLIE KSLIELELSY CIEIGEDNRS IEPLTYGRIA SYYYLKHQTV KMFKDRL KP ECSTEELLSI LSDAEEYTDL PVRHNEDHMN SELAKCLPIE SNPHSFDSPH TKAHLLLQAH LSRAMLPCPD YDTDTKTV L DQALRVCQAM LDVAANQGWL VTVLNITNLI QMVIQGRWLK DSSLLTLPNI ENHHLHLFKK WKPIMKGPHA RGRTSIESL PELIHACGGK DHVFSSMVES ELHAAKTKQA WNFLSHLPVI NVGISVKGSW DDLVEGHNEL SVSTLTADKR DDNKWIKLHA DQEYVLQVS LQRVHFGFHK GKPESCAVTP RFPKSKDEGW FLILGEVDKR ELIALKRVGY IRNHHVASLS FYTPEIPGRY I YTLYFMSD CYLGLDQQYD IYLNVTQASL SAQVNTKVSD SLTDLALK UniProtKB: Activating signal cointegrator 1 complex subunit 3 |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.15 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solutions were made fresh from concentrated to avoid microbial contamination. | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 6267 / 平均露光時間: 40.57 sec. / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)