+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of crescentin filaments (stutter mutant, C2 symmetry and large box) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cytoskeleton / cell shape / intermediate filaments / coiled coil / assembly / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 | :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Caulobacter vibrioides (バクテリア) / Caulobacter vibrioides (バクテリア) /  Camelidae (哺乳類) Camelidae (哺乳類) | |||||||||
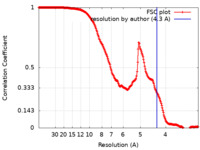
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Lowe J | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Filament structure and subcellular organization of the bacterial intermediate filament-like protein crescentin. 著者: Yue Liu / Fusinita van den Ent / Jan Löwe /  要旨: The protein crescentin is required for the crescent shape of the freshwater bacterium (). Crescentin forms a filamentous structure on the inner, concave side of the curved cells. It shares features ...The protein crescentin is required for the crescent shape of the freshwater bacterium (). Crescentin forms a filamentous structure on the inner, concave side of the curved cells. It shares features with eukaryotic intermediate filament (IF) proteins, including the formation of static filaments based on long and parallel coiled coils, the protein's length, structural roles in cell and organelle shape determination and the presence of a coiled coil discontinuity called the "stutter." Here, we have used electron cryomicroscopy (cryo-EM) to determine the structure of the full-length protein and its filament, exploiting a crescentin-specific nanobody. The filament is formed by two strands, related by twofold symmetry, that each consist of two dimers, resulting in an octameric assembly. Crescentin subunits form longitudinal contacts head-to-head and tail-to-tail, making the entire filament non-polar. Using in vivo site-directed cysteine cross-linking, we demonstrated that contacts observed in the in vitro filament structure exist in cells. Electron cryotomography (cryo-ET) of cells expressing crescentin showed filaments on the concave side of the curved cells, close to the inner membrane, where they form a band. When comparing with current models of IF proteins and their filaments, which are also built from parallel coiled coil dimers and lack overall polarity, it emerges that IF proteins form head-to-tail longitudinal contacts in contrast to crescentin and hence several inter-dimer contacts in IFs have no equivalents in crescentin filaments. Our work supports the idea that intermediate filament-like proteins achieve their shared polymerization and mechanical properties through a variety of filament architectures. #1:  ジャーナル: to be published ジャーナル: to be publishedタイトル: Assembly and organization of the bacterial intermediate filament-like protein crescentin 著者: Liu Y / Lowe J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15476.map.gz emd_15476.map.gz | 735.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15476-v30.xml emd-15476-v30.xml emd-15476.xml emd-15476.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15476_fsc.xml emd_15476_fsc.xml | 21 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15476.png emd_15476.png | 29.5 KB | ||
| マスクデータ |  emd_15476_msk_1.map emd_15476_msk_1.map | 824 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15476.cif.gz emd-15476.cif.gz | 6.5 KB | ||
| その他 |  emd_15476_half_map_1.map.gz emd_15476_half_map_1.map.gz emd_15476_half_map_2.map.gz emd_15476_half_map_2.map.gz | 765.7 MB 765.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15476 http://ftp.pdbj.org/pub/emdb/structures/EMD-15476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15476 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15476_validation.pdf.gz emd_15476_validation.pdf.gz | 678.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15476_full_validation.pdf.gz emd_15476_full_validation.pdf.gz | 677.8 KB | 表示 | |
| XML形式データ |  emd_15476_validation.xml.gz emd_15476_validation.xml.gz | 28.1 KB | 表示 | |
| CIF形式データ |  emd_15476_validation.cif.gz emd_15476_validation.cif.gz | 37.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15476 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15476 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15476 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15476 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ajbMC  8afeC  8afhC  8aflC  8afmC  8ahlC  8aiaC  8aixC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15476.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15476.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.59 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15476_msk_1.map emd_15476_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15476_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15476_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Crescentin filament in complex with megabody MB13
| 全体 | 名称: Crescentin filament in complex with megabody MB13 |
|---|---|
| 要素 |
|
-超分子 #1: Crescentin filament in complex with megabody MB13
| 超分子 | 名称: Crescentin filament in complex with megabody MB13 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Crescentin
| 超分子 | 名称: Crescentin / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides (バクテリア) Caulobacter vibrioides (バクテリア) |
-超分子 #3: Crescentin-specific megabody MB13
| 超分子 | 名称: Crescentin-specific megabody MB13 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Camelidae (哺乳類) Camelidae (哺乳類) |
-分子 #1: Crescentin
| 分子 | 名称: Crescentin / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides (バクテリア) Caulobacter vibrioides (バクテリア) |
| 分子量 | 理論値: 50.177816 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRLLSKNSRE TKNGKPTVLG DEARAEAMQH QIESTQAIGQ RYETIHGGLD SIGRVMEHLK AIEPLIAEIR GPVSQEFEAR RAEHAELIA VRANLDQAQR QIALIQAEER EVSARLAAAE TALGESDARR QTQDAALEDN ALEIDRLRNA LLQSDLKVSS L DASLRDAT ...文字列: MRLLSKNSRE TKNGKPTVLG DEARAEAMQH QIESTQAIGQ RYETIHGGLD SIGRVMEHLK AIEPLIAEIR GPVSQEFEAR RAEHAELIA VRANLDQAQR QIALIQAEER EVSARLAAAE TALGESDARR QTQDAALEDN ALEIDRLRNA LLQSDLKVSS L DASLRDAT ARIEHLVQDV EGLRVQAQDI DARRGDAEAA LARANQDNAL LGEEAATLKK RVDQAGLDLA RLSRIETDLE AQ LAAERAR VQAVENALAA HQADSGRTIR GLESQVEANR AEISALQTRL ETATGRADKL EEMNGQISAR LADSSAQQKA VER RAGDLN VALERALDRI RALEEEADGL RQRHAGVDTA RATAIERADQ LAKSAVAQEK ALKRAEERAQ QLRARLDAMQ EAQD QVRRD SATHEAKIAE LQATIERLTS EAALAEGALE AARRDRSRLQ MALLGASDGD VAASA UniProtKB: UNIPROTKB: A0A8F8EC09 |
-分子 #2: Crescentin-specific megabody MB13
| 分子 | 名称: Crescentin-specific megabody MB13 / タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Camelidae (哺乳類) Camelidae (哺乳類) |
| 分子量 | 理論値: 101.79018 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLQESGGG LVYKEETQSG LNNYARVVEK GQYDSLEIPA QVAASWESGR DDAAVFGFID KEQLDKYVAN GGKRSDWTVK FAENRSQDG TLLGYSLLQE SVDQASYMYS DNHYLAEMAT ILGKPEEAKR YRQLAQQLAD YINTCMFDPT TQFYYDVRIE D KPLANGCA ...文字列: EVQLQESGGG LVYKEETQSG LNNYARVVEK GQYDSLEIPA QVAASWESGR DDAAVFGFID KEQLDKYVAN GGKRSDWTVK FAENRSQDG TLLGYSLLQE SVDQASYMYS DNHYLAEMAT ILGKPEEAKR YRQLAQQLAD YINTCMFDPT TQFYYDVRIE D KPLANGCA GKPIVERGKG PEGWSPLFNG AATQANADAV VKVMLDPKEF NTFVPLGTAA LTNPAFGADI YWRGRVWVDQ FW FGLKGME RYGYRDDALK LADTFFRHAK GLTADGPIQE NYNPLTGAQQ GAPNFSWSAA HLYMLYNDFF RKQASGGGSG GGG SGGGGS GNADNYKNVI NRTGAPQYMK DYDYDDHQRF NPFFDLGAWH GHLLPDGPNT MGGFPGVALL TEEYINFMAS NFDR LTVWQ DGKKVDFTLE AYSIPGALVQ KLTAKDVQVE MTLRFATPRT SLLETKITSN KPLDLVWDGE LLEKLEAKEG KPLSD KTIA GEYPDYQRKI SATRDGLKVT FGKVRATWDL LTSGESEYQV HKSLPVQTEI NGNRFTSKAH INGSTTLYTT YSHLLT AQE VSKEQMQIRD ILARPAFYLT ASQQRWEEYL KKGLTNPDAT PEQTRVAVKA IETLNGNWRS PGGAVKFNTV TPSVTGR WF SGNQTWPWDT WKQAFAMAHF NPDIAKENIR AVFSWQIQPG DSVRPQDVGF VPDLIAWNLS PERGGDGGNW NERNTKPS L AAWSVMEVYN VTQDKTWVAE MYPKLVAYHD WWLRNRDHNG NGVPEYGATR DKAHNTESGE MLFTVKKDSL RLSCASSRS IDGINIMRWY RQAPGKQRGM VAVVTGWGST NYVDSVKGRF IISRDSAKDT VYLQMNNLKP EDTAVYSCNA IYRGSEYWGQ GTQVTVSSG ENLYFQGSHH HHHHHHHH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 / 詳細: PIPES 25mM pH 6.5, 0.05% CHAPS |
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 平均露光時間: 2.4 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8ajb: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)