+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human NKCC1 (TM domain) | |||||||||||||||||||||
 マップデータ マップデータ | Sharpened map of human NKCC1 transmembrane domains | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | SLC12 family / NKCC1 in complex with Na+ / K+ and 2Cl- / LeuT-fold / TRANSPORT PROTEIN | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of cell volume / positive regulation of aspartate secretion / transepithelial ammonium transport / regulation of matrix metallopeptidase secretion / cell body membrane / inorganic anion import across plasma membrane / inorganic cation import across plasma membrane / metal ion transmembrane transporter activity / chloride:monoatomic cation symporter activity / sodium:potassium:chloride symporter activity ...positive regulation of cell volume / positive regulation of aspartate secretion / transepithelial ammonium transport / regulation of matrix metallopeptidase secretion / cell body membrane / inorganic anion import across plasma membrane / inorganic cation import across plasma membrane / metal ion transmembrane transporter activity / chloride:monoatomic cation symporter activity / sodium:potassium:chloride symporter activity / transepithelial chloride transport / potassium ion transmembrane transporter activity / Cation-coupled Chloride cotransporters / intracellular chloride ion homeostasis / sodium ion homeostasis / negative regulation of vascular wound healing / ammonium transmembrane transport / chloride ion homeostasis / ammonium channel activity / cellular response to potassium ion / cell projection membrane / intracellular potassium ion homeostasis / cellular response to chemokine / T cell chemotaxis / sodium ion import across plasma membrane / potassium ion homeostasis / intracellular sodium ion homeostasis / hyperosmotic response / cell volume homeostasis / gamma-aminobutyric acid signaling pathway / regulation of spontaneous synaptic transmission / maintenance of blood-brain barrier / potassium ion import across plasma membrane / transport across blood-brain barrier / lateral plasma membrane / sodium ion transmembrane transport / monoatomic ion transport / chloride transmembrane transport / basal plasma membrane / cell projection / cell periphery / Hsp90 protein binding / cytoplasmic vesicle membrane / extracellular vesicle / protein-folding chaperone binding / cell body / basolateral plasma membrane / neuron projection / apical plasma membrane / neuronal cell body / protein kinase binding / extracellular exosome / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
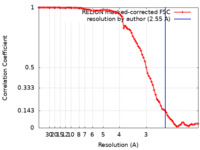
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.55 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Nissen P / Fenton R / Neumann C / Lindtoft Rosenbaek L / Kock Flygaard R / Habeck M / Lykkegaard Karlsen J / Wang Y / Lindorff-Larsen K / Gad H ...Nissen P / Fenton R / Neumann C / Lindtoft Rosenbaek L / Kock Flygaard R / Habeck M / Lykkegaard Karlsen J / Wang Y / Lindorff-Larsen K / Gad H / Hartmann R / Lyons J | |||||||||||||||||||||
| 資金援助 |  デンマーク, デンマーク,  フランス, 6件 フランス, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2022 ジャーナル: EMBO J / 年: 2022タイトル: Cryo-EM structure of the human NKCC1 transporter reveals mechanisms of ion coupling and specificity. 著者: Caroline Neumann / Lena Lindtoft Rosenbaek / Rasmus Kock Flygaard / Michael Habeck / Jesper Lykkegaard Karlsen / Yong Wang / Kresten Lindorff-Larsen / Hans Henrik Gad / Rune Hartmann / Joseph ...著者: Caroline Neumann / Lena Lindtoft Rosenbaek / Rasmus Kock Flygaard / Michael Habeck / Jesper Lykkegaard Karlsen / Yong Wang / Kresten Lindorff-Larsen / Hans Henrik Gad / Rune Hartmann / Joseph Anthony Lyons / Robert A Fenton / Poul Nissen /   要旨: The sodium-potassium-chloride transporter NKCC1 of the SLC12 family performs Na -dependent Cl - and K -ion uptake across plasma membranes. NKCC1 is important for regulating cell volume, hearing, ...The sodium-potassium-chloride transporter NKCC1 of the SLC12 family performs Na -dependent Cl - and K -ion uptake across plasma membranes. NKCC1 is important for regulating cell volume, hearing, blood pressure, and regulation of hyperpolarizing GABAergic and glycinergic signaling in the central nervous system. Here, we present a 2.6 Å resolution cryo-electron microscopy structure of human NKCC1 in the substrate-loaded (Na , K , and 2 Cl ) and occluded, inward-facing state that has also been observed for the SLC6-type transporters MhsT and LeuT. Cl binding at the Cl1 site together with the nearby K ion provides a crucial bridge between the LeuT-fold scaffold and bundle domains. Cl -ion binding at the Cl2 site seems to undertake a structural role similar to conserved glutamate of SLC6 transporters and may allow for Cl -sensitive regulation of transport. Supported by functional studies in mammalian cells and computational simulations, we describe a putative Na release pathway along transmembrane helix 5 coupled to the Cl2 site. The results provide insight into the structure-function relationship of NKCC1 with broader implications for other SLC12 family members. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14709.map.gz emd_14709.map.gz | 49.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14709-v30.xml emd-14709-v30.xml emd-14709.xml emd-14709.xml | 27.1 KB 27.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14709_fsc.xml emd_14709_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14709.png emd_14709.png | 102.2 KB | ||
| マスクデータ |  emd_14709_msk_1.map emd_14709_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14709.cif.gz emd-14709.cif.gz | 7.6 KB | ||
| その他 |  emd_14709_additional_1.map.gz emd_14709_additional_1.map.gz emd_14709_half_map_1.map.gz emd_14709_half_map_1.map.gz emd_14709_half_map_2.map.gz emd_14709_half_map_2.map.gz | 39.6 MB 39.8 MB 39.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14709 http://ftp.pdbj.org/pub/emdb/structures/EMD-14709 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14709 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14709 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14709_validation.pdf.gz emd_14709_validation.pdf.gz | 975.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14709_full_validation.pdf.gz emd_14709_full_validation.pdf.gz | 975.5 KB | 表示 | |
| XML形式データ |  emd_14709_validation.xml.gz emd_14709_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  emd_14709_validation.cif.gz emd_14709_validation.cif.gz | 20.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14709 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14709 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14709 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14709 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zgoMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14709.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14709.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of human NKCC1 transmembrane domains | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.014 Å | ||||||||||||||||||||||||||||||||||||
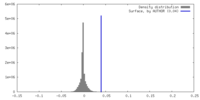
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14709_msk_1.map emd_14709_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
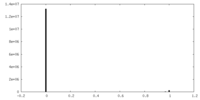
| 投影像・断面図 |
| ||||||||||||
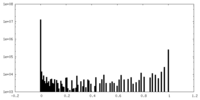
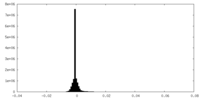
| 密度ヒストグラム |
-追加マップ: Full map, refined with a mask around transmembrane...
| ファイル | emd_14709_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full map, refined with a mask around transmembrane domains, unsharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
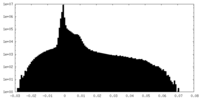
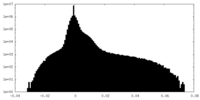
| 密度ヒストグラム |
-ハーフマップ: Unfiltered, unsharpened halfmap1
| ファイル | emd_14709_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered, unsharpened halfmap1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
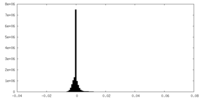
| 密度ヒストグラム |
-ハーフマップ: Unfiltered, unsharpened halfmap2
| ファイル | emd_14709_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered, unsharpened halfmap2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
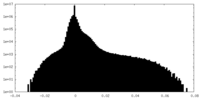
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Dimeric NKCC1 in complex with Na+, K+ and 2Cl-
+超分子 #1: Dimeric NKCC1 in complex with Na+, K+ and 2Cl-
+超分子 #2: NKCC1 in complex with Na+, K+ and 2Cl-
+超分子 #3: NKCC1 in complex with Na+, K+ and 2Cl-
+分子 #1: Solute carrier family 12 member 2
+分子 #2: SODIUM ION
+分子 #3: POTASSIUM ION
+分子 #4: CHLORIDE ION
+分子 #5: (2S)-3-(hexadecanoyloxy)-2-[(9Z)-octadec-9-enoyloxy]propyl 2-(tri...
+分子 #6: CHOLESTEROL HEMISUCCINATE
+分子 #7: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 詳細: 0.01% glycol-diosgenin (GDN, Anatrace), 200mM NaCl, 200mM KCl, 20mM Tris-HCl pH 8.0 and 0.07mM bumetanide |
|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 詳細: GloQube (Quorum) 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 詳細: 3 uL sample was applied to the gold foil side of the grid and blotted iusing a blot force of 0 and blot time of 3-4 seconds before plunge- freezing into liquid ethane cooled by liquid nitrogen.. |
| 詳細 | concentration (mg/mL): 0.9-1.5 |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #0 - 実像数: 10063 / #0 - 平均電子線量: 40.12 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #1 - 実像数: 9122 / #1 - 平均電子線量: 30.78 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 165000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | Image recording ID: 3 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 6190 / 平均電子線量: 60.438 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)