+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
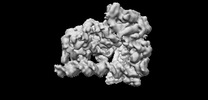
| タイトル | Mot1:TBP:DNA - post hydrolysis state | |||||||||
 マップデータ マップデータ | main density | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATP-dependent chromatin remodeler activity / RNA polymerase II general transcription initiation factor activity / TBP-class protein binding / helicase activity / DNA-templated transcription initiation / ATP hydrolysis activity / DNA binding / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetomium thermophilum (菌類) / DNA molecule (その他) Chaetomium thermophilum (菌類) / DNA molecule (その他) | |||||||||
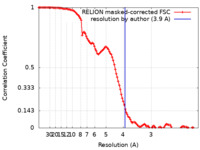
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Woike S / Eustermann S / Jung J / Wenzl SJ / Hagemann G / Bartho JD / Lammens K / Butryn A / Herzog F / Hopfner K-P | |||||||||
| 資金援助 |  ドイツ, European Union, 2件 ドイツ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structural basis for TBP displacement from TATA box DNA by the Swi2/Snf2 ATPase Mot1. 著者: Stephan Woike / Sebastian Eustermann / James Jung / Simon Josef Wenzl / Götz Hagemann / Joseph Bartho / Katja Lammens / Agata Butryn / Franz Herzog / Karl-Peter Hopfner /     要旨: The Swi2/Snf2 family transcription regulator Modifier of Transcription 1 (Mot1) uses adenosine triphosphate (ATP) to dissociate and reallocate the TATA box-binding protein (TBP) from and between ...The Swi2/Snf2 family transcription regulator Modifier of Transcription 1 (Mot1) uses adenosine triphosphate (ATP) to dissociate and reallocate the TATA box-binding protein (TBP) from and between promoters. To reveal how Mot1 removes TBP from TATA box DNA, we determined cryogenic electron microscopy structures that capture different states of the remodeling reaction. The resulting molecular video reveals how Mot1 dissociates TBP in a process that, intriguingly, does not require DNA groove tracking. Instead, the motor grips DNA in the presence of ATP and swings back after ATP hydrolysis, moving TBP to a thermodynamically less stable position on DNA. Dislodged TBP is trapped by a chaperone element that blocks TBP's DNA binding site. Our results show how Swi2/Snf2 proteins can remodel protein-DNA complexes through DNA bending without processive DNA tracking and reveal mechanistic similarities to RNA gripping DEAD box helicases and RIG-I-like immune sensors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14562.map.gz emd_14562.map.gz | 117.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14562-v30.xml emd-14562-v30.xml emd-14562.xml emd-14562.xml | 21.6 KB 21.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14562_fsc.xml emd_14562_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14562.png emd_14562.png | 52.4 KB | ||
| マスクデータ |  emd_14562_msk_1.map emd_14562_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14562_half_map_1.map.gz emd_14562_half_map_1.map.gz emd_14562_half_map_2.map.gz emd_14562_half_map_2.map.gz | 98.4 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14562 http://ftp.pdbj.org/pub/emdb/structures/EMD-14562 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14562 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14562 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14562_validation.pdf.gz emd_14562_validation.pdf.gz | 945.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14562_full_validation.pdf.gz emd_14562_full_validation.pdf.gz | 945.4 KB | 表示 | |
| XML形式データ |  emd_14562_validation.xml.gz emd_14562_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_14562_validation.cif.gz emd_14562_validation.cif.gz | 24.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14562 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14562 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14562 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14562 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14562.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14562.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main density | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14562_msk_1.map emd_14562_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
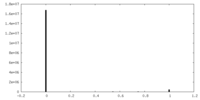
| 投影像・断面図 |
| ||||||||||||
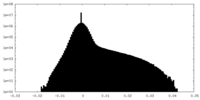
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_14562_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
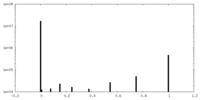
| 投影像・断面図 |
| ||||||||||||
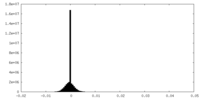
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_14562_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mot1:TBP:DNA post hydrolysis complex
| 全体 | 名称: Mot1:TBP:DNA post hydrolysis complex |
|---|---|
| 要素 |
|
-超分子 #1: Mot1:TBP:DNA post hydrolysis complex
| 超分子 | 名称: Mot1:TBP:DNA post hydrolysis complex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
-分子 #1: DNA (36-MER)
| 分子 | 名称: DNA (36-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: DNA molecule (その他) |
| 分子量 | 理論値: 11.155128 KDa |
| 配列 | 文字列: (DC)(DG)(DG)(DC)(DC)(DG)(DG)(DG)(DC)(DG) (DC)(DC)(DC)(DG)(DG)(DC)(DA)(DT)(DG)(DG) (DC)(DG)(DG)(DC)(DC)(DT)(DA)(DT)(DA) (DA)(DA)(DA)(DG)(DG)(DG)(DC) |
-分子 #2: DNA (36-MER)
| 分子 | 名称: DNA (36-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: DNA molecule (その他) |
| 分子量 | 理論値: 11.008016 KDa |
| 配列 | 文字列: (DG)(DC)(DC)(DC)(DT)(DT)(DT)(DT)(DA)(DT) (DA)(DG)(DG)(DC)(DC)(DG)(DC)(DC)(DA)(DT) (DG)(DC)(DC)(DG)(DG)(DG)(DC)(DG)(DC) (DC)(DC)(DG)(DG)(DC)(DC)(DG) |
-分子 #3: Putative tata-box binding protein
| 分子 | 名称: Putative tata-box binding protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 29.676973 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGENLYFQG HMEAIQTHPA NAAQAKAFTA PGSLSFPGGA SEIANSTAPT NGASNGGQQQ GVQATSGAGV TPATPAATP GAGPAGPSGI TPTLQNIVAT VNLDCRLDLK TIALHARNAE YNPKRFAAVI MRIREPKTTA LIFASGKMVV T GAKSEDDS ...文字列: MGSSHHHHHH SSGENLYFQG HMEAIQTHPA NAAQAKAFTA PGSLSFPGGA SEIANSTAPT NGASNGGQQQ GVQATSGAGV TPATPAATP GAGPAGPSGI TPTLQNIVAT VNLDCRLDLK TIALHARNAE YNPKRFAAVI MRIREPKTTA LIFASGKMVV T GAKSEDDS KLASRKYARI IQKLGFNAKF TDFKIQNIVG SCDIKFPIRL EGLASKHHNF SSYEPELFPG LIYRMIKPKI VL LIFVSGK IVLTGAKVRE EIYQAFEMIY PVLQDFRKV |
-分子 #4: Helicase-like protein
| 分子 | 名称: Helicase-like protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 Chaetomium thermophilum (菌類) / 株: DSM 1495 / CBS 144.50 / IMI 039719 |
| 分子量 | 理論値: 211.832688 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MATRLDRLVT ILETGSTRLI RDTAVNQLAD WQKQHPEELF NLLSRVVPYL RHKDWETRTT AAKAIGKIIE NAPLYDPNAG QDEAAPEPT NGSFEVKKEE EKDVLEQDNF FRLESLDVAT IVKYGRPLLR GGPVDYNLAA LDPQKRLAHL KKTLNGRLGL L GRVFEDEE ...文字列: MATRLDRLVT ILETGSTRLI RDTAVNQLAD WQKQHPEELF NLLSRVVPYL RHKDWETRTT AAKAIGKIIE NAPLYDPNAG QDEAAPEPT NGSFEVKKEE EKDVLEQDNF FRLESLDVAT IVKYGRPLLR GGPVDYNLAA LDPQKRLAHL KKTLNGRLGL L GRVFEDEE MPVEQIASPI TPNDAAGANG VGRQDGASND NQSQAIDESK MSARQLNVLK RKRKREAQKA AQGKSGFGDL SL RRSTTAG SDAFGEDTPM PDADSKKNKL AEYFSLDRPE NTEEDTKIVS EFKGPVLPIK SEIEADDSLE GAEWPFERLC EFL KVDLFD PQWETRHGAA MGLREVIRVH GAGAGRRRGK TRKENNDLNR QWLDDLAYRL LCVLMLDKFT DYSSDTSVAP IRET VGQTL GAVLRHISVE SVHAIYRLLY CMVTQEDLPS EQNMWAVCHG GMVGLRYVVA VRKDLLLQDG DMIDGVVRCV MQGLG DIDD DVRSVSAATL IPMAKEFVMM RRSALDSLIN IVWESLSNLG DDLSASTGKI MDLLATLCSF PEVLEAMKVS ASQDEE RSF TLLVPRLYPF LRHTITSVRL AVLKALMTFA NLGGETSQGW LNGRILRLIF QNIIVERDQD TLNMSLELWT TLVRRLA AR DPAILADEFE AHAEPMMQLA LHPIGVPRHP IPMNPALFQK PSGGTYSLPG ASQTNSRRSS PPEGERATKR RRKSTKAE D VAPSTHTHDV DGHMIQGEVD LVGVDVLIRS RISAAKAMGL IMSFIPTPRL ASYDTAVLQA LSSPYASTQL AAAMVIDEY AKNCSTPEVA SRFIEPLQKI IDLERPSHYR DLVTYVQRVR SASQQLINLF RDHGKVSQGK LPTLAVVVQG EPEAGPGAFS IANAEKVVN EDFERLKRLM APGQRLIALP QLNEAREQTV EVIEEAKAAK EARDARIKAA AACALVAMKV LPKKPSPLIK A IMDSIKTE ENQELQSRSA ATIARLVQLF TESGRRGPAE KVVANLVKFS CVEVAETPEF PIHAHKTNVI LSMQKEEDRV DH PDAVKYA REAKAARITR RGAKEALEIL SKNFGAELLE RVPTLRTFME EPLVRAFSGD LPPEARDPEN AFGQEIVDAM SVI RTMTPT LHPALHPFVM QQVPLVIKAL RSDLSVFRYM AAKCMATICS VITVDGMTAL VEKVLPSINN PLDLSFRQGA IEVI YHLIA VMGDAILPYV IFLIVPVLGR MSDSDNQIRL IATTSFATLV KLVPLEAGIP DPPGLSEELL KGRDRERTFI AQLLD PKKI EPFKIPVAIK AELRSYQQEG VNWLAFLNKY HLHGILCDDM GLGKTLQTIC IVASDHHQRA EEFARTGAPE VRKLPS LII CPPTLSGHWQ QEIKTYAPFL TVTAYVGSPA ERRAMKDSLD KTDIVITSYD VCRNDIDVIE KYNWNYCVLD EGHLIKN PK AKITLAVKRL TSNHRLILTG TPIQNNVLEL WSLFDFLMPG FLGAEKVFLD RFAKPIANSR YSKASSKEQE AGALAIEA L HKQVLPFLLR RLKEEVLNDL PPKILQNYYC DLSDLQRKLF EDFTKREGKK ITETAGRDDK EAKQHIFQAL QYMRKLCNS PALVMKPGHK AYEDTQKYLA KHGTTLEDPI HAPKLGALRD LLVDCGIGVE GQESSDPLYT PIKPHRALIF CQMKEMLDMV QNTVLKQML PSVSYLRLDG SVEANKRQDI VNKFNSDPSY DVLLLTTSVG GLGLNLTGAD TVIFVEHDWN PQKDLQAMDR A HRIGQKKV VNVYRIITRG TLEEKILSLQ RFKIDVASTV VNQQNAGLAT MDTDQILDLF NLGESGPSLI TDNKESIEGR EE DMVDIET GDVRRPGKKA AWLEGLGELW DNAQYEESFD LDGFLKTMQA AAWSHPQFEK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Incubation with 1 mM ATP-gamma-S before size exclusion chromatography. 0.05% beta-octyl glucoside added before blotting. | ||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 7 sec. / 前処理 - 雰囲気: AIR | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 283 K / 装置: LEICA EM GP 詳細: Incubation on the grid for 20 seconds before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 48.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X