+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of MuvB complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Quiescence / transcription / cell cycle | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Myb complex / CAF-1 complex / NURF complex / NuRD complex / regulation of cell fate specification / negative regulation of stem cell population maintenance / DNA replication-dependent chromatin assembly / ESC/E(Z) complex / Transcription of E2F targets under negative control by p107 (RBL1) and p130 (RBL2) in complex with HDAC1 / regulation of stem cell differentiation ...Myb complex / CAF-1 complex / NURF complex / NuRD complex / regulation of cell fate specification / negative regulation of stem cell population maintenance / DNA replication-dependent chromatin assembly / ESC/E(Z) complex / Transcription of E2F targets under negative control by p107 (RBL1) and p130 (RBL2) in complex with HDAC1 / regulation of stem cell differentiation / Transcription of E2F targets under negative control by DREAM complex / Polo-like kinase mediated events / DNA biosynthetic process / G1/S-Specific Transcription / positive regulation of stem cell population maintenance / ATPase complex / Sin3-type complex / Transcriptional Regulation by E2F6 / RNA Polymerase I Transcription Initiation / histone deacetylase complex / G0 and Early G1 / Cyclin E associated events during G1/S transition / Transcriptional regulation of brown and beige adipocyte differentiation by EBF2 / Cyclin A:Cdk2-associated events at S phase entry / Deposition of new CENPA-containing nucleosomes at the centromere / Regulation of TP53 Activity through Acetylation / transcription repressor complex / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / negative regulation of cell migration / PRC2 methylates histones and DNA / Regulation of PTEN gene transcription / Regulation of endogenous retroelements by KRAB-ZFP proteins / Defective pyroptosis / Regulation of endogenous retroelements by Piwi-interacting RNAs (piRNAs) / HDACs deacetylate histones / negative regulation of transforming growth factor beta receptor signaling pathway / brain development / PKMTs methylate histone lysines / histone deacetylase binding / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events / nucleosome assembly / histone binding / Oxidative Stress Induced Senescence / DNA replication / Potential therapeutics for SARS / chromosome, telomeric region / regulation of cell cycle / chromatin remodeling / RNA polymerase II cis-regulatory region sequence-specific DNA binding / negative regulation of cell population proliferation / negative regulation of DNA-templated transcription / DNA-templated transcription / regulation of DNA-templated transcription / chromatin / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / protein-containing complex / DNA binding / nucleoplasm / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Koliopoulos MG / Alfieri C | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of a nucleosome-bound MuvB transcription factor complex reveals DNA remodelling. 著者: Marios G Koliopoulos / Reyhan Muhammad / Theodoros I Roumeliotis / Fabienne Beuron / Jyoti S Choudhary / Claudio Alfieri /  要旨: Genes encoding the core cell cycle machinery are transcriptionally regulated by the MuvB family of protein complexes in a cell cycle-specific manner. Complexes of MuvB with the transcription factors ...Genes encoding the core cell cycle machinery are transcriptionally regulated by the MuvB family of protein complexes in a cell cycle-specific manner. Complexes of MuvB with the transcription factors B-MYB and FOXM1 activate mitotic genes during cell proliferation. The mechanisms of transcriptional regulation by these complexes are still poorly characterised. Here, we combine biochemical analysis and in vitro reconstitution, with structural analysis by cryo-electron microscopy and cross-linking mass spectrometry, to functionally examine these complexes. We find that the MuvB:B-MYB complex binds and remodels nucleosomes, thereby exposing nucleosomal DNA. This remodelling activity is supported by B-MYB which directly binds the remodelled DNA. Given the remodelling activity on the nucleosome, we propose that the MuvB:B-MYB complex functions as a pioneer transcription factor complex. In this work, we rationalise prior biochemical and cellular studies and provide a molecular framework of interactions on a protein complex that is key for cell cycle regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14239.map.gz emd_14239.map.gz | 2.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14239-v30.xml emd-14239-v30.xml emd-14239.xml emd-14239.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14239.png emd_14239.png | 99 KB | ||
| Filedesc metadata |  emd-14239.cif.gz emd-14239.cif.gz | 5.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14239 http://ftp.pdbj.org/pub/emdb/structures/EMD-14239 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14239 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14239 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14239_validation.pdf.gz emd_14239_validation.pdf.gz | 324.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14239_full_validation.pdf.gz emd_14239_full_validation.pdf.gz | 324.5 KB | 表示 | |
| XML形式データ |  emd_14239_validation.xml.gz emd_14239_validation.xml.gz | 5.8 KB | 表示 | |
| CIF形式データ |  emd_14239_validation.cif.gz emd_14239_validation.cif.gz | 6.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14239 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14239 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14239 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14239 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7r1dMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14239.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14239.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.134 Å | ||||||||||||||||||||||||||||||||||||
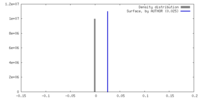
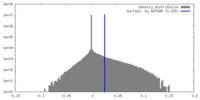
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : MuvB complex
| 全体 | 名称: MuvB complex |
|---|---|
| 要素 |
|
-超分子 #1: MuvB complex
| 超分子 | 名称: MuvB complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 180 KDa |
-分子 #1: Protein lin-9 homolog
| 分子 | 名称: Protein lin-9 homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 62.03566 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAELDQLPDE SSSAKALVSL KEGSLSNTWN EKYSSLQKTP VWKGRNTSSA VEMPFRNSKR SRLFSDEDDR QINTRSPKRN QRVAMVPQK FTATMSTPDK KASQKIGFRL RNLLKLPKAH KWCIYEWFYS NIDKPLFEGD NDFCVCLKES FPNLKTRKLT R VEWGKIRR ...文字列: MAELDQLPDE SSSAKALVSL KEGSLSNTWN EKYSSLQKTP VWKGRNTSSA VEMPFRNSKR SRLFSDEDDR QINTRSPKRN QRVAMVPQK FTATMSTPDK KASQKIGFRL RNLLKLPKAH KWCIYEWFYS NIDKPLFEGD NDFCVCLKES FPNLKTRKLT R VEWGKIRR LMGKPRRCSS AFFEEERSAL KQKRQKIRLL QQRKVADVSQ FKDLPDEIPL PLVIGTKVTA RLRGVHDGLF TG QIDAVDT LNATYRVTFD RTGLGTHTIP DYEVLSNEPH ETMPIAAFGQ KQRPSRFFMT PPRLHYTPPL QSPIIDNDPL LGQ SPWRSK ISGSDTETLG GFPVEFLIQV TRLSKILMIK KEHIKKLREM NTEAEKLKSY SMPISIEFQR RYATIVLELE QLNK DLNKV LHKVQQYCYE LAPDQGLQPA DQPTDMRRRC EEEAQEIVRH ANSSTGQPCV ENENLTDLIS RLTAILLQIK CLAEG GDLN SFEFKSLTDS LNDIKSTIDA SNISCFQNNV EIHVAHIQSG LSQMGNLHAF AANNTNRD UniProtKB: Protein lin-9 homolog |
-分子 #2: Histone-binding protein RBBP4
| 分子 | 名称: Histone-binding protein RBBP4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 47.709527 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MADKEAAFDD AVEERVINEE YKIWKKNTPF LYDLVMTHAL EWPSLTAQWL PDVTRPEGKD FSIHRLVLGT HTSDEQNHLV IASVQLPND DAQFDASHYD SEKGEFGGFG SVSGKIEIEI KINHEGEVNR ARYMPQNPCI IATKTPSSDV LVFDYTKHPS K PDPSGECN ...文字列: MADKEAAFDD AVEERVINEE YKIWKKNTPF LYDLVMTHAL EWPSLTAQWL PDVTRPEGKD FSIHRLVLGT HTSDEQNHLV IASVQLPND DAQFDASHYD SEKGEFGGFG SVSGKIEIEI KINHEGEVNR ARYMPQNPCI IATKTPSSDV LVFDYTKHPS K PDPSGECN PDLRLRGHQK EGYGLSWNPN LSGHLLSASD DHTICLWDIS AVPKEGKVVD AKTIFTGHTA VVEDVSWHLL HE SLFGSVA DDQKLMIWDT RSNNTSKPSH SVDAHTAEVN CLSFNPYSEF ILATGSADKT VALWDLRNLK LKLHSFESHK DEI FQVQWS PHNETILASS GTDRRLNVWD LSKIGEEQSP EDAEDGPPEL LFIHGGHTAK ISDFSWNPNE PWVICSVSED NIMQ VWQMA ENIYNDEDPE GSVDPEGQGS UniProtKB: Histone-binding protein RBBP4 |
-分子 #3: Protein lin-37 homolog
| 分子 | 名称: Protein lin-37 homolog / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.37524 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MFPVKVKVEK SELEMAKARN QLDAVLQCLL EKSHMDRERL DEEAGKTPSD THNKDCSIAA TGKRPSARFP HQRRKKRREM DDGLAEGGP QRSNTYVIKL FDRSVDLAQF SENTPLYPIC RAWMRNSPSV RERECSPSSP LPPLPEDEEG SEVTNSKSRD V YKLPPPTP ...文字列: MFPVKVKVEK SELEMAKARN QLDAVLQCLL EKSHMDRERL DEEAGKTPSD THNKDCSIAA TGKRPSARFP HQRRKKRREM DDGLAEGGP QRSNTYVIKL FDRSVDLAQF SENTPLYPIC RAWMRNSPSV RERECSPSSP LPPLPEDEEG SEVTNSKSRD V YKLPPPTP PGPPGDACRS RIPSPLQPEM QGTPDDEPSE PEPSPSTLIY RNMQRWKRIR QRWKEASHRN QLRYSESMKI LR EMYERQG SALEVLFQ UniProtKB: Protein lin-37 homolog |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | 材質: COPPER |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 26641 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)