+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-14120 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human RZZ kinetochore corona complex. | |||||||||
 マップデータ マップデータ | Composite map from focussed refinement maps | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein localization to kinetochore involved in kinetochore assembly / RZZ complex / Dsl1/NZR complex / kinetochore microtubule / centromeric DNA binding / regulation of attachment of spindle microtubules to kinetochore / regulation of exit from mitosis / protein localization to kinetochore / COPI-dependent Golgi-to-ER retrograde traffic / mitotic metaphase chromosome alignment ...protein localization to kinetochore involved in kinetochore assembly / RZZ complex / Dsl1/NZR complex / kinetochore microtubule / centromeric DNA binding / regulation of attachment of spindle microtubules to kinetochore / regulation of exit from mitosis / protein localization to kinetochore / COPI-dependent Golgi-to-ER retrograde traffic / mitotic metaphase chromosome alignment / mitotic spindle assembly checkpoint signaling / Golgi organization / establishment of mitotic spindle orientation / mitotic sister chromatid segregation / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / endoplasmic reticulum to Golgi vesicle-mediated transport / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / Resolution of Sister Chromatid Cohesion / lipid droplet / bioluminescence / meiotic cell cycle / generation of precursor metabolites and energy / RHO GTPases Activate Formins / kinetochore / small GTPase binding / spindle pole / Separation of Sister Chromatids / protein transport / actin cytoskeleton / protein-containing complex assembly / cell division / endoplasmic reticulum membrane / endoplasmic reticulum / membrane / nucleus / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
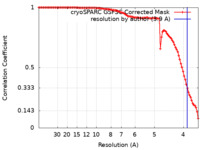
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Raisch T / Ciossani G / d'Amico E / Cmetowski V / Carmignani S / Maffini S / Merino F / Wohlgemuth S / Vetter IR / Raunser S / Musacchio A | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2022 ジャーナル: EMBO J / 年: 2022タイトル: Structure of the RZZ complex and molecular basis of Spindly-driven corona assembly at human kinetochores. 著者: Tobias Raisch / Giuseppe Ciossani / Ennio d'Amico / Verena Cmentowski / Sara Carmignani / Stefano Maffini / Felipe Merino / Sabine Wohlgemuth / Ingrid R Vetter / Stefan Raunser / Andrea Musacchio /  要旨: In metazoans, a ≈1 megadalton (MDa) multiprotein complex comprising the dynein-dynactin adaptor Spindly and the ROD-Zwilch-ZW10 (RZZ) complex is the building block of a fibrous biopolymer, the ...In metazoans, a ≈1 megadalton (MDa) multiprotein complex comprising the dynein-dynactin adaptor Spindly and the ROD-Zwilch-ZW10 (RZZ) complex is the building block of a fibrous biopolymer, the kinetochore fibrous corona. The corona assembles on mitotic kinetochores to promote microtubule capture and spindle assembly checkpoint (SAC) signaling. We report here a high-resolution cryo-EM structure that captures the essential features of the RZZ complex, including a farnesyl-binding site required for Spindly binding. Using a highly predictive in vitro assay, we demonstrate that the SAC kinase MPS1 is necessary and sufficient for corona assembly at supercritical concentrations of the RZZ-Spindly (RZZS) complex, and describe the molecular mechanism of phosphorylation-dependent filament nucleation. We identify several structural requirements for RZZS polymerization in rings and sheets. Finally, we identify determinants of kinetochore localization and corona assembly of Spindly. Our results describe a framework for the long-sought-for molecular basis of corona assembly on metazoan kinetochores. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14120.map.gz emd_14120.map.gz | 1.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14120-v30.xml emd-14120-v30.xml emd-14120.xml emd-14120.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14120_fsc.xml emd_14120_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14120.png emd_14120.png | 52.8 KB | ||
| マスクデータ |  emd_14120_msk_1.map emd_14120_msk_1.map emd_14120_msk_2.map emd_14120_msk_2.map | 103 MB 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14120_additional_1.map.gz emd_14120_additional_1.map.gz emd_14120_additional_2.map.gz emd_14120_additional_2.map.gz | 97 MB 2.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14120 http://ftp.pdbj.org/pub/emdb/structures/EMD-14120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14120 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14120 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14120_validation.pdf.gz emd_14120_validation.pdf.gz | 370.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14120_full_validation.pdf.gz emd_14120_full_validation.pdf.gz | 370 KB | 表示 | |
| XML形式データ |  emd_14120_validation.xml.gz emd_14120_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_14120_validation.cif.gz emd_14120_validation.cif.gz | 14.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14120 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14120 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14120 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14120 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14120.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14120.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map from focussed refinement maps | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_14120_msk_1.map emd_14120_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
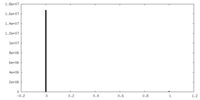
| 投影像・断面図 |
| ||||||||||||
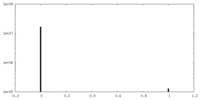
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_14120_msk_2.map emd_14120_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
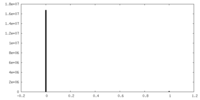
| 投影像・断面図 |
| ||||||||||||
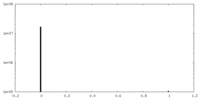
| 密度ヒストグラム |
-追加マップ: Map from global Non-uniform refinement
| ファイル | emd_14120_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from global Non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Locally filtered map
| ファイル | emd_14120_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RZZ complex
| 全体 | 名称: RZZ complex |
|---|---|
| 要素 |
|
-超分子 #1: RZZ complex
| 超分子 | 名称: RZZ complex / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-分子 #1: Protein zwilch homolog
| 分子 | 名称: Protein zwilch homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 67.293805 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MWERLNCAAE DFYSRLLQKF NEEKKGIRKD PFLYEADVQV QLISKGQPNP LKNILNENDI VFIVEKVPLE KEETSHIEEL QSEETAISD FSTGENVGPL ALPVGKARQL IGLYTMAHNP NMTHLKINLP VTALPPLWVR CDSSDPEGTC WLGAELITTN N SITGIVLY ...文字列: MWERLNCAAE DFYSRLLQKF NEEKKGIRKD PFLYEADVQV QLISKGQPNP LKNILNENDI VFIVEKVPLE KEETSHIEEL QSEETAISD FSTGENVGPL ALPVGKARQL IGLYTMAHNP NMTHLKINLP VTALPPLWVR CDSSDPEGTC WLGAELITTN N SITGIVLY VVSCKADKNY SVNLENLKNL HKKRHHLSTV TSKGFAQYEL FKSSALDDTI TASQTAIALD ISWSPVDEIL QI PPLSSTA TLNIKVESGE PRGPLNHLYR ELKFLLVLAD GLRTGVTEWL EPLEAKSAVE LVQEFLNDLN KLDGFGDSTK KDT EVETLK HDTAAVDRSV KRLFKVRSDL DFAEQLWCKM SSSVISYQDL VKCFTLIIQS LQRGDIQPWL HSGSNSLLSK LIHQ SYHGT MDTVSLSGTI PVQMLLEIGL DKLKKDYISF FIGQELASLN HLEYFIAPSV DIQEQVYRVQ KLHHILEILV SCMPF IKSQ HELLFSLTQI CIKYYKQNPL DEQHIFQLPV RPTAVKNLYQ SEKPQKWRVE IYSGQKKIKT VWQLSDSSPI DHLNFH KPD FSELTLNGSL EERIFFTNMV TCSQVHFK |
-分子 #2: Kinetochore-associated protein 1
| 分子 | 名称: Kinetochore-associated protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 279.805562 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAHHHHHHSS GVSKGEEDNM AIIKEFMRFK VHMEGSVNGH EFEIEGEGEG RPYEGTQTAK LKVTKGGPLP FAWDILSPQF MYGSKAYVK HPADIPDYLK LSFPEGFKWE RVMNFEDGGV VTVTQDSSLQ DGEFIYKVKL RGTNFPSDGP VMQKKTMGWE A SSERMYPE ...文字列: MAHHHHHHSS GVSKGEEDNM AIIKEFMRFK VHMEGSVNGH EFEIEGEGEG RPYEGTQTAK LKVTKGGPLP FAWDILSPQF MYGSKAYVK HPADIPDYLK LSFPEGFKWE RVMNFEDGGV VTVTQDSSLQ DGEFIYKVKL RGTNFPSDGP VMQKKTMGWE A SSERMYPE DGALKGEIKQ RLKLKDGGHY DAEVKTTYKA KKPVQLPGAY NVNIKLDITS HNEDYTIVEQ YERAEGRHST GG MDELYKL EVLFQGPGSW NDIELLTNDD TGSGYLSVGS RKEHGTALYQ VDLLVKISSE KASLNPKIQA CSLSDGFIIV ADQ SVILLD SICRSLQLHL VFDTEVDVVG LCQEGKFLLV GERSGNLHLI HVTSKQTLLT NAFVQKANDE NRRTYQNLVI EKDG SNEGT YYMLLLTYSG FFCITNLQLL KIQQAIENVD FSTAKKLQGQ IKSSFISTEN YHTLGCLSLV AGDLASEVPV IIGGT GNCA FSKWEPDSSK KGMTVKNLID AEIIKGAKKF QLIDNLLFVL DTDNVLSLWD IYTLTPVWNW PSLHVEEFLL TTEADS PSS VTWQGITNLK LIALTASANK KMKNLMVYSL PTMEILYSLE VSSVSSLVQT GISTDTIYLL EGVCKNDPKL SEDSVSV LV LRCLTEALPE NRLSRLLHKH RFAEAESFAI QFGLDVELVY KVKSNHILEK LALSSVDASE QTEWQQLVDD AKENLHKI Q DDEFVVNYCL KAQWITYETT QEMLNYAKTR LLKKEDKTAL IYSDGLKEVL RAHAKLTTFY GAFGPEKFSG SSWIEFLNN EDDLKDIFLQ LKEGNLVCAQ YLWLRHRANF ESRFDVKMLE SLLNSMSASV SLQKLCPWFK NDVIPFVRRT VPEGQIILAK WLEQAARNL ELTDKANWPE NGLQLAEIFF TAEKTDELGL ASSWHWISLK DYQNTEEVCQ LRTLVNNLRE LITLHRKYNC K LALSDFEK ENTTTIVFRM FDKVLAPELI PSILEKFIRV YMREHDLQEE ELLLLYIEDL LNRCSSKSTS LFETAWEAKA MA VIACLSD TDLIFDAVLK IMYAAVVPWS AAVEQLVKQH LEMDHPKVKL LQESYKLMEM KKLLRGYGIR EVNLLNKEIM RVV RYILKQ DVPSSLEDAL KVAQAFMLSD DEIYSLRIID LIDREQGEDC LLLLKSLPPA EAEKTAERVI IWARLALQEE PDHS KEGKA WRMSVAKTSV DILKILCDIQ KDNLQKKDEC EEMLKLFKEV ASLQENFEVF LSFEDYSNSS LVADLREQHI KAHEV AQAK HKPGSTPEPI AAEVRSPSME SKLHRQALAL QMSKQELEAE LTLRALKDGN IKTALKKCSD LFKYHCNADT GKLLFL TCQ KLCQMLADNV PVTVPVGLNL PSMIHDLASQ AATICSPDFL LDALELCKHT LMAVELSRQC QMDDCGILMK ASFGTHK DP YEEWSYSDFF SEDGIVLESQ MVLPVIYELI SSLVPLAESK RYPLESTSLP YCSLNEGDGL VLPVINSISA LLQNLQES S QWELALRFVV GSFGTCLQHS VSNFMNATLS EKLFGETTLV KSRHVVMELK EKAVIFIREN ATTLLHKVFN CRLVDLDLA LGYCTLLPQK DVFENLWKLI DKAWQNYDKI LAISLVGSEL ASLYQEIEMG LKFRELSTDA QWGIRLGKLG ISFQPVFRQH FLTKKDLIK ALVENIDMDT SLILEYCSTF QLDCDAVLQL FIETLLHNTN AGQGQGDASM DSAKRRHPKL LAKALEMVPL L TSTKDLVI SLSGILHKLD PYDYEMIEVV LKVIERADEK ITNININQAL SILKHLKSYR RISPPVDLEY QYMLEHVITL PS AAQTRLP FHLIFFGTAQ NFWKILSTEL SEESFPTLLL ISKLMKFSLD TLYVSTAKHV FEKKLKPKLL KLTQAKSSTL INK EITKIT QTIESCLLSI VNPEWAVAIA ISLAQDIPEG SFKISALKFC LYLAERWLQN IPSQDEKREK AEALLKKLHI QYRR SGTEA VLIAHKLNTE EYLRVIGKPA HLIVSLYEHP SINQRIQNSS GTDYPDIHAA AKEIAEVNEI NLEKVWDMLL EKWLC PSTK PGEKPSELFE LQEDEALRRV QYLLLSRPID YSSRMLFVFA TSTTTTLGMH QLTFAHRTRA LQCLFYLADK ETIESL FKK PIEEVKSYLR CITFLASFET LNIPITYELF CSSPKEGMIK GLWKNHSHES MAVRLVTELC LEYKIYDLQL WNGLLQK LL GFNMIPYLRK VLKAISSIHS LWQVPYFSKA WQRVIQIPLL SASCPLSPDQ LSDCSESLIA VLECPVSGDL DLIGVARQ Y IQLELPAFAL ACLMLMPHSE KRHQQIKNFL GSCDPQVILK QLEEHMNTGQ LAGFSHQIRS LILNNIINKK EFGILAKTK YFQMLKMHAM NTNNITELVN YLANDLSLDE ASVLITEYSK HCGKPVPPDT APCEILKMFL SGLS |
-分子 #3: Centromere/kinetochore protein zw10 homolog
| 分子 | 名称: Centromere/kinetochore protein zw10 homolog / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 88.940336 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MASFVTEVLA HSGRLEKEDL GTRISRLTRR VEEIKGEVCN MISKKYSEFL PSMQSAQGLI TQVDKLSEDI DLLKSRIESE VRRDLHVST GEFTDLKQQL ERDSVVLSLL KQLQEFSTAI EEYNCALTEK KYVTGAQRLE EAQKCLKLLK SRKCFDLKIL K SLSMELTI ...文字列: MASFVTEVLA HSGRLEKEDL GTRISRLTRR VEEIKGEVCN MISKKYSEFL PSMQSAQGLI TQVDKLSEDI DLLKSRIESE VRRDLHVST GEFTDLKQQL ERDSVVLSLL KQLQEFSTAI EEYNCALTEK KYVTGAQRLE EAQKCLKLLK SRKCFDLKIL K SLSMELTI QKQNILYHLG EEWQKLIVWK FPPSKDTSSL ESYLQTELHL YTEQSHKEEK TPMPPISSVL LAFSVLGELH SK LKSFGQM LLKYILRPLA SCPSLHAVIE SQPNIVIIRF ESIMTNLEYP SPSEVFTKIR LVLEVLQKQL LDLPLDTDLE NEK TSTVPL AEMLGDMIWE DLSECLIKNC LVYSIPTNSS KLQQYEEIIQ STEEFENALK EMRFLKGDTT DLLKYARNIN SHFA NKKCQ DVIVAARNLM TSEIHNTVKI IPDSKINVPE LPTPDEDNKL EVQKVSNTQY HEVMNLEPEN TLDQHSFSLP TCRIS ESVK KLMELAYQTL LEATTSSDQC AVQLFYSVRN IFHLFHDVVP TYHKENLQKL PQLAAIHHNN CMYIAHHLLT LGHQFR LRL APILCDGTAT FVDLVPGFRR LGTECFLAQM RAQKGELLER LSSARNFSNM DDEENYSAAS KAVRQVLHQL KRLGIVW QD VLPVNIYCKA MGTLLNTAIS EVIGKITALE DISTEDGDRL YSLCKTVMDE GPQVFAPLSE ESKNKKYQEE VPVYVPKW M PFKELMMMLQ ASLQEIGDRW ADGKGPLAAA FSSSEVKALI RALFQNTERR AAALAKIK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X