+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S.c. Condensin core in DNA- and ATP-bound state | |||||||||
 マップデータ マップデータ | deepEMhancer processed cryoEM map S.c. Condensin in DNA- and ATP-bound state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SMC-motor protein / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic DNA double-strand break formation / meiotic chromosome condensation / Condensation of Prometaphase Chromosomes / tRNA gene clustering / meiotic chromosome separation / condensin complex / DNA secondary structure binding / maintenance of rDNA / rDNA chromatin condensation / nucleophagy ...negative regulation of meiotic DNA double-strand break formation / meiotic chromosome condensation / Condensation of Prometaphase Chromosomes / tRNA gene clustering / meiotic chromosome separation / condensin complex / DNA secondary structure binding / maintenance of rDNA / rDNA chromatin condensation / nucleophagy / synaptonemal complex assembly / condensed chromosome, centromeric region / mitotic chromosome condensation / chromosome condensation / silent mating-type cassette heterochromatin formation / minor groove of adenine-thymine-rich DNA binding / mitotic sister chromatid segregation / condensed chromosome / histone binding / double-stranded DNA binding / cell division / chromatin binding / chromatin / nucleolus / ATP hydrolysis activity / mitochondrion / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
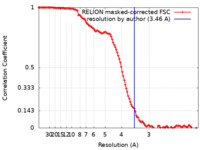
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.46 Å | |||||||||
 データ登録者 データ登録者 | Lecomte L / Hassler M / Haering C / Eustermann S | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: A hold-and-feed mechanism drives directional DNA loop extrusion by condensin. 著者: Indra A Shaltiel / Sumanjit Datta / Léa Lecomte / Markus Hassler / Marc Kschonsak / Sol Bravo / Catherine Stober / Jenny Ormanns / Sebastian Eustermann / Christian H Haering /  要旨: Structural maintenance of chromosomes (SMC) protein complexes structure genomes by extruding DNA loops, but the molecular mechanism that underlies their activity has remained unknown. We show that ...Structural maintenance of chromosomes (SMC) protein complexes structure genomes by extruding DNA loops, but the molecular mechanism that underlies their activity has remained unknown. We show that the active condensin complex entraps the bases of a DNA loop transiently in two separate chambers. Single-molecule imaging and cryo-electron microscopy suggest a putative power-stroke movement at the first chamber that feeds DNA into the SMC-kleisin ring upon adenosine triphosphate binding, whereas the second chamber holds on upstream of the same DNA double helix. Unlocking the strict separation of "motor" and "anchor" chambers turns condensin from a one-sided into a bidirectional DNA loop extruder. We conclude that the orientation of two topologically bound DNA segments during the SMC reaction cycle determines the directionality of DNA loop extrusion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13934.map.gz emd_13934.map.gz | 106.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13934-v30.xml emd-13934-v30.xml emd-13934.xml emd-13934.xml | 35.5 KB 35.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13934_fsc.xml emd_13934_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13934.png emd_13934.png | 72.1 KB | ||
| マスクデータ |  emd_13934_msk_1.map emd_13934_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13934.cif.gz emd-13934.cif.gz | 10.4 KB | ||
| その他 |  emd_13934_additional_1.map.gz emd_13934_additional_1.map.gz emd_13934_additional_2.map.gz emd_13934_additional_2.map.gz emd_13934_half_map_1.map.gz emd_13934_half_map_1.map.gz emd_13934_half_map_2.map.gz emd_13934_half_map_2.map.gz | 98.6 MB 76.4 MB 98.6 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13934 http://ftp.pdbj.org/pub/emdb/structures/EMD-13934 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13934 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13934 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13934_validation.pdf.gz emd_13934_validation.pdf.gz | 832.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13934_full_validation.pdf.gz emd_13934_full_validation.pdf.gz | 832 KB | 表示 | |
| XML形式データ |  emd_13934_validation.xml.gz emd_13934_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_13934_validation.cif.gz emd_13934_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13934 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13934 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13934 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13934 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qenMC  7qfwC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13934.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13934.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | deepEMhancer processed cryoEM map S.c. Condensin in DNA- and ATP-bound state | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13934_msk_1.map emd_13934_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
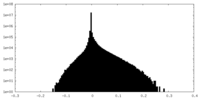
| 密度ヒストグラム |
-追加マップ: unsharpened cryoEM map S.c. Condensin in DNA- and ATP-bound state
| ファイル | emd_13934_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened cryoEM map S.c. Condensin in DNA- and ATP-bound state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
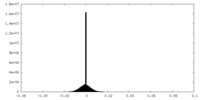
| 密度ヒストグラム |
-追加マップ: local resolution filtered and sharpened cryoEM map S.c....
| ファイル | emd_13934_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution filtered and sharpened cryoEM map S.c. Condensin in DNA- and ATP-bound state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
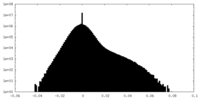
| 密度ヒストグラム |
-ハーフマップ: cryoEM half map 1 S.c. Condensin in DNA- and ATP-bound state
| ファイル | emd_13934_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM half map 1 S.c. Condensin in DNA- and ATP-bound state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
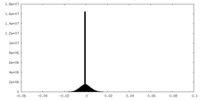
| 密度ヒストグラム |
-ハーフマップ: cryoEM half map 1 S.c. Condensin in DNA- and ATP-bound state
| ファイル | emd_13934_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM half map 1 S.c. Condensin in DNA- and ATP-bound state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ATP- and DNA- bound Sc Condensin core complex
| 全体 | 名称: ATP- and DNA- bound Sc Condensin core complex |
|---|---|
| 要素 |
|
-超分子 #1: ATP- and DNA- bound Sc Condensin core complex
| 超分子 | 名称: ATP- and DNA- bound Sc Condensin core complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 645 kDa/nm |
-分子 #1: DNA (35-MER)
| 分子 | 名称: DNA (35-MER) / タイプ: dna / ID: 1 詳細: 50-mer DNA ligand (sequence below) was modelled as poly-dA due to missing sequence register 5'-GTTGACAGTG TCGCAACCTG CACAGGCAAG CTGCTGAGTC TGGTGTAGAC-3' コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.615376 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA) |
-分子 #2: DNA (35-MER)
| 分子 | 名称: DNA (35-MER) / タイプ: dna / ID: 2 詳細: 50-mer DNA ligand (sequence below) was modelled as poly-dT due to missing sequence register. 5'-GTCTACACCAGACTCAGCAGCTTGCCTGTGCAGGTTGCGACACTGTCAAC-3' コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.164683 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT) |
-分子 #3: Structural maintenance of chromosomes protein 4
| 分子 | 名称: Structural maintenance of chromosomes protein 4 / タイプ: protein_or_peptide / ID: 3 詳細: ATPase deficient Walker B motif mutant central coiled-coil region results in faulty sequence alignment as it was not build in the structure To circumvent this technical problem, we had to ...詳細: ATPase deficient Walker B motif mutant central coiled-coil region results in faulty sequence alignment as it was not build in the structure To circumvent this technical problem, we had to delete the sequence corresponding to the coiled coil regions. This error needs to be corrected in communication with the pdb curator.,ATPase deficient Walker B motif mutant central coiled-coil region results in faulty sequence alignment as it was not build in the structure To circumvent this technical problem, we had to delete the sequence corresponding to the coiled coil regions. This error needs to be corrected in communication with the pdb curator.,ATPase deficient Walker B motif mutant central coiled-coil region results in faulty sequence alignment as it was not build in the structure To circumvent this technical problem, we had to delete the sequence corresponding to the coiled coil regions. This error needs to be corrected in communication with the pdb curator. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 167.716125 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDSPLSKRQ KRKAAQEPEL SLDQGDAEEE SQVENRVNLS ENTPEPDLPA LEASYSKSYT PRKLVLSSGE NRYAFSQPTN STTTSLHVP NLQPPKTSSR GRDHKSYSQS PPRSPGRSPT RRLEL(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK) ...文字列: MSDSPLSKRQ KRKAAQEPEL SLDQGDAEEE SQVENRVNLS ENTPEPDLPA LEASYSKSYT PRKLVLSSGE NRYAFSQPTN STTTSLHVP NLQPPKTSSR GRDHKSYSQS PPRSPGRSPT RRLEL(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)YDSHQSSS KQQSRLFINE LVLENFKSYA GKQVVGP FH TSFSAVVGPN GSGKSNVIDS MLFVFGFRAN KMRQDRLSDL IHKSEAFPSL QSCSVAVHFQ YVIDESSGTS RIDEEKPG L IITRKAFKNN SSKYYINEKE SSYTEVTKLL KNEGIDLDHK RFLILQGEVE NIAQMKPKAE KESDDGLLEY LEDIIGTAN YKPLIEERMG QIENLNEVCL EKENRFEIVD REKNSLESGK ETALEFLEKE KQLTLLRSKL FQFKLLQSNS KLASTLEKIS SSNKDLEDE RMKFQESLKK VDEIKAQRKE IKDRISSCSS KEKTLVLERR ELEGTRVSLE ERTKNLVSKM EKAEKTLKST K HSISEAEN MLEELRGQQT EHETEIKDLT QLLEKERSIL DDIKLSLKDK TKDISAEIIR HEKELEPWDL QLQEKESQIQ LA ESELSLL EETQAKLKKN VETLEEKILA KKTHKQELQD LILDLKKKLN SLKDERSQGE KNFTSAHLKL KEMQKVLNAH RQR AMEARS SLSKAQNKSK VLTALSRLQK SGRINGFHGR LGDLGAIDDS FDIAISTACP RLDDVVVDTV ECAQHCIDYL RKNK LGYAR FILLDRLRQF NLQPISTPEN VPRLFDLVKP KNPKFSNAFY SVLRDTLVAQ NLKQANNVAY GKKRFRVVTV DGKLI DISG TMSGGGNHVA KGLMKLGTNQ SDKVDDYTPE EVDKIERELS ERENNFRVAS DTVHEMEEEL KKLRDHEPDL ESQISK AEM EADSLASELT LAEQQVKEAE MAYVKAVSDK AQLNVVMKNL ERLRGEYNDL QSETKTKKEK IKGLQDEIMK IGGIKLQ MQ NSKVESVCQK LDILVAKLKK VKSASKKSGG DVVKFQKLLQ NSERDVELSS NELKVIEEQL KHTKLALAEN DTNMTETL N LKVELKEQSE QLKEQMEDME ESINEFKSIE IEMKNKLEKL NSLLTYIKSE ITQQEKGLNE LSIRDVTHTL GMLDDNKMD SVKEDVKNNQ ELDQEYRSCE TQDESEIKDD ETSCDNYHPM NVDETSDEVS RGIPRLSEDE LRELDVELIE SKINELSYYV EETNVDIGV LEEYARRLAE FKRRKLDLNN AVQKRDEVKE QLGILKKKRF DEFMAGFNII SMTLKEMYQM ITMGGNAELE L VDSLDPFS EGVTFSVMPP KKSWRNITNL SGGEKTLSSL ALVFALHKYK PTPLYVMDQI DAALDFRNVS IVANYIKERT KN AQFIVIS LRNNMFELAQ QLVGVYKRDN RTKSTTIKNI DILNRTRIPG LINGATGWSH PQFEKAGGGS GGGSGGGSWS HPQ FEKGGG SGGGSGGGSW SHPQFEK UniProtKB: Structural maintenance of chromosomes protein 4, Structural maintenance of chromosomes protein 4 |
-分子 #4: Structural maintenance of chromosomes protein 2
| 分子 | 名称: Structural maintenance of chromosomes protein 2 / タイプ: protein_or_peptide / ID: 4 / 詳細: ATPase deficient Walker B motif mutant / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 134.124891 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKVEELIIDG FKSYATRTVI TDWDPQFNAI TGLNGSGKSN ILDAICFVLG IASMSTVRAS SLQDLIYKRG QAGVTKASVT IVFDNTDKS NSPIGFTNSP QISVTRQVVL GGTSKYLING HRAPQQSVLQ LFQSVQLNIN NPNFLIMQGK ITKVLNMKPS E ILSLIEEA ...文字列: MKVEELIIDG FKSYATRTVI TDWDPQFNAI TGLNGSGKSN ILDAICFVLG IASMSTVRAS SLQDLIYKRG QAGVTKASVT IVFDNTDKS NSPIGFTNSP QISVTRQVVL GGTSKYLING HRAPQQSVLQ LFQSVQLNIN NPNFLIMQGK ITKVLNMKPS E ILSLIEEA AGTKMFEDRR EKAERTMSKK ETKLQENRTL LTEEIEPKLE KLRNEKRMFL EFQSTQTDLE KTERIVVSYE YY NIKHKHT SIRETLENGE TRMKMLNEFV KKTSEEIDSL NEDVEEIKLQ KEKELHKEGT ISKLENKENG LLNEISRLKT SLS IKVENL NDTTEKSKAL ESEIASSSAK LIEKKSAYAN TEKDYKMVQE QLSKQRDLYK RKEELVSTLT TGISSTGAAD GGYN AQLAK AKTELNEVSL AIKKSSMKME LLKKELLTIE PKLKEATKDN ELNVKHVKQC QETCDKLRAR LVEYGFDPSR IKDLK QRED KLKSHYYQTC KNSEYLKRRV TNLEFNYTKP YPNFEASFVH GVVGQLFQID NDNIRYATAL QTCAGGRLFN VVVQDS QTA TQLLERGRLR KRVTIIPLDK IYTRPISSQV LDLAKKIAPG KVELAINLIR FDESITKAME FIFGNSLICE DPETAKK IT FHPKIRARSI TLQGDVYDPE GTLSGGSRNT SESLLVDIQK YNQIQKQIET IQADLNHVTE ELQTQYATSQ KTKTIQSD L NLSLHKLDLA KRNLDANPSS QIIARNEEIL RDIGECENEI KTKQMSLKKC QEEVSTIEKD MKEYDSDKGS KLNELKKEL KLLAKELEEQ ESESERKYDL FQNLELETEQ LSSELDSNKT LLHNHLKSIE SLKLENSDLE GKIRGVEDDL VTVQTELNEE KKRLMDIDD ELNELETLIK KKQDEKKSSE LELQKLVHDL NKYKSNTNNM EKIIEDLRQK HEFLEDFDLV RNIVKQNEGI D LDTYRERS KQLNEKFQEL RKKVNPNIMN MIENVEKKEA ALKTMIKTIE KDKMKIQETI SKLNEYKRET LVKTWEKVTL DF GNIFADL LPNSFAKLVP CEGKDVTQGL EVKVKLGNIW KESLIELSGG QRSLIALSLI MALLQFRPAP MYILDQVDAA LDL SHTQNI GHLIKTRFKG SQFIVVSLKE GMFANANRVF RTRFQDGTSV VSIM UniProtKB: Structural maintenance of chromosomes protein 2 |
-分子 #5: Condensin complex subunit 2
| 分子 | 名称: Condensin complex subunit 2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 92.721219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTTQLRYENN DDDERVEYNL FTNRSTMMAN FEEWIKMATD NKINSRNSWN FALIDYFYDL DVLKDGENNI NFQKASATLD GCIKIYSSR VDSVTTETGK LLSGLAQRKT NGASNGDDSN GGNGEGLGGD SDEANIEIDP LTGMPISNDP DVNNTRRRVY N RVLETTLV ...文字列: MTTQLRYENN DDDERVEYNL FTNRSTMMAN FEEWIKMATD NKINSRNSWN FALIDYFYDL DVLKDGENNI NFQKASATLD GCIKIYSSR VDSVTTETGK LLSGLAQRKT NGASNGDDSN GGNGEGLGGD SDEANIEIDP LTGMPISNDP DVNNTRRRVY N RVLETTLV EFETIKMKEL DQELIIDPLF KKALVDFDEG GAKSLLLNTL NIDNTARVIF DASIKDTQNV GQGKLQRKEE EL IERDSLV DDENEPSQSL ISTRNDSTVN DSVISAPSME DEILSLGMDF IKFDQIAVCE ISGSIEQLRN VVEDINQAKD FIE NVNNRF DNFLTEEELQ AAVPDNAEDD SDGFDMGMQQ ELCYPDENHD NTSHDEQDDD NVNSTTGSIF EKDLMAYFDE NLNR NWRGR EHWKVRNFKK ANLVNKESDL LEETRTTIGD TTDKNTTDDK SMDTKKKHKQ KKVLEIDFFK TDDSFEDKVF ASKGR TKID MPIKNRKNDT HYLLPDDFHF STDRITRLFI KPAQKMSLFS HRKHTRGDVS SGLFEKSTVS ANHSNNDIPT IADEHF WAD NYERKEQEEK EKEQSKEVGD VVGGALDNPF EDDMDGVDFN QAFEGTDDNE EASVKLDLQD DEDHKFPIRE NKVTYSR VS KKVDVRRLKK NVWRSINNLI QEHDSRKNRE QSSNDSETHT EDESTKELKF SDIIQGISKM YSDDTLKDIS TSFCFICL L HLANEHGLQI THTENYNDLI VNYEDLATTQ AASLVGGGHH RPHHGGHHHH HHGGRIFYPY DVPDYAGYPY DVPDYAGSY PYDVPNYAAG H UniProtKB: Condensin complex subunit 2 |
-分子 #6: Condensin complex subunit 1
| 分子 | 名称: Condensin complex subunit 1 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 133.116766 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGFSLSEYL TKFQTTDRES YPRLQDPSRE LNVIIDQLAV SPEQIDASPD SLEALIDLCH DFPHLTPKLQ TQLSYLISSS LSNLSKDIK ANLSSNVNFT EIGGLIPQWK RHLEEYGYLI QVLLTFLQDE LHKVSSQSTN LNRSAKNSKN DSANVELFKR D CNQMENLL ...文字列: MSGFSLSEYL TKFQTTDRES YPRLQDPSRE LNVIIDQLAV SPEQIDASPD SLEALIDLCH DFPHLTPKLQ TQLSYLISSS LSNLSKDIK ANLSSNVNFT EIGGLIPQWK RHLEEYGYLI QVLLTFLQDE LHKVSSQSTN LNRSAKNSKN DSANVELFKR D CNQMENLL ESITKLLEIN LSKIFQTTPE KDLFIGLFTR PLFVLLEIEP VTKVSSLKMF IQRILAMCVK NHGQSSSIQS SL MTNLTYF LHLSVFNAEL LKLLNDEYNY PQLTEDILKE ISTRVFNAKD TTGPKAISNF LIKLSELSPG IMLRQMNLVI TLL NNSSIT LRCSVVEACG NIVAELAQDP QTMEHYKQQI AVLIELLEER FQDSNPYVRT KAIQGCSKIC DLSSKFNKSK AKFT SLAVR SLQDRSSLVR RNSVKLLSKL LLKHPFKAIH GSQLRLSEWE EYLKGSESQL NSTLKKVESQ ETLNDTIERS LIEEE VEQD EGQCRTELEG SFNKSAELSR IENEVENINA TNTSVLMKLK LMIVYYKDAI SFIKEIHKSI ELISNLLFSK NRNEVL ESM DFLVLADAFD IELSEFGIKK MLHLVWMKGT NDEGTSISVH LIECYKQLFL TAPDSCNMQE KAAHIAKNLI NLSIGAS IA DLASLEQLLG MMYEQKLIDQ HVINILWAIY NSASKASMQK EQNVNNRDSE KGFSKEQIHG SIIILGMLSL ADNEIALK G LESLLNIGLG AVGLKDLTLC RYSCLALERM VPKRSTIITK AINQELEDVA VKKLYAIIIN YTKDNEYYPM CEQALSALF TISSKPDILA TDLIREKTMM TFGKPEEEDS ILSLEQSSRV VSLSQLLFIV GQVAIKTLVY LEKCEAEFKK RKIEAETRNG KVKNQGADV TNTTQDNGGD KELEMIGGTN EDDFTDAIQF VKENELLFGE KSILGKFCPI VEEIVSNSSR FSDPMLQRTA T LCLEKLMC LSSKYCEKSL PLLITVMEKS PDPTIRSNAV LGLGDMAVCF NNLVDENTDY LYRRLHDENL MVQRTCLMTV TF LILAGQV KVKGQLGEMA KCLDNPDQGI SDMCRLFFTE LASKDNAIYN GFIDIFSNLS SDDLLGKESF KKIIKFLLTF IDK ERHQKQ LNEKLVGRLR KCETQKQWDD IAFVLNNLPY KNEDVTALLE QGFKVVSAKE UniProtKB: Condensin complex subunit 1 |
-分子 #7: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 2 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #8: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 8 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.645 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 2 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: OTHER | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 6544 / 平均露光時間: 8.0 sec. / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 130000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X