+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13544 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HsPepT2 bound to Ala-Phe in the inward facing partially occluded conformation | |||||||||
 マップデータ マップデータ | post processed in Phenix. Used for refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HsPepT2 / PepT2 / Peptide transporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tripeptide import across plasma membrane / dipeptide transport / Proton/oligopeptide cotransporters / peptidoglycan transport / tripeptide transmembrane transporter activity / dipeptide import across plasma membrane / peptide:proton symporter activity / dipeptide transmembrane transporter activity / antibacterial innate immune response / regulation of nucleotide-binding domain, leucine rich repeat containing receptor signaling pathway ...tripeptide import across plasma membrane / dipeptide transport / Proton/oligopeptide cotransporters / peptidoglycan transport / tripeptide transmembrane transporter activity / dipeptide import across plasma membrane / peptide:proton symporter activity / dipeptide transmembrane transporter activity / antibacterial innate immune response / regulation of nucleotide-binding domain, leucine rich repeat containing receptor signaling pathway / xenobiotic detoxification by transmembrane export across the plasma membrane / peptide transport / xenobiotic transport / renal absorption / transport across blood-brain barrier / monoatomic ion transport / phagocytic vesicle membrane / protein transport / apical plasma membrane / extracellular exosome / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
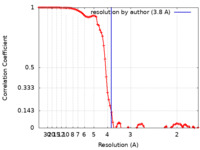
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Killer M / Wald J | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Structural snapshots of human PepT1 and PepT2 reveal mechanistic insights into substrate and drug transport across epithelial membranes. 著者: Maxime Killer / Jiri Wald / Joanna Pieprzyk / Thomas C Marlovits / Christian Löw /  要旨: The uptake of peptides in mammals plays a crucial role in nutrition and inflammatory diseases. This process is mediated by promiscuous transporters of the solute carrier family 15, which form part of ...The uptake of peptides in mammals plays a crucial role in nutrition and inflammatory diseases. This process is mediated by promiscuous transporters of the solute carrier family 15, which form part of the major facilitator superfamily. Besides the uptake of short peptides, peptide transporter 1 (PepT1) is a highly abundant drug transporter in the intestine and represents a major route for oral drug delivery. PepT2 also allows renal drug reabsorption from ultrafiltration and brain-to-blood efflux of neurotoxic compounds. Here, we present cryogenic electron microscopy (cryo-EM) structures of human PepT1 and PepT2 captured in four different states throughout the transport cycle. The structures reveal the architecture of human peptide transporters and provide mechanistic insights into substrate recognition and conformational transitions during transport. This may support future drug design efforts to increase the bioavailability of different drugs in the human body. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13544.map.gz emd_13544.map.gz | 57.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13544-v30.xml emd-13544-v30.xml emd-13544.xml emd-13544.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13544_fsc.xml emd_13544_fsc.xml | 8.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13544.png emd_13544.png | 71.3 KB | ||
| マスクデータ |  emd_13544_msk_1.map emd_13544_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13544.cif.gz emd-13544.cif.gz | 5.7 KB | ||
| その他 |  emd_13544_additional_1.map.gz emd_13544_additional_1.map.gz emd_13544_half_map_1.map.gz emd_13544_half_map_1.map.gz emd_13544_half_map_2.map.gz emd_13544_half_map_2.map.gz | 56.5 MB 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13544 http://ftp.pdbj.org/pub/emdb/structures/EMD-13544 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13544 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13544 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13544_validation.pdf.gz emd_13544_validation.pdf.gz | 788.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13544_full_validation.pdf.gz emd_13544_full_validation.pdf.gz | 787.8 KB | 表示 | |
| XML形式データ |  emd_13544_validation.xml.gz emd_13544_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_13544_validation.cif.gz emd_13544_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13544 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13544 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13544 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13544 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13544.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13544.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post processed in Phenix. Used for refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13544_msk_1.map emd_13544_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_13544_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13544_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13544_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human PepT2
| 全体 | 名称: Human PepT2 |
|---|---|
| 要素 |
|
-超分子 #1: Human PepT2
| 超分子 | 名称: Human PepT2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: ALA-PHE
| 超分子 | 名称: ALA-PHE / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Solute carrier family 15 member 2
| 分子 | 名称: Solute carrier family 15 member 2 / タイプ: protein_or_peptide / ID: 1 / 詳細: HsPepT2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 81.861125 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MNPFQKNESK ETLFSPVSIE EVPPRPPSPP KKPSPTICGS NYPLSIAFIV VNEFCERFSY YGMKAVLILY FLYFLHWNED TSTSIYHAF SSLCYFTPIL GAAIADSWLG KFKTIIYLSL VYVLGHVIKS LGALPILGGQ VVHTVLSLIG LSLIALGTGG I KPCVAAFG ...文字列: MNPFQKNESK ETLFSPVSIE EVPPRPPSPP KKPSPTICGS NYPLSIAFIV VNEFCERFSY YGMKAVLILY FLYFLHWNED TSTSIYHAF SSLCYFTPIL GAAIADSWLG KFKTIIYLSL VYVLGHVIKS LGALPILGGQ VVHTVLSLIG LSLIALGTGG I KPCVAAFG GDQFEEKHAE ERTRYFSVFY LSINAGSLIS TFITPMLRGD VQCFGEDCYA LAFGVPGLLM VIALVVFAMG SK IYNKPPP EGNIVAQVFK CIWFAISNRF KNRSGDIPKR QHWLDWAAEK YPKQLIMDVK ALTRVLFLYI PLPMFWALLD QQG SRWTLQ AIRMNRNLGF FVLQPDQMQV LNPLLVLIFI PLFDFVIYRL VSKCGINFSS LRKMAVGMIL ACLAFAVAAA VEIK INEMA PAQPGPQEVF LQVLNLADDE VKVTVVGNEN NSLLIESIKS FQKTPHYSKL HLKTKSQDFH FHLKYHNLSL YTEHS VQEK NWYSLVIRED GNSISSMMVK DTESRTTNGM TTVRFVNTLH KDVNISLSTD TSLNVGEDYG VSAYRTVQRG EYPAVH CRT EDKNFSLNLG LLDFGAAYLF VITNNTNQGL QAWKIEDIPA NKMSIAWQLP QYALVTAGEV MFSVTGLEFS YSQAPSS MK SVLQAAWLLT IAVGNIIVLV VAQFSGLVQW AEFILFSCLL LVICLIFSIM GYYYVPVKTE DMRGPADKHI PHIQGNMI K LETKKTKL UniProtKB: Solute carrier family 15 member 2 |
-分子 #2: ALA-PHE
| 分子 | 名称: ALA-PHE / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 236.267 Da |
| 配列 | 文字列: AF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 81.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)