+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10258 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab | ||||||||||||
 マップデータ マップデータ | Nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab | ||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dolichyl-P-Glc:Man9GlcNAc2-PP-dolichol alpha-1,3-glucosyltransferase / oligosaccharide-lipid intermediate biosynthetic process / dolichyl pyrophosphate Man9GlcNAc2 alpha-1,3-glucosyltransferase activity / Biosynthesis of the N-glycan precursor (dolichol lipid-linked oligosaccharide, LLO) and transfer to a nascent protein / dolichol-linked oligosaccharide biosynthetic process / hexosyltransferase activity / protein N-linked glycosylation / protein glycosylation / aerobic respiration / endoplasmic reticulum membrane / endoplasmic reticulum 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
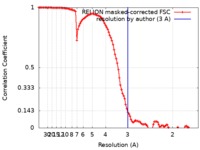
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||
 データ登録者 データ登録者 | Bloch JS / Pesciullesi G / Boilevin J / Nosol K / Irobalieva RN / Darbre T / Aebi M / Kossiakoff AA / Reymond JL / Locher KP | ||||||||||||
| 資金援助 |  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structure and mechanism of the ER-based glucosyltransferase ALG6. 著者: Joël S Bloch / Giorgio Pesciullesi / Jérémy Boilevin / Kamil Nosol / Rossitza N Irobalieva / Tamis Darbre / Markus Aebi / Anthony A Kossiakoff / Jean-Louis Reymond / Kaspar P Locher /   要旨: In eukaryotic protein N-glycosylation, a series of glycosyltransferases catalyse the biosynthesis of a dolichylpyrophosphate-linked oligosaccharide before its transfer onto acceptor proteins. The ...In eukaryotic protein N-glycosylation, a series of glycosyltransferases catalyse the biosynthesis of a dolichylpyrophosphate-linked oligosaccharide before its transfer onto acceptor proteins. The final seven steps occur in the lumen of the endoplasmic reticulum (ER) and require dolichylphosphate-activated mannose and glucose as donor substrates. The responsible enzymes-ALG3, ALG9, ALG12, ALG6, ALG8 and ALG10-are glycosyltransferases of the C-superfamily (GT-Cs), which are loosely defined as containing membrane-spanning helices and processing an isoprenoid-linked carbohydrate donor substrate. Here we present the cryo-electron microscopy structure of yeast ALG6 at 3.0 Å resolution, which reveals a previously undescribed transmembrane protein fold. Comparison with reported GT-C structures suggests that GT-C enzymes contain a modular architecture with a conserved module and a variable module, each with distinct functional roles. We used synthetic analogues of dolichylphosphate-linked and dolichylpyrophosphate-linked sugars and enzymatic glycan extension to generate donor and acceptor substrates using purified enzymes of the ALG pathway to recapitulate the activity of ALG6 in vitro. A second cryo-electron microscopy structure of ALG6 bound to an analogue of dolichylphosphate-glucose at 3.9 Å resolution revealed the active site of the enzyme. Functional analysis of ALG6 variants identified a catalytic aspartate residue that probably acts as a general base. This residue is conserved in the GT-C superfamily. Our results define the architecture of ER-luminal GT-C enzymes and provide a structural basis for understanding their catalytic mechanisms. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10258.map.gz emd_10258.map.gz | 96.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10258-v30.xml emd-10258-v30.xml emd-10258.xml emd-10258.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10258_fsc.xml emd_10258_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10258.png emd_10258.png | 110.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10258 http://ftp.pdbj.org/pub/emdb/structures/EMD-10258 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10258 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10258 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10258_validation.pdf.gz emd_10258_validation.pdf.gz | 292.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10258_full_validation.pdf.gz emd_10258_full_validation.pdf.gz | 291.5 KB | 表示 | |
| XML形式データ |  emd_10258_validation.xml.gz emd_10258_validation.xml.gz | 11.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10258 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10258 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10258 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10258 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6sniMC  6snhC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10429 (タイトル: 3 Å resolution single particle reconstruction of glucosyltransferase ALG6 in nanodisc EMPIAR-10429 (タイトル: 3 Å resolution single particle reconstruction of glucosyltransferase ALG6 in nanodiscData size: 2.8 TB Data #1: Motion corrected micrographs of the Alg6-Fab complex [micrographs - single frame] Data #2: Unaligned multi-frame micrographs of the Alg6-Fab complex [micrographs - multiframe] Data #3: Unaligned multi-frame micrographs of the Alg6-Fab complex [micrographs - multiframe] Data #4: Unaligned multi-frame micrographs of the Alg6-Fab complex [micrographs - multiframe] Data #5: Unaligned multi-frame micrographs of the Alg6-Fab complex [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10258.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10258.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab
| 全体 | 名称: Nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab |
|---|---|
| 要素 |
|
-超分子 #1: Nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab
| 超分子 | 名称: Nanodisc reconstituted yeast ALG6 in complex with 6AG9 Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 112.89472 KDa |
-超分子 #2: ALG6
| 超分子 | 名称: ALG6 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
-超分子 #3: 6AG9 Fab
| 超分子 | 名称: 6AG9 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
-分子 #1: Dolichyl pyrophosphate Man9GlcNAc2 alpha-1,3-glucosyltransferase
| 分子 | 名称: Dolichyl pyrophosphate Man9GlcNAc2 alpha-1,3-glucosyltransferase タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: dolichyl-P-Glc:Man9GlcNAc2-PP-dolichol alpha-1,3-glucosyltransferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 64.423223 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPGGSTGRLA GAGGEFVDMA IGKRLLVNKP AEESFYASPM YDFLYPFRPV GNQWLPEYII FVCAVILRCT IGLGPYSGKG SPPLYGDFE AQRHWMEITQ HLPLSKWYWY DLQYWGLDYP PLTAFHSYLL GLIGSFFNPS WFALEKSRGF ESPDNGLKTY M RSTVIISD ...文字列: GPGGSTGRLA GAGGEFVDMA IGKRLLVNKP AEESFYASPM YDFLYPFRPV GNQWLPEYII FVCAVILRCT IGLGPYSGKG SPPLYGDFE AQRHWMEITQ HLPLSKWYWY DLQYWGLDYP PLTAFHSYLL GLIGSFFNPS WFALEKSRGF ESPDNGLKTY M RSTVIISD ILFYFPAVIY FTKWLGRYRN QSPIGQSIAA SAILFQPSLM LIDHGHFQYN SVMLGLTAYA INNLLDEYYA MA AVCFVLS ICFKQMALYY APIFFAYLLS RSLLFPKFNI ARLTVIAFAT LATFAIIFAP LYFLGGGLKN IHQCIHRIFP FAR GIFEDK VANFWCVTNV FVKYKERFTI QQLQLYSLIA TVIGFLPAMI MTLLHPKKHL LPYVLIACSM SFFLFSFQVH EKTI LIPLL PITLLYSSTD WNVLSLVSWI NNVALFTLWP LLKKDGLHLQ YAVSFLLSNW LIGNFSFITP RFLPKSLTPG PSISS INSD YRRRSLLPYN VVWKSFIIGT YIAMGFYHFL DQFVAPPSKY PDLWVLLNCA VGFICFSIFW LWSYYKIFTS GSKSMK DL |
-分子 #2: 6AG9-Fab heavy chain
| 分子 | 名称: 6AG9-Fab heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 24.953664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVYSSSIHWV RQAPGKGLEW VASISSYSGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC AREYWSWYSY SYGIDYWGQG TLVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF P EPVTVSWN ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVYSSSIHWV RQAPGKGLEW VASISSYSGY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC AREYWSWYSY SYGIDYWGQG TLVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF P EPVTVSWN SGALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKKVEPKS CDKTHT |
-分子 #3: 6AG9-Fab light chain
| 分子 | 名称: 6AG9-Fab light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 23.65025 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSYWVGYPI TFGQGTKVEI KRTVAAPSVF IFPPSDSQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS G NSQESVTE ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSYWVGYPI TFGQGTKVEI KRTVAAPSVF IFPPSDSQLK SGTASVVCLL NNFYPREAKV QWKVDNALQS G NSQESVTE QDSKDSTYSL SSTLTLSKAD YEKHKVYACE VTHQGLSSPV TKSFNRGEC |
-分子 #4: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 4 / コピー数: 5 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-分子 #5: CHOLESTEROL HEMISUCCINATE
| 分子 | 名称: CHOLESTEROL HEMISUCCINATE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: Y01 |
|---|---|
| 分子量 | 理論値: 486.726 Da |
| Chemical component information |  ChemComp-Y01: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 7 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 2.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー