+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10193 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Apo2 state of the heptameric Bcs1 AAA-ATPase (C7 and C1 symmetrized maps). | |||||||||
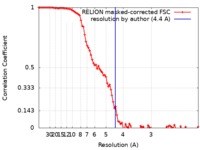
 マップデータ マップデータ | C7 symmetrized map of the Bcs1 Apo1 state resolved at 4.4 A after relion postprocessing. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | translocation / Rieske / mitochondira / inner mitochondiral membrane / TRANSLOCASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein insertion into mitochondrial inner membrane from matrix / mitochondrial respiratory chain complex III assembly / ATPase-coupled transmembrane transporter activity / protein transmembrane transporter activity / chaperone-mediated protein complex assembly / mitochondrial intermembrane space / mitochondrial inner membrane / ATP hydrolysis activity / mitochondrion / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Kater L / Beckmann R | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Structure of the Bcs1 AAA-ATPase suggests an airlock-like translocation mechanism for folded proteins. 著者: Lukas Kater / Nikola Wagener / Otto Berninghausen / Thomas Becker / Walter Neupert / Roland Beckmann /  要旨: Some proteins require completion of folding before translocation across a membrane into another cellular compartment. Yet the permeability barrier of the membrane should not be compromised and ...Some proteins require completion of folding before translocation across a membrane into another cellular compartment. Yet the permeability barrier of the membrane should not be compromised and mechanisms have remained mostly elusive. Here, we present the structure of Saccharomyces cerevisiae Bcs1, an AAA-ATPase of the inner mitochondrial membrane. Bcs1 facilitates the translocation of the Rieske protein, Rip1, which requires folding and incorporation of a 2Fe-2S cluster before translocation and subsequent integration into the bc1 complex. Surprisingly, Bcs1 assembles into exclusively heptameric homo-oligomers, with each protomer consisting of an amphipathic transmembrane helix, a middle domain and an ATPase domain. Together they form two aqueous vestibules, the first being accessible from the mitochondrial matrix and the second positioned in the inner membrane, with both separated by the seal-forming middle domain. On the basis of this unique architecture, we propose an airlock-like translocation mechanism for folded Rip1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10193.map.gz emd_10193.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10193-v30.xml emd-10193-v30.xml emd-10193.xml emd-10193.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10193_fsc.xml emd_10193_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10193.png emd_10193.png | 175.2 KB | ||
| Filedesc metadata |  emd-10193.cif.gz emd-10193.cif.gz | 5.2 KB | ||
| その他 |  emd_10193_additional_1.map.gz emd_10193_additional_1.map.gz emd_10193_additional_2.map.gz emd_10193_additional_2.map.gz | 40.4 MB 40.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10193 http://ftp.pdbj.org/pub/emdb/structures/EMD-10193 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10193 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10193 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10193_validation.pdf.gz emd_10193_validation.pdf.gz | 582.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10193_full_validation.pdf.gz emd_10193_full_validation.pdf.gz | 582.2 KB | 表示 | |
| XML形式データ |  emd_10193_validation.xml.gz emd_10193_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_10193_validation.cif.gz emd_10193_validation.cif.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10193 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10193 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10193 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10193 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10193.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10193.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C7 symmetrized map of the Bcs1 Apo1 state resolved at 4.4 A after relion postprocessing. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: C7 symmetrized map of the Bcs1 Apo1 state...
| ファイル | emd_10193_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C7 symmetrized map of the Bcs1 Apo1 state resolved at 4.4 A after relion local resolution filtering. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: C7 symmetrized map of the Bcs1 Apo1 state...
| ファイル | emd_10193_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | C7 symmetrized map of the Bcs1 Apo1 state resolved at 4.4 A after relion local resolution filtering. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heptameric complex of ADP bound Bcs1
| 全体 | 名称: Heptameric complex of ADP bound Bcs1 |
|---|---|
| 要素 |
|
-超分子 #1: Heptameric complex of ADP bound Bcs1
| 超分子 | 名称: Heptameric complex of ADP bound Bcs1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Mitochondrial chaperone BCS1
| 分子 | 名称: Mitochondrial chaperone BCS1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 51.172336 KDa |
| 配列 | 文字列: MSDKPIDIQY DKQATPNLSG VITPPTNETG NDSVREKLSK LVGDAMSNNP YFAAGGGLMI LGTGLAVARS GIIKASRVLY RQMIVDLEI QSKDKSYAWF LTWMAKHPQR VSRHLSVRTN YIQHDNGSVS TKFSLVPGPG NHWIRYKGAF ILIKRERSAK M IDIANGSP ...文字列: MSDKPIDIQY DKQATPNLSG VITPPTNETG NDSVREKLSK LVGDAMSNNP YFAAGGGLMI LGTGLAVARS GIIKASRVLY RQMIVDLEI QSKDKSYAWF LTWMAKHPQR VSRHLSVRTN YIQHDNGSVS TKFSLVPGPG NHWIRYKGAF ILIKRERSAK M IDIANGSP FETVTLTTLY RDKHLFDDIL NEAKDIALKT TEGKTVIYTS FGPEWRKFGQ PKAKRMLPSV ILDSGIKEGI LD DVYDFMK NGKWYSDRGI PYRRGYLLYG PPGSGKTSFI QALAGELDYN ICILNLSENN LTDDRLNHLM NNMPERSILL LED IDAAFN KRSQTGEQGF HSSVTFSGLL NALDGVTSSE ETITFMTTNH PEKLDAAIMR PGRIDYKVFV GNATPYQVEK MFMK FYPGE TDICKKFVNS VKELDITVST AQLQGLFVMN KDAPHDALKM VSSLRNANHI F UniProtKB: Mitochondrial chaperone BCS1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 12.0 sec. / 平均電子線量: 65.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)