+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9996 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
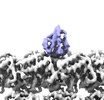
| タイトル | Complex of yeast cytoplasmic dynein MTBD-High and MT without DTT | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MOTOR PROTEIN-STRUCTURAL PROTEIN COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報karyogamy / astral microtubule / establishment of mitotic spindle localization / nuclear migration along microtubule / spindle pole body / minus-end-directed microtubule motor activity / dynein light intermediate chain binding / cytoplasmic dynein complex / motile cilium / dynein intermediate chain binding ...karyogamy / astral microtubule / establishment of mitotic spindle localization / nuclear migration along microtubule / spindle pole body / minus-end-directed microtubule motor activity / dynein light intermediate chain binding / cytoplasmic dynein complex / motile cilium / dynein intermediate chain binding / nuclear migration / establishment of mitotic spindle orientation / mitotic sister chromatid segregation / cytoplasmic microtubule / cytoplasmic microtubule organization / Neutrophil degranulation / mitotic spindle organization / structural constituent of cytoskeleton / microtubule cytoskeleton organization / mitotic cell cycle / cell cortex / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / microtubule / hydrolase activity / GTPase activity / GTP binding / ATP hydrolysis activity / ATP binding / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.94 Å | |||||||||
 データ登録者 データ登録者 | Komori Y / Nishida N | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural basis for two-way communication between dynein and microtubules. 著者: Noritaka Nishida / Yuta Komori / Osamu Takarada / Atsushi Watanabe / Satoko Tamura / Satoshi Kubo / Ichio Shimada / Masahide Kikkawa /  要旨: The movements of cytoplasmic dynein on microtubule (MT) tracks is achieved by two-way communication between the microtubule-binding domain (MTBD) and the ATPase domain via a coiled-coil stalk, but ...The movements of cytoplasmic dynein on microtubule (MT) tracks is achieved by two-way communication between the microtubule-binding domain (MTBD) and the ATPase domain via a coiled-coil stalk, but the structural basis of this communication remains elusive. Here, we regulate MTBD either in high-affinity or low-affinity states by introducing a disulfide bond to the stalk and analyze the resulting structures by NMR and cryo-EM. In the MT-unbound state, the affinity changes of MTBD are achieved by sliding of the stalk α-helix by a half-turn, which suggests that structural changes propagate from the ATPase-domain to MTBD. In addition, MT binding induces further sliding of the stalk α-helix even without the disulfide bond, suggesting how the MT-induced conformational changes propagate toward the ATPase domain. Based on differences in the MT-binding surface between the high- and low-affinity states, we propose a potential mechanism for the directional bias of dynein movement on MT tracks. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9996.map.gz emd_9996.map.gz | 14.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9996-v30.xml emd-9996-v30.xml emd-9996.xml emd-9996.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9996.png emd_9996.png | 104.2 KB | ||
| Filedesc metadata |  emd-9996.cif.gz emd-9996.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9996 http://ftp.pdbj.org/pub/emdb/structures/EMD-9996 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9996 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9996 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9996_validation.pdf.gz emd_9996_validation.pdf.gz | 490 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9996_full_validation.pdf.gz emd_9996_full_validation.pdf.gz | 489.6 KB | 表示 | |
| XML形式データ |  emd_9996_validation.xml.gz emd_9996_validation.xml.gz | 5.7 KB | 表示 | |
| CIF形式データ |  emd_9996_validation.cif.gz emd_9996_validation.cif.gz | 6.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9996 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9996 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9996 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9996 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6kioMC  9997C  6kiqC  6kjnC  6kjoC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10657 (タイトル: Complex of yeast cytoplasmic dynein MTBD-High and MT without DTT EMPIAR-10657 (タイトル: Complex of yeast cytoplasmic dynein MTBD-High and MT without DTTData size: 360.5 Data #1: Unaligned multi-frame micrographs of the complex of yeast cytoplasmic dynein MTBD-High and MT without DTT [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9996.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9996.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : MTBD-High/MT complex without DTT
| 全体 | 名称: MTBD-High/MT complex without DTT |
|---|---|
| 要素 |
|
-超分子 #1: MTBD-High/MT complex without DTT
| 超分子 | 名称: MTBD-High/MT complex without DTT / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Tubulin beta chain
| 分子 | 名称: Tubulin beta chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 47.809746 KDa |
| 配列 | 文字列: REIVHIQAGQ CGNQIGAKFW EVISDEHGID PTGSYHGDSD LQLERINVYY NEAAGNKYVP RAILVDLEPG TMDSVRSGPF GQIFRPDNF VFGQSGAGNN WAKGHYTEGA ELVDSVLDVV RKESESCDCL QGFQLTHSLG GGTGSGMGTL LISKIREEYP D RIMNTFSV ...文字列: REIVHIQAGQ CGNQIGAKFW EVISDEHGID PTGSYHGDSD LQLERINVYY NEAAGNKYVP RAILVDLEPG TMDSVRSGPF GQIFRPDNF VFGQSGAGNN WAKGHYTEGA ELVDSVLDVV RKESESCDCL QGFQLTHSLG GGTGSGMGTL LISKIREEYP D RIMNTFSV VPSPKVSDTV VEPYNATLSV HQLVENTDET YCIDNEALYD ICFRTLKLTT PTYGDLNHLV SATMSGVTTC LR FPGQLNA DLRKLAVNMV PFPRLHFFMP GFAPLTSRGS QQYRALTVPE LTQQMFDAKN MMAACDPRHG RYLTVAAVFR GRM SMKEVD EQMLNVQNKN SSYFVEWIPN NVKTAVCDIP PRGLKMSATF IGNSTAIQEL FKRISEQFTA MFRRKAFLHW YTGE GMDEM EFTEAESNMN DLVSEYQQYQ D UniProtKB: Tubulin beta chain |
-分子 #2: Dynein heavy chain, cytoplasmic
| 分子 | 名称: Dynein heavy chain, cytoplasmic / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.264564 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSIQDCEPT ILEAQRGVKN IKKQQLTEIR SMVNPPSGVK IVMEAVCAIL GYQFSNWRDI QQFIRKDDFI HNIVHYDTTL HMKPQIRKY MEEEFLSDPN FTYETINRAS KACGPLYQWV NAQINFSKCL E UniProtKB: Dynein heavy chain, cytoplasmic |
-分子 #3: Tubulin alpha-1A chain
| 分子 | 名称: Tubulin alpha-1A chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.95998 KDa |
| 配列 | 文字列: RECISIHVGQ AGVQIGNACW ELYCLEHGIQ PDGHVPRAVF VDLEPTVIDE VRTGTYRQLF HPEQLITGKE DAANNYARGH YTIGKEIID LVLDRIRKLA DQCTGLQGFS VFHSFGGGTG SGFTSLLMER LSVDYGKKSK LEFSIYPAPQ VSTAVVEPYN S ILTTHTTL ...文字列: RECISIHVGQ AGVQIGNACW ELYCLEHGIQ PDGHVPRAVF VDLEPTVIDE VRTGTYRQLF HPEQLITGKE DAANNYARGH YTIGKEIID LVLDRIRKLA DQCTGLQGFS VFHSFGGGTG SGFTSLLMER LSVDYGKKSK LEFSIYPAPQ VSTAVVEPYN S ILTTHTTL EHSDCAFMVD NEAIYDICRR NLDIERPTYT NLNRLIGQIV SSITASLRFD GALNVDLTEF QTNLVPYPRG HF PLATYAP VISAEKAYHE QLSVAEITNA CFEPANQMVK CDPRHGKYMA CCLLYRGDVV PKDVNAAIAT IKTKRTIQFV DWC PTGFKV GINYEPPTVV PGGDLAKVQR AVCMLSNTTA IAEAWARLDH KFDLMYAKRA FVHWYVGEGM EEGEFSEARE DMAA LEKDY EEVGVDS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER/RHODIUM | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 1820 / 平均露光時間: 10.0 sec. / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.25 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 9.20776 Å 想定した対称性 - らせんパラメータ - ΔΦ: -25.7561 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.94 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 詳細: FSCtrue was calculated to validate the resolution. / 使用した粒子像数: 58999 |
|---|---|
| Segment selection | 選択した数: 76377 / 詳細: particles were collected using PyFilamentPicker |
| 初期モデル | モデルのタイプ: OTHER / 詳細: Cryo-EM map of microtubule filtered to 8 Angstroms |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: FREALIGN (ver. 9.11) 詳細: FREALIGN v9.11 and "super-particle" based approach were used to refine the angular assignment |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6kio: |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















