+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Pseudomonas aeruginosa TonB-dependent transporter PhuR in complex with synthetic antibody and heme | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | beta barrel / outer membrane protein / heme binding protein / heme transport / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報heme transport / siderophore transmembrane transport / heme transmembrane transporter activity / siderophore uptake transmembrane transporter activity / outer membrane / cellular response to iron ion / cell outer membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Knejski P / Erramilli SK / Kossiakoff AA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Chaperone-assisted cryo-EM structure of P. aeruginosa PhuR reveals molecular basis for heme binding. 著者: Paweł P Knejski / Satchal K Erramilli / Anthony A Kossiakoff /   要旨: Pathogenic bacteria, such as Pseudomonas aeruginosa, depend on scavenging heme for the acquisition of iron, an essential nutrient. The TonB-dependent transporter (TBDT) PhuR is the major heme uptake ...Pathogenic bacteria, such as Pseudomonas aeruginosa, depend on scavenging heme for the acquisition of iron, an essential nutrient. The TonB-dependent transporter (TBDT) PhuR is the major heme uptake protein in P. aeruginosa clinical isolates. However, a comprehensive understanding of heme recognition and TBDT transport mechanisms, especially PhuR, remains limited. In this study, we employed single-particle cryogenic electron microscopy (cryo-EM) and a phage display-generated synthetic antibody (sAB) as a fiducial marker to enable the determination of a high-resolution (2.5 Å) structure of PhuR with a bound heme. Notably, the structure reveals iron coordination by Y529 on a conserved extracellular loop, shedding light on the role of tyrosine in heme binding. Biochemical assays and negative-stain EM demonstrated that the sAB specifically targets the heme-bound state of PhuR. These findings provide insights into PhuR's heme binding and offer a template for developing conformation-specific sABs against outer membrane proteins (OMPs) for structure-function investigations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
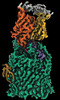
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41255.map.gz emd_41255.map.gz | 168.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41255-v30.xml emd-41255-v30.xml emd-41255.xml emd-41255.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41255.png emd_41255.png | 115.3 KB | ||
| マスクデータ |  emd_41255_msk_1.map emd_41255_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41255.cif.gz emd-41255.cif.gz | 6.7 KB | ||
| その他 |  emd_41255_half_map_1.map.gz emd_41255_half_map_1.map.gz emd_41255_half_map_2.map.gz emd_41255_half_map_2.map.gz | 165.1 MB 165.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41255 http://ftp.pdbj.org/pub/emdb/structures/EMD-41255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41255 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41255 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41255_validation.pdf.gz emd_41255_validation.pdf.gz | 966.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41255_full_validation.pdf.gz emd_41255_full_validation.pdf.gz | 966.3 KB | 表示 | |
| XML形式データ |  emd_41255_validation.xml.gz emd_41255_validation.xml.gz | 15 KB | 表示 | |
| CIF形式データ |  emd_41255_validation.cif.gz emd_41255_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41255 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41255 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41255 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8theMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41255.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41255.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.068 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41255_msk_1.map emd_41255_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
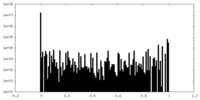
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41255_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
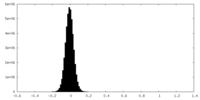
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41255_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
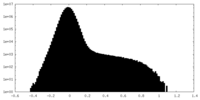
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Heme bound P. aeruginosa PhuR with bound synthetic antibody
| 全体 | 名称: Heme bound P. aeruginosa PhuR with bound synthetic antibody |
|---|---|
| 要素 |
|
-超分子 #1: Heme bound P. aeruginosa PhuR with bound synthetic antibody
| 超分子 | 名称: Heme bound P. aeruginosa PhuR with bound synthetic antibody タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Heme/hemoglobin uptake outer membrane receptor PhuR
| 分子 | 名称: Heme/hemoglobin uptake outer membrane receptor PhuR / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 86.37032 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKYLLPTAAA GLLLLAAQPA MAMGHHHHHH GENLYFQGGN AVPLTPTTIT ATRTEQAVDS VPSTVSVQTR EQLDRQNVNN IKELVRYEP GVSVGGAGQR AGITGYNIRG IDGNRILTQI DGVELPNDFF SGPYAQTHRN YVDPDIVKRV EILRGPASAL Y GSNAIGGA ...文字列: MKYLLPTAAA GLLLLAAQPA MAMGHHHHHH GENLYFQGGN AVPLTPTTIT ATRTEQAVDS VPSTVSVQTR EQLDRQNVNN IKELVRYEP GVSVGGAGQR AGITGYNIRG IDGNRILTQI DGVELPNDFF SGPYAQTHRN YVDPDIVKRV EILRGPASAL Y GSNAIGGA VSYFTLDPSD IIKDGKDVGA RLKAGYESAS HSWLTSATVA GRADDFDGLL HYGYRQGHET ESNGGHGGTG LS RSEANPE DADSYSLLGK LGWNYAEGSR FGLVFEKYKS DVDTDQKSAY GGPYDKGKPA IPPSMLPGGM YQWRKGNDTL TRE RYGLEH HFLLDSQVAD RIQWSLNYQL AKTDQATREF YYPITRKVLR TRDTTYKERL WVFDSQLDKS FAIGETEHLL SYGI NLKHQ KVTGMRSGTG TNLDTGADSP RDALERSSDF PDPTVKTYAL FAQDSISWND WTFTPGLRYD YTRMEPHITD EFLRT MKQS QNTAVDESDK KWHRVSPKFG VTYDFAQHYT WYGQYAQGFR TPTAKALYGR FENLQAGYHI EPNPNLKPEK SQSFET GLR GKFDEGSFGV AVFYNKYRDF IDEDALNTDS TGGNGQTFQS NNIERAVIKG VELKGRLELG AFGAPQGLYT QGSVAYA YG RNKDNGEPIN SVNPLTGVFG LGYDEADGNY GGLLSWTLVK RKDRVDDSTF HTPDGTASQF KTPGFGVLDL SAYYRLSK D LTLNAGLYNL TDKKYWLWDD VRGYDSVGEA SALAPANIDR LSQPGRNFAV NLVWDI UniProtKB: Heme/hemoglobin uptake outer membrane receptor PhuR |
-分子 #2: Synthetic Antibody Heavy Chain
| 分子 | 名称: Synthetic Antibody Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.525445 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NFYYYSIHWV RQAPGKGLEW VASISPYYGS TYYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARSMNWYYSS PYGMSQGMDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NFYYYSIHWV RQAPGKGLEW VASISPYYGS TYYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARSMNWYYSS PYGMSQGMDY WGQGTLVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EPKSCDKTHT |
-分子 #3: Synthetic Antibody Light Chain
| 分子 | 名称: Synthetic Antibody Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.212715 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQGSSSPLTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQGSSSPLTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #5: (2~{R},4~{R},5~{S},6~{S})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[[...
| 分子 | 名称: (2~{R},4~{R},5~{S},6~{S})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[[(2~{R},3~{S},4~{S},5~{R},6~{S})-5-[[(3~{R})-3-dodecanoyloxytetradecanoyl]amino]-6-[[(2~{R},3~{R},4~{R},5~{S},6~{S})-3-oxidanyl- ...名称: (2~{R},4~{R},5~{S},6~{S})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[[(2~{R},3~{S},4~{S},5~{R},6~{S})-5-[[(3~{R})-3-dodecanoyloxytetradecanoyl]amino]-6-[[(2~{R},3~{R},4~{R},5~{S},6~{S})-3-oxidanyl-5-[[(3~{R})-3-oxidanyltetradecanoyl]amino]-4-[(3~{R})-3-oxidanyltetradecanoyl]oxy-6-phosphonooxy-oxan-2-yl]methoxy]-3-phosphonooxy-4-[(3~{S})-3-tetradecanoyloxytetradecanoyl]oxy-oxan-2-yl]methoxy]-4,5-bis(oxidanyl)oxane-2-carboxylic acid タイプ: ligand / ID: 5 / コピー数: 1 / 式: CTQ |
|---|---|
| 分子量 | 理論値: 2.018542 KDa |
| Chemical component information | 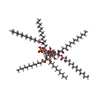 ChemComp-CTQ: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 81 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 5031 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3) / 使用した粒子像数: 292512 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.3) |
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8the: |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)