+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2842 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of TMV at 3.35 A resolution | |||||||||
 マップデータ マップデータ | Cryo-EM structure of TMV at 3.35 A resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | tobacco mosaic virus / direct electron detectors / single particle helical reconstruction / high resolution | |||||||||
| 機能・相同性 | Tobacco mosaic virus-like, coat protein / Tobacco mosaic virus-like, coat protein superfamily / Virus coat protein (TMV like) / helical viral capsid / structural molecule activity / identical protein binding / Capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Tobacco mosaic virus (ウイルス) Tobacco mosaic virus (ウイルス) | |||||||||
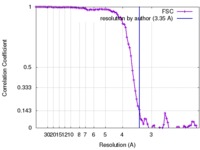
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Fromm SA / Bharat TAM / Jakobi AJ / Hagen WJH / Sachse C | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2015 ジャーナル: J Struct Biol / 年: 2015タイトル: Seeing tobacco mosaic virus through direct electron detectors. 著者: Simon A Fromm / Tanmay A M Bharat / Arjen J Jakobi / Wim J H Hagen / Carsten Sachse /   要旨: With the introduction of direct electron detectors (DED) to the field of electron cryo-microscopy, a wave of atomic-resolution structures has become available. As the new detectors still require ...With the introduction of direct electron detectors (DED) to the field of electron cryo-microscopy, a wave of atomic-resolution structures has become available. As the new detectors still require comparative characterization, we have used tobacco mosaic virus (TMV) as a test specimen to study the quality of 3D image reconstructions from data recorded on the two direct electron detector cameras, K2 Summit and Falcon II. Using DED movie frames, we explored related image-processing aspects and compared the performance of micrograph-based and segment-based motion correction approaches. In addition, we investigated the effect of dose deposition on the atomic-resolution structure of TMV and show that radiation damage affects negative carboxyl chains first in a side-chain specific manner. Finally, using 450,000 asymmetric units and limiting the effects of radiation damage, we determined a high-resolution cryo-EM map at 3.35Å resolution. Here, we provide a comparative case study of highly ordered TMV recorded on different direct electron detectors to establish recording and processing conditions that enable structure determination up to 3.2Å in resolution using cryo-EM. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2842.map.gz emd_2842.map.gz | 23.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2842-v30.xml emd-2842-v30.xml emd-2842.xml emd-2842.xml | 10.8 KB 10.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2842_fsc.xml emd_2842_fsc.xml | 6.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_2842.png emd_2842.png | 297.1 KB | ||
| その他 |  emd_2842_additional_1.map.gz emd_2842_additional_1.map.gz emd_2842_additional_2.map.gz emd_2842_additional_2.map.gz emd_2842_half_map_1.map.gz emd_2842_half_map_1.map.gz emd_2842_half_map_2.map.gz emd_2842_half_map_2.map.gz | 47.8 MB 8.7 MB 25.9 MB 25.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2842 http://ftp.pdbj.org/pub/emdb/structures/EMD-2842 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2842 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2842 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2842_validation.pdf.gz emd_2842_validation.pdf.gz | 360.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2842_full_validation.pdf.gz emd_2842_full_validation.pdf.gz | 359.6 KB | 表示 | |
| XML形式データ |  emd_2842_validation.xml.gz emd_2842_validation.xml.gz | 9.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2842 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2842 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2842 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2842 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  4udvMC  2833C  2834C  2835C  2836C  2837C  2838C  2839C  2840C  2841C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10022 (タイトル: Tobacco Mosaic Virus Falcon II dataset including manually boxed helix coordinates EMPIAR-10022 (タイトル: Tobacco Mosaic Virus Falcon II dataset including manually boxed helix coordinatesData size: 47.7 Data #1: Tobacco Mosaic Virus micrographs [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2842.map.gz / 形式: CCP4 / 大きさ: 31.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2842.map.gz / 形式: CCP4 / 大きさ: 31.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of TMV at 3.35 A resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.062 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
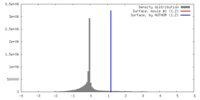
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 2842 additional 1.map
| ファイル | emd_2842_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
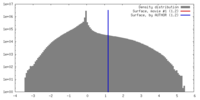
| 密度ヒストグラム |
-添付マップデータ: emd 2842 additional 2.map
| ファイル | emd_2842_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
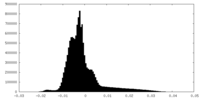
| 密度ヒストグラム |
-添付マップデータ: emd 2842 half map 1.map
| ファイル | emd_2842_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
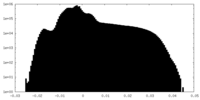
| 密度ヒストグラム |
-添付マップデータ: emd 2842 half map 2.map
| ファイル | emd_2842_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tobacco Mosaic Virus
| 全体 | 名称:   Tobacco Mosaic Virus (ウイルス) Tobacco Mosaic Virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: Tobacco Mosaic Virus
| 超分子 | 名称: Tobacco Mosaic Virus / タイプ: sample / ID: 1000 / 詳細: The sample was purified from infected leaves. / 集合状態: helical / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 39.5 MDa |
-超分子 #1: Tobacco mosaic virus
| 超分子 | 名称: Tobacco mosaic virus / タイプ: virus / ID: 1 / Name.synonym: TMV / NCBI-ID: 12242 / 生物種: Tobacco mosaic virus / Sci species strain: vulgare / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: TMV |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 39.5 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: CP / 直径: 180 Å |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 11.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: 50 mM NaPO4 |
| グリッド | 詳細: Quantifoil 2/2 Cu 200 mesh grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 95 K / 装置: FEI VITROBOT MARK III 手法: Blot for 8 seconds with an offset of -2 mm ~30-45 seconds after sample application |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: CTFTILT |
| 詳細 | Nanoprobe mode, dose rate ~41 e-/px/s on the camera level, fully automated acquisition using FEI EPU software |
| 日付 | 2013年10月16日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 109 / 平均電子線量: 30.7 e/Å2 詳細: each micrograph was recorded as a movie of 7 frames excluding the roll-in frame corresponding to 2 e-/A^2 accumulated dose which was discarded |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 131827 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)