+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Focused reconstruction of influenza A RNP-like particle | |||||||||
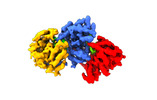
 マップデータ マップデータ | Focused reconstruction map post-processed. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Influenza virus / Nucleocapsid-like / RNA binding protein. / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helical viral capsid / viral penetration into host nucleus / host cell / viral nucleocapsid / ribonucleoprotein complex / symbiont entry into host cell / host cell nucleus / structural molecule activity / RNA binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Influenza A virus (A型インフルエンザウイルス) / synthetic construct (人工物) Influenza A virus (A型インフルエンザウイルス) / synthetic construct (人工物) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 5.3 Å | |||||||||
 データ登録者 データ登録者 | Chenavier F / Estrozi LF / Zarkadas E / Ruigrok RWH / Schoehn G / Ballandras-Colas A / Crepin T | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: Cryo-EM structure of influenza helical nucleocapsid reveals NP-NP and NP-RNA interactions as a model for the genome encapsidation. 著者: Florian Chenavier / Leandro F Estrozi / Jean-Marie Teulon / Eleftherios Zarkadas / Lily-Lorette Freslon / Jean-Luc Pellequer / Rob W H Ruigrok / Guy Schoehn / Allison Ballandras-Colas / Thibaut Crépin /  要旨: Influenza virus genome encapsidation is essential for the formation of a helical viral ribonucleoprotein (vRNP) complex composed of nucleoproteins (NP), the trimeric polymerase, and the viral genome. ...Influenza virus genome encapsidation is essential for the formation of a helical viral ribonucleoprotein (vRNP) complex composed of nucleoproteins (NP), the trimeric polymerase, and the viral genome. Although low-resolution vRNP structures are available, it remains unclear how the viral RNA is encapsidated and how NPs assemble into the helical filament specific of influenza vRNPs. In this study, we established a biological tool, the RNP-like particles assembled from recombinant influenza A virus NP and synthetic RNA, and we present the first subnanometric cryo-electron microscopy structure of the helical NP-RNA complex (8.7 to 5.3 Å). The helical RNP-like structure reveals a parallel double-stranded conformation, allowing the visualization of NP-NP and NP-RNA interactions. The RNA, located at the interface of neighboring NP protomers, interacts with conserved residues previously described as essential for the NP-RNA interaction. The NP undergoes conformational changes to enable RNA binding and helix formation. Together, our findings provide relevant insights for understanding the mechanism for influenza genome encapsidation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18044.map.gz emd_18044.map.gz | 4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18044-v30.xml emd-18044-v30.xml emd-18044.xml emd-18044.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18044_fsc.xml emd_18044_fsc.xml | 3.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18044.png emd_18044.png | 50.7 KB | ||
| マスクデータ |  emd_18044_msk_1.map emd_18044_msk_1.map | 4.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18044.cif.gz emd-18044.cif.gz | 7.1 KB | ||
| その他 |  emd_18044_additional_1.map.gz emd_18044_additional_1.map.gz emd_18044_half_map_1.map.gz emd_18044_half_map_1.map.gz emd_18044_half_map_2.map.gz emd_18044_half_map_2.map.gz | 2.3 MB 4.2 MB 4.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18044 http://ftp.pdbj.org/pub/emdb/structures/EMD-18044 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18044 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18044 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18044_validation.pdf.gz emd_18044_validation.pdf.gz | 690.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18044_full_validation.pdf.gz emd_18044_full_validation.pdf.gz | 690.2 KB | 表示 | |
| XML形式データ |  emd_18044_validation.xml.gz emd_18044_validation.xml.gz | 9.7 KB | 表示 | |
| CIF形式データ |  emd_18044_validation.cif.gz emd_18044_validation.cif.gz | 12.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18044 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18044 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18044 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18044 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pzqMC  8pzpC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18044.map.gz / 形式: CCP4 / 大きさ: 4.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18044.map.gz / 形式: CCP4 / 大きさ: 4.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused reconstruction map post-processed. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.68 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18044_msk_1.map emd_18044_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
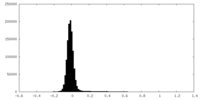
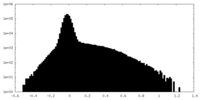
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Focused reconstruction map non post-processed.
| ファイル | emd_18044_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused reconstruction map non post-processed. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1.
| ファイル | emd_18044_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map_1. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2.
| ファイル | emd_18044_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map_2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza A NP-RNA complex
| 全体 | 名称: Influenza A NP-RNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Influenza A NP-RNA complex
| 超分子 | 名称: Influenza A NP-RNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Nucleoprotein
| 超分子 | 名称: Nucleoprotein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
-超分子 #3: RNA (5'P-(UC)6-FAM3')
| 超分子 | 名称: RNA (5'P-(UC)6-FAM3') / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 分子量 | 理論値: 57.525965 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MATKGTKRSY EQMETDGERQ NATEIRASVG KMIDGIGRFY IQMCTELKLS DYEGRLIQNS LTIERMVLSA FDERRNKYLE EHPSAGKDP KKTGGPIYRR VDGKWRRELI LYDKEEIRRI WRQANNGDDA TAGLTHMMIW HSNLNDATYQ RTRALVRTGM D PRMCSLMQ ...文字列: MATKGTKRSY EQMETDGERQ NATEIRASVG KMIDGIGRFY IQMCTELKLS DYEGRLIQNS LTIERMVLSA FDERRNKYLE EHPSAGKDP KKTGGPIYRR VDGKWRRELI LYDKEEIRRI WRQANNGDDA TAGLTHMMIW HSNLNDATYQ RTRALVRTGM D PRMCSLMQ GSTLPRRSGA AGAAVKGVGT MVMELIRMIK RGINDRNFWR GENGRRTRIA YERMCNILKG KFQTAAQRTM VD QVRESRN PGNAEFEDLI FLARSALILR GSVAHKSCLP ACVYGSAVAS GYDFEREGYS LVGIDPFRLL QNSQVYSLIR PNE NPAHKS QLVWMACHSA AFEDLRVSSF IRGTKVVPRG KLSTRGVQIA SNENMETMES STLELRSRYW AIRTRSGGNT NQQR ASSGQ ISIQPTFSVQ RNLPFDRPTI MAAFTGNTEG RTSDMRTEII RLMESARPED VSFQGRGVFE LSDEKATSPI VPSFD MSNE GSYFFGDNAE EYDNLEHHHH HH UniProtKB: Nucleoprotein |
-分子 #2: RNA (5'P-(UC)6-FAM3')
| 分子 | 名称: RNA (5'P-(UC)6-FAM3') / タイプ: rna / ID: 2 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 3.623128 KDa |
| 配列 | 文字列: UCUCUCUCUC UC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.23 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20 mM HEPES 150 mM NaCl 5 mM beta-mercaptoethanol 2 mM methyl-PEG8-NHS | ||||||||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 詳細: 25 mA | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | 20 mM HEPES 150 mM NaCl 5 mM beta-mercaptoethanol Incubation overnight with 0.004 uM RNA (5'P-(UC)6-FAM3') Prior freezing, the sample was incubated 30 min on ice with 2 mM methyl-PEG8-NHS |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 26515 / 平均露光時間: 1.9 sec. / 平均電子線量: 40.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.2 µm / 最小 デフォーカス(補正後): 0.8 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 21-490 / Chain - Source name: PDB / Chain - Initial model type: experimental model / 詳細: Fitting of the NPcore by using ChimeraX |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8pzq: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X