+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of E. coli 70S ribosome | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cryo-ET / subtomogram averaging / ribosome / 70S / E. coli | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 5.3 Å | |||||||||
 データ登録者 データ登録者 | Khusainov I / Romanov N / Goemans C / Turonova B / Zimmerli CE / Welsch S / Langer JD / Typas A / Beck M | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Bactericidal effect of tetracycline in E. coli strain ED1a may be associated with ribosome dysfunction. 著者: Iskander Khusainov / Natalie Romanov / Camille Goemans / Beata Turoňová / Christian E Zimmerli / Sonja Welsch / Julian D Langer / Athanasios Typas / Martin Beck /    要旨: Ribosomes translate the genetic code into proteins. Recent technical advances have facilitated in situ structural analyses of ribosome functional states inside eukaryotic cells and the minimal ...Ribosomes translate the genetic code into proteins. Recent technical advances have facilitated in situ structural analyses of ribosome functional states inside eukaryotic cells and the minimal bacterium Mycoplasma. However, such analyses of Gram-negative bacteria are lacking, despite their ribosomes being major antimicrobial drug targets. Here we compare two E. coli strains, a lab E. coli K-12 and human gut isolate E. coli ED1a, for which tetracycline exhibits bacteriostatic and bactericidal action, respectively. Using our approach for close-to-native E. coli sample preparation, we assess the two strains by cryo-ET and visualize their ribosomes at high resolution in situ. Upon tetracycline treatment, these exhibit virtually identical drug binding sites, yet the conformation distribution of ribosomal complexes differs. While K-12 retains ribosomes in a translation-competent state, tRNAs are lost in the vast majority of ED1a ribosomes. These structural findings together with the proteome-wide abundance and thermal stability assessments indicate that antibiotic responses are complex in cells and can differ between different strains of a single species, thus arguing that all relevant bacterial strains should be analyzed in situ when addressing antibiotic mode of action. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18036.map.gz emd_18036.map.gz | 90.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18036-v30.xml emd-18036-v30.xml emd-18036.xml emd-18036.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18036.png emd_18036.png | 113.9 KB | ||
| Filedesc metadata |  emd-18036.cif.gz emd-18036.cif.gz | 4.4 KB | ||
| その他 |  emd_18036_half_map_1.map.gz emd_18036_half_map_1.map.gz emd_18036_half_map_2.map.gz emd_18036_half_map_2.map.gz | 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18036 http://ftp.pdbj.org/pub/emdb/structures/EMD-18036 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18036 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18036 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18036_validation.pdf.gz emd_18036_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18036_full_validation.pdf.gz emd_18036_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_18036_validation.xml.gz emd_18036_validation.xml.gz | 12.5 KB | 表示 | |
| CIF形式データ |  emd_18036_validation.cif.gz emd_18036_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18036 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18036 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18036 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18036 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18036.map.gz / 形式: CCP4 / 大きさ: 96.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18036.map.gz / 形式: CCP4 / 大きさ: 96.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.697 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_18036_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
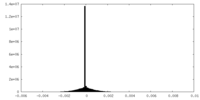
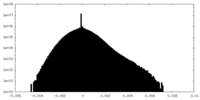
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18036_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
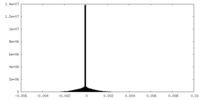
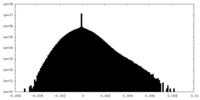
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : In situ structure of E. coli 70S ribosome determined by subtomogr...
| 全体 | 名称: In situ structure of E. coli 70S ribosome determined by subtomogram averaging |
|---|---|
| 要素 |
|
-超分子 #1: In situ structure of E. coli 70S ribosome determined by subtomogr...
| 超分子 | 名称: In situ structure of E. coli 70S ribosome determined by subtomogram averaging タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2.5 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 6.5 / 詳細: LB medium |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 3.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 53000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 5.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: Warp (ver. 1.0.9) / 使用したサブトモグラム数: 75670 |
|---|---|
| 抽出 | トモグラム数: 104 / 使用した粒子像数: 208000 / 参照モデル: 70S average derived from manual picking / 手法: template matching / ソフトウェア - 名称: Warp (ver. 1.0.9) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 3.1.3) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1.3) |
 ムービー
ムービー コントローラー
コントローラー



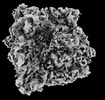









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)