+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
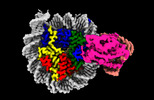
| タイトル | Cryo-EM structure of ALC1 bound to an asymmetric, site-specifically PARylated nucleosome | ||||||||||||
 マップデータ マップデータ | Map from non-uniform refinement of 212 256 particles in cryoSPARC. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | ALC1 / CHD1L / nucleosome / PARylation / ADP-ribosylation / post-translational modification / chromatin / DNA BINDING PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報poly-ADP-D-ribose modification-dependent protein binding / ATP-dependent chromatin remodeler activity / site of DNA damage / nucleosome binding / DNA helicase activity / histone reader activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / Dual Incision in GG-NER / Formation of Incision Complex in GG-NER / structural constituent of chromatin ...poly-ADP-D-ribose modification-dependent protein binding / ATP-dependent chromatin remodeler activity / site of DNA damage / nucleosome binding / DNA helicase activity / histone reader activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / Dual Incision in GG-NER / Formation of Incision Complex in GG-NER / structural constituent of chromatin / nucleosome / nucleosome assembly / site of double-strand break / chromatin remodeling / protein heterodimerization activity / DNA repair / nucleotide binding / DNA damage response / ATP hydrolysis activity / DNA binding / nucleoplasm / ATP binding / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) / | ||||||||||||
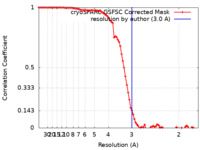
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||
 データ登録者 データ登録者 | Bacic L / Gaullier G / Deindl S | ||||||||||||
| 資金援助 | European Union,  スウェーデン, 3件 スウェーデン, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Asymmetric nucleosome PARylation at DNA breaks mediates directional nucleosome sliding by ALC1. 著者: Luka Bacic / Guillaume Gaullier / Jugal Mohapatra / Guanzhong Mao / Klaus Brackmann / Mikhail Panfilov / Glen Liszczak / Anton Sabantsev / Sebastian Deindl /   要旨: The chromatin remodeler ALC1 is activated by DNA damage-induced poly(ADP-ribose) deposited by PARP1/PARP2 and their co-factor HPF1. ALC1 has emerged as a cancer drug target, but how it is recruited ...The chromatin remodeler ALC1 is activated by DNA damage-induced poly(ADP-ribose) deposited by PARP1/PARP2 and their co-factor HPF1. ALC1 has emerged as a cancer drug target, but how it is recruited to ADP-ribosylated nucleosomes to affect their positioning near DNA breaks is unknown. Here we find that PARP1/HPF1 preferentially initiates ADP-ribosylation on the histone H2B tail closest to the DNA break. To dissect the consequences of such asymmetry, we generate nucleosomes with a defined ADP-ribosylated H2B tail on one side only. The cryo-electron microscopy structure of ALC1 bound to such an asymmetric nucleosome indicates preferential engagement on one side. Using single-molecule FRET, we demonstrate that this asymmetric recruitment gives rise to directed sliding away from the DNA linker closest to the ADP-ribosylation site. Our data suggest a mechanism by which ALC1 slides nucleosomes away from a DNA break to render it more accessible to repair factors. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15777.map.gz emd_15777.map.gz | 32 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15777-v30.xml emd-15777-v30.xml emd-15777.xml emd-15777.xml | 28 KB 28 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15777_fsc.xml emd_15777_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15777.png emd_15777.png | 81.7 KB | ||
| マスクデータ |  emd_15777_msk_1.map emd_15777_msk_1.map emd_15777_msk_2.map emd_15777_msk_2.map | 64 MB 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15777.cif.gz emd-15777.cif.gz | 7.7 KB | ||
| その他 |  emd_15777_additional_1.map.gz emd_15777_additional_1.map.gz emd_15777_half_map_1.map.gz emd_15777_half_map_1.map.gz emd_15777_half_map_2.map.gz emd_15777_half_map_2.map.gz | 54.5 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15777 http://ftp.pdbj.org/pub/emdb/structures/EMD-15777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15777 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15777_validation.pdf.gz emd_15777_validation.pdf.gz | 921.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15777_full_validation.pdf.gz emd_15777_full_validation.pdf.gz | 921 KB | 表示 | |
| XML形式データ |  emd_15777_validation.xml.gz emd_15777_validation.xml.gz | 16.6 KB | 表示 | |
| CIF形式データ |  emd_15777_validation.cif.gz emd_15777_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15777 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15777 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15777 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15777 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8b0aMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15777.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15777.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from non-uniform refinement of 212 256 particles in cryoSPARC. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8617 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15777_msk_1.map emd_15777_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
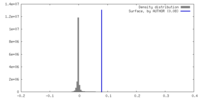
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_15777_msk_2.map emd_15777_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
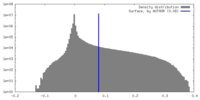
| 密度ヒストグラム |
-追加マップ: Main map denoised with deepEMhancer, using the "highRes"...
| ファイル | emd_15777_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map denoised with deepEMhancer, using the "highRes" denoising model. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
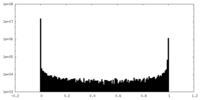
| 密度ヒストグラム |
-ハーフマップ: Half-map A from non-uniform refinement in cryoSPARC.
| ファイル | emd_15777_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map A from non-uniform refinement in cryoSPARC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
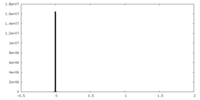
| 密度ヒストグラム |
-ハーフマップ: Half-map B from non-uniform refinement in cryoSPARC.
| ファイル | emd_15777_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map B from non-uniform refinement in cryoSPARC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex between ALC1 and a site-specifically PARylated nucleosome
| 全体 | 名称: Complex between ALC1 and a site-specifically PARylated nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: Complex between ALC1 and a site-specifically PARylated nucleosome
| 超分子 | 名称: Complex between ALC1 and a site-specifically PARylated nucleosome タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Site-specific ADP-ribosylation was introduced as described in https://doi.org/10.7554/elife.71502 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 304.57 KDa |
-分子 #1: Chromodomain-helicase-DNA-binding protein 1-like
| 分子 | 名称: Chromodomain-helicase-DNA-binding protein 1-like / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 98.906766 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFLLRLHTEG RAEAARVQEQ DLRQWGLTGI HLRSYQLEGV NWLAQRFHCQ NGCILGDEMG LGKTCQTIAL FIYLAGRLND EGPFLILCP LSVLSNWKEE MQRFAPGLSC VTYAGDKEER ACLQQDLKQE SRFHVLLTTY EICLKDASFL KSFPWSVLVV D EAHRLKNQ ...文字列: MFLLRLHTEG RAEAARVQEQ DLRQWGLTGI HLRSYQLEGV NWLAQRFHCQ NGCILGDEMG LGKTCQTIAL FIYLAGRLND EGPFLILCP LSVLSNWKEE MQRFAPGLSC VTYAGDKEER ACLQQDLKQE SRFHVLLTTY EICLKDASFL KSFPWSVLVV D EAHRLKNQ SSLLHKTLSE FSVVFSLLLT GTPIQNSLQE LYSLLSFVEP DLFSKEEVGD FIQRYQDIEK ESESASELHK LL QPFLLRR VKAEVATELP KKTEVVIYHG MSALQKKYYK AILMKDLDAF ENETAKKVKL QNILSQLRKC VDHPYLFDGV EPE PFEVGD HLTEASGKLH LLDKLLAFLY SGGHRVLLFS QMTQMLDILQ DYMDYRGYSY ERVDGSVRGE ERHLAIKNFG QQPI FVFLL STRAGGVGMN LTAADTVIFV DSDFNPQNDL QAAARAHRIG QNKSVKVIRL IGRDTVEEIV YRKAASKLQL TNMII EGGH FTLGAQKPAA DADLQLSEIL KFGLDKLLAS EGSTMDEIDL ESILGETKDG QWVSDALPAA EGGSRDQEEG KNHMYL FEG KDYSKEPSKE DRKSFEQLVN LQKTLLEKAS QEGRSLRNKG SVLIPGLVEG STKRKRVLSP EELEDRQKKR QEAAAKR RR LIEEKKRQKE EAEHKKKMAW WESNNYQSFC LPSEESEPED LENGEESSAE LDYQDPDATS LKYVSGDVTH PQAGAEDA L IVHCVDDSGH WGRGGLFTAL EKRSAEPRKI YELAGKMKDL SLGGVLLFPV DDKESRNKGQ DLLALIVAQH RDRSNVLSG IKMAALEEGL KKIFLAAKKK KASVHLPRIG HATKGFNWYG TERLIRKHLA ARGIPTYIYY FPRSKAHHHH HH UniProtKB: Chromodomain-helicase-DNA-binding protein 1-like |
-分子 #2: Histone H3
| 分子 | 名称: Histone H3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.403062 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARTKQTARK STGGKAPRKQ LATKAARKSA PATGGVKKPH RYRPGTVALR EIRRYQKSTE LLIRKLPFQR LVREIAQDFK TDLRFQSSA VMALQEASEA YLVALFEDTN LAAIHAKRVT IMPKDIQLAR RIRGERA UniProtKB: Histone H3 |
-分子 #3: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.394426 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKGGKG LGKGGAKRHR KVLRDNIQGI TKPAIRRLAR RGGVKRISGL IYEETRGVLK VFLENVIRDA VTYTEHAKRK TVTAMDVVY ALKRQGRTLY GFGG UniProtKB: Histone H4 |
-分子 #4: Histone H2A type 1
| 分子 | 名称: Histone H2A type 1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 14.109436 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGRGKQGGK TRAKAKTRSS RAGLQFPVGR VHRLLRKGNY AERVGAGAPV YLAAVLEYLT AEILELAGNA ARDNKKTRII PRHLQLAVR NDEELNKLLG RVTIAQGGVL PNIQSVLLPK KTESSKSAKS K UniProtKB: Histone H2A type 1 |
-分子 #5: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.655948 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKSAPAPKK GSKKAVTKTQ KKDGKKRRKT RKESYAIYVY KVLKQVHPDT GISSKAMSIM NSFVNDVFER IAGEASRLAH YNKRSTITS REIQTAVRLL LPGELAKHAV SEGTKAVTKY TSAK UniProtKB: Histone H2B 1.1 |
-分子 #6: DNA (149-MER) Widom 601 sequence
| 分子 | 名称: DNA (149-MER) Widom 601 sequence / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 49.175336 KDa |
| 配列 | 文字列: (DT)(DC)(DT)(DA)(DG)(DG)(DT)(DG)(DA)(DC) (DC)(DA)(DT)(DC)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG) (DA)(DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA) (DT)(DT)(DG)(DG)(DT) ...文字列: (DT)(DC)(DT)(DA)(DG)(DG)(DT)(DG)(DA)(DC) (DC)(DA)(DT)(DC)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG) (DA)(DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT) (DA)(DG)(DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT) (DA)(DG) (DC)(DA)(DC)(DC)(DG)(DC)(DT) (DT)(DA)(DA)(DA)(DC)(DG)(DC)(DA)(DC)(DG) (DT)(DA)(DC) (DG)(DC)(DG)(DC)(DT)(DG) (DT)(DC)(DC)(DC)(DC)(DC)(DG)(DC)(DG)(DT) (DT)(DT)(DT)(DA) (DA)(DC)(DC)(DG)(DC) (DC)(DA)(DA)(DG)(DG)(DG)(DG)(DA)(DT)(DT) (DA)(DC)(DT)(DC)(DC) (DC)(DT)(DA)(DG) (DT)(DC)(DT)(DC)(DC)(DA)(DG)(DG)(DC)(DA) (DC)(DG)(DT)(DG)(DT)(DC) (DA)(DG)(DA) (DT)(DA)(DT)(DA)(DT)(DA)(DC)(DA)(DT)(DC) (DG)(DA)(DT)(DA)(DG)(DG)(DC) |
-分子 #7: DNA (149-MER) Widom 601 sequence
| 分子 | 名称: DNA (149-MER) Widom 601 sequence / タイプ: dna / ID: 7 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 49.606586 KDa |
| 配列 | 文字列: (DG)(DC)(DC)(DT)(DA)(DT)(DC)(DG)(DA)(DT) (DG)(DT)(DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT) (DG)(DA)(DC)(DA)(DC)(DG)(DT)(DG)(DC) (DC)(DT)(DG)(DG)(DA)(DG)(DA)(DC)(DT)(DA) (DG) (DG)(DG)(DA)(DG)(DT) ...文字列: (DG)(DC)(DC)(DT)(DA)(DT)(DC)(DG)(DA)(DT) (DG)(DT)(DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT) (DG)(DA)(DC)(DA)(DC)(DG)(DT)(DG)(DC) (DC)(DT)(DG)(DG)(DA)(DG)(DA)(DC)(DT)(DA) (DG) (DG)(DG)(DA)(DG)(DT)(DA)(DA)(DT) (DC)(DC)(DC)(DC)(DT)(DT)(DG)(DG)(DC)(DG) (DG)(DT) (DT)(DA)(DA)(DA)(DA)(DC)(DG) (DC)(DG)(DG)(DG)(DG)(DG)(DA)(DC)(DA)(DG) (DC)(DG)(DC) (DG)(DT)(DA)(DC)(DG)(DT) (DG)(DC)(DG)(DT)(DT)(DT)(DA)(DA)(DG)(DC) (DG)(DG)(DT)(DG) (DC)(DT)(DA)(DG)(DA) (DG)(DC)(DT)(DG)(DT)(DC)(DT)(DA)(DC)(DG) (DA)(DC)(DC)(DA)(DA) (DT)(DT)(DG)(DA) (DG)(DC)(DG)(DG)(DC)(DC)(DT)(DC)(DG)(DG) (DC)(DA)(DC)(DC)(DG)(DG) (DG)(DA)(DT) (DT)(DC)(DT)(DG)(DA)(DT)(DG)(DG)(DT)(DC) (DA)(DC)(DC)(DT)(DA)(DG)(DA) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 実像数: 22155 / 平均電子線量: 37.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)