+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12114 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast C complex spliceosome, overall map after focused classification on Prp18 | |||||||||
 マップデータ マップデータ | Yeast C complex spliceosome, overall map after focused classification on Prp18 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Wilkinson ME / Fica SM / Galej WP / Nagai K | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Structural basis for conformational equilibrium of the catalytic spliceosome. 著者: Max E Wilkinson / Sebastian M Fica / Wojciech P Galej / Kiyoshi Nagai /  要旨: The ATPase Prp16 governs equilibrium between the branching (B/C) and exon ligation (C/P) conformations of the spliceosome. Here, we present the electron cryomicroscopy reconstruction of the ...The ATPase Prp16 governs equilibrium between the branching (B/C) and exon ligation (C/P) conformations of the spliceosome. Here, we present the electron cryomicroscopy reconstruction of the Saccharomyces cerevisiae C-complex spliceosome at 2.8 Å resolution and identify a novel C-complex intermediate (C) that elucidates the molecular basis for this equilibrium. The exon-ligation factors Prp18 and Slu7 bind to C before ATP hydrolysis by Prp16 can destabilize the branching conformation. Biochemical assays suggest that these pre-bound factors prime the C complex for conversion to C by Prp16. A complete model of the Prp19 complex (NTC) reveals how the branching factors Yju2 and Isy1 are recruited by the NTC before branching. Prp16 remodels Yju2 binding after branching, allowing Yju2 to remain tethered to the NTC in the C complex to promote exon ligation. Our results explain how Prp16 action modulates the dynamic binding of step-specific factors to alternatively stabilize the C or C conformation and establish equilibrium of the catalytic spliceosome. #1:  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structural basis for conformational equilibrium of the catalytic spliceosome 著者: Wilkinson ME / Fica SM / Galej WP / Nagai K | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12114.map.gz emd_12114.map.gz | 223.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12114-v30.xml emd-12114-v30.xml emd-12114.xml emd-12114.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
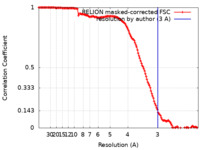
| FSC (解像度算出) |  emd_12114_fsc.xml emd_12114_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12114.png emd_12114.png | 172.1 KB | ||
| その他 |  emd_12114_half_map_1.map.gz emd_12114_half_map_1.map.gz emd_12114_half_map_2.map.gz emd_12114_half_map_2.map.gz | 181.4 MB 181.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12114 http://ftp.pdbj.org/pub/emdb/structures/EMD-12114 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12114 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12114 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12114_validation.pdf.gz emd_12114_validation.pdf.gz | 416.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12114_full_validation.pdf.gz emd_12114_full_validation.pdf.gz | 415.7 KB | 表示 | |
| XML形式データ |  emd_12114_validation.xml.gz emd_12114_validation.xml.gz | 20 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12114 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12114 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12114 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12114 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7b9vC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10687 (タイトル: Yeast C, Ci, C*, and P complex spliceosomes / Data size: 8.9 TB EMPIAR-10687 (タイトル: Yeast C, Ci, C*, and P complex spliceosomes / Data size: 8.9 TBData #1: Unaligned movies of C-complex spliceosome with 3' splice site AG to AC mutation (Dataset 1) [micrographs - multiframe] Data #2: Unaligned movies of C and C*-complex spliceosomes with 3' splice site AG to AdG mutation (Dataset 2) [micrographs - multiframe] Data #3: Unaligned movies of C and C*-complex spliceosomes with 3' splice site AG to AdG mutation (Dataset 3) [micrographs - multiframe] Data #4: Aligned movies of C-complex spliceosomes with cold-sensitive prp16-302 mutation, purified with Cwc25 (Dataset 4) [micrographs - multiframe] Data #5: Unaligned movies of C-complex spliceosomes with cold-sensitive prp16-302 mutation, purified with Cwc25 and incubated with ATP and Mg (Dataset 5) [micrographs - multiframe] Data #6: Unaligned movies of C, C*, and P-complex spliceosomes with dominant-negative Prp22 mutation K512A, purified with Slu7 (Dataset 6) [micrographs - multiframe] Data #7: Unaligned movies of P-complex spliceosomes with dominant-negative Prp22 mutation K512A, treated with anti-3'exon RNaseH oligo, purified in presence of Mg (Dataset 9) [micrographs - single frame] Data #8: Selected C-complex particles after polishing [picked particles - single frame - processed] Data #9: Selected P-complex particles after polishing [picked particles - single frame - processed] Data #10: Various signal subtractions for C- and P-complex spliceosomes [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12114.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12114.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Yeast C complex spliceosome, overall map after focused classification on Prp18 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.145 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Yeast C complex spliceosome, overall map after focused...
| ファイル | emd_12114_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Yeast C complex spliceosome, overall map after focused classification on Prp18, half-map 1 | ||||||||||||
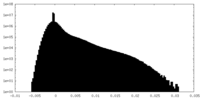
| 投影像・断面図 |
| ||||||||||||
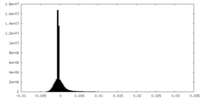
| 密度ヒストグラム |
-ハーフマップ: Yeast C complex spliceosome, overall map after focused...
| ファイル | emd_12114_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Yeast C complex spliceosome, overall map after focused classification on Prp18, half-map 2 | ||||||||||||
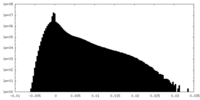
| 投影像・断面図 |
| ||||||||||||
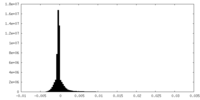
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Yeast C complex spliceosome with Prp18/Slu7 bound
| 全体 | 名称: Yeast C complex spliceosome with Prp18/Slu7 bound |
|---|---|
| 要素 |
|
-超分子 #1: Yeast C complex spliceosome with Prp18/Slu7 bound
| 超分子 | 名称: Yeast C complex spliceosome with Prp18/Slu7 bound / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#40 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 2 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 9 / 実像数: 24115 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X