+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9873 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of PaaZ, a bifunctional enzyme | |||||||||||||||
 マップデータ マップデータ | PaaZ is a bifucntional enzyme that performs hydratase and dehydrogenase reactions. This structure is the native protein. This map is final sharpened map used for interpretation. | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | substrate channeling / bi-functional enzyme / hydrolase / dehydrogenase | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報3-oxo-5,6-dehydrosuberyl-CoA semialdehyde dehydrogenase / oxepin-CoA hydrolase / hydrolase activity, acting on acid carbon-carbon bonds, in ketonic substances / ether hydrolase activity / oxidoreductase activity, acting on CH or CH2 groups, NAD or NADP as acceptor / phenylacetate catabolic process / oxidoreductase activity, acting on the aldehyde or oxo group of donors, NAD or NADP as acceptor / enoyl-CoA hydratase activity / identical protein binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  | |||||||||||||||
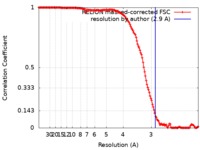
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||
 データ登録者 データ登録者 | Gakher L / Vinothkumar KR / Katagihallimath N / Sowdhamini R / Sathyanarayanan N / Cannone G | |||||||||||||||
| 資金援助 |  インド, インド,  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Molecular basis for metabolite channeling in a ring opening enzyme of the phenylacetate degradation pathway. 著者: Nitish Sathyanarayanan / Giuseppe Cannone / Lokesh Gakhar / Nainesh Katagihallimath / Ramanathan Sowdhamini / Subramanian Ramaswamy / Kutti R Vinothkumar /    要旨: Substrate channeling is a mechanism for the internal transfer of hydrophobic, unstable or toxic intermediates from the active site of one enzyme to another. Such transfer has previously been ...Substrate channeling is a mechanism for the internal transfer of hydrophobic, unstable or toxic intermediates from the active site of one enzyme to another. Such transfer has previously been described to be mediated by a hydrophobic tunnel, the use of electrostatic highways or pivoting and by conformational changes. The enzyme PaaZ is used by many bacteria to degrade environmental pollutants. PaaZ is a bifunctional enzyme that catalyzes the ring opening of oxepin-CoA and converts it to 3-oxo-5,6-dehydrosuberyl-CoA. Here we report the structures of PaaZ determined by electron cryomicroscopy with and without bound ligands. The structures reveal that three domain-swapped dimers of the enzyme form a trilobed structure. A combination of small-angle X-ray scattering (SAXS), computational studies, mutagenesis and microbial growth experiments suggests that the key intermediate is transferred from one active site to the other by a mechanism of electrostatic pivoting of the CoA moiety, mediated by a set of conserved positively charged residues. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9873.map.gz emd_9873.map.gz | 250 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9873-v30.xml emd-9873-v30.xml emd-9873.xml emd-9873.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9873_fsc.xml emd_9873_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9873.png emd_9873.png | 32.7 KB | ||
| Filedesc metadata |  emd-9873.cif.gz emd-9873.cif.gz | 7 KB | ||
| その他 |  emd_9873_additional_1.map.gz emd_9873_additional_1.map.gz emd_9873_additional_2.map.gz emd_9873_additional_2.map.gz | 212.6 MB 212.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9873 http://ftp.pdbj.org/pub/emdb/structures/EMD-9873 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9873 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9873 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9873_validation.pdf.gz emd_9873_validation.pdf.gz | 628.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9873_full_validation.pdf.gz emd_9873_full_validation.pdf.gz | 627.7 KB | 表示 | |
| XML形式データ |  emd_9873_validation.xml.gz emd_9873_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  emd_9873_validation.cif.gz emd_9873_validation.cif.gz | 19.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9873 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9873 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9873 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9873 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9873.map.gz / 形式: CCP4 / 大きさ: 266.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9873.map.gz / 形式: CCP4 / 大きさ: 266.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PaaZ is a bifucntional enzyme that performs hydratase and dehydrogenase reactions. This structure is the native protein. This map is final sharpened map used for interpretation. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
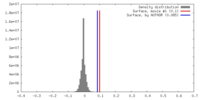
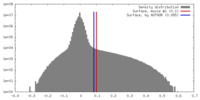
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: This map is one of the half-maps after refinement.
| ファイル | emd_9873_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
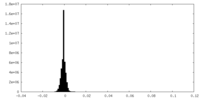
| 注釈 | This map is one of the half-maps after refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: This map is one of the half-maps after refinement.
| ファイル | emd_9873_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
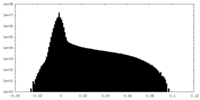
| 注釈 | This map is one of the half-maps after refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PaaZ
| 全体 | 名称: PaaZ |
|---|---|
| 要素 |
|
-超分子 #1: PaaZ
| 超分子 | 名称: PaaZ / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: PaaZ is a bifunctional enzyme that has hydrolase and dehydrogenase activity. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 440 KDa |
-分子 #1: Bifunctional protein PaaZ
| 分子 | 名称: Bifunctional protein PaaZ / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: oxepin-CoA hydrolase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 73.969391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHQQ LASFLSGTWQ SGRGRSRLIH HAISGEALWE VTSEGLDMAA ARQFAIEKGA PALRAMTFIE RAAMLKAVAK HLLSEKERF YALSAQTGAT RADSWVDIEG GIGTLFTYAS LGSRELPDDT LWPEDELIPL SKEGGFAARH LLTSKSGVAV H INAFNFPC ...文字列: MGHHHHHHQQ LASFLSGTWQ SGRGRSRLIH HAISGEALWE VTSEGLDMAA ARQFAIEKGA PALRAMTFIE RAAMLKAVAK HLLSEKERF YALSAQTGAT RADSWVDIEG GIGTLFTYAS LGSRELPDDT LWPEDELIPL SKEGGFAARH LLTSKSGVAV H INAFNFPC WGMLEKLAPT WLGGMPAIIK PATATAQLTQ AMVKSIVDSG LVPEGAISLI CGSAGDLLDH LDSQDVVTFT GS AATGQML RVQPNIVAKS IPFTMEADSL NCCVLGEDVT PDQPEFALFI REVVREMTTK AGQKCTAIRR IIVPQALVNA VSD ALVARL QKVVVGDPAQ EGVKMGALVN AEQRADVQEK VNILLAAGCE IRLGGQADLS AAGAFFPPTL LYCPQPDETP AVHA TEAFG PVATLMPAQN QRHALQLACA GGGSLAGTLV TADPQIARQF IADAARTHGR IQILNEESAK ESTGHGSPLP QLVHG GPGR AGGGEELGGL RAVKHYMQRT AVQGSPTMLA AISKQWVRGA KVEEDRIHPF RKYFEELQPG DSLLTPRRTM TEADIV NFA CLSGDHFYAH MDKIAAAESI FGERVVHGYF VLSAAAGLFV DAGVGPVIAN YGLESLRFIE PVKPGDTIQV RLTCKRK TL KKQRSAEEKP TGVVEWAVEV FNQHQTPVAL YSILTLVARQ HGDFVD UniProtKB: Bifunctional protein PaaZ |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.015 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 / 構成要素:
詳細: Protein was purified and kept in 25mM Hepes buffer and 50 mM NaCl | ||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 300 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa 詳細: The Ultrafoil grids was glow discharged in air for 5 minutes. Subsequently, 0.2 mg/ml of graphene oxide solution was placed and washed twice with water. The grids were air dried and used directly for freezing. | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV 詳細: blotting force 10, blotting time 4 sec, waiting time 15 sec, drying time 0, blotting times 1.. | ||||||
| 詳細 | The peak fraction from gel filtration was used for grid preparation. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 詳細 | Data was collected with EPU software |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 552 / 平均露光時間: 60.0 sec. / 平均電子線量: 27.0 e/Å2 詳細: The 60 second exposure was saved into 75 frames with each frame ~0.36 e-. The frames were then grouped into 3 for alignment and summed images were used for data processing |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 132075 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 2.2 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Real space refinement with secondary structure enabled, minimization and adp |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 44.4 |
| 得られたモデル |  PDB-6jql: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)