+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9233 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | T20S proteasome | |||||||||
 マップデータ マップデータ | T20S sharpened masked map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) | |||||||||
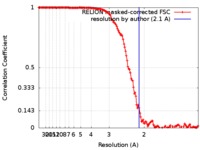
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.1 Å | |||||||||
 データ登録者 データ登録者 | Eng ET / Kopylov M / Jordan KJ / Rice WJ / Carragher BO / Potter CS | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2019 ジャーナル: J Struct Biol / 年: 2019タイトル: Reducing cryoEM file storage using lossy image formats. 著者: Edward T Eng / Mykhailo Kopylov / Carl J Negro / Sarkis Dallaykan / William J Rice / Kelsey D Jordan / Kotaro Kelley / Bridget Carragher / Clinton S Potter /  要旨: Recent advances in instrumentation and software for cryoEM have increased the applicability and utility of this method. High levels of automation and faster data acquisition rates require hard ...Recent advances in instrumentation and software for cryoEM have increased the applicability and utility of this method. High levels of automation and faster data acquisition rates require hard decisions to be made regarding data retention. Here we investigate the efficacy of data compression applied to aligned summed movie files. Surprisingly, these images can be compressed using a standard lossy method that reduces file storage by 90-95% and yet can still be processed to provide sub-2 Å reconstructed maps. We do not advocate this as an archival method, but it may provide a useful means for retaining images as an historical record, especially at large facilities. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9233.map.gz emd_9233.map.gz | 30.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9233-v30.xml emd-9233-v30.xml emd-9233.xml emd-9233.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9233_fsc.xml emd_9233_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9233.png emd_9233.png | 217.7 KB | ||
| マスクデータ |  emd_9233_msk_1.map emd_9233_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_9233_additional.map.gz emd_9233_additional.map.gz emd_9233_half_map_1.map.gz emd_9233_half_map_1.map.gz emd_9233_half_map_2.map.gz emd_9233_half_map_2.map.gz | 192.9 MB 194 MB 194.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9233 http://ftp.pdbj.org/pub/emdb/structures/EMD-9233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9233 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9233_validation.pdf.gz emd_9233_validation.pdf.gz | 78.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9233_full_validation.pdf.gz emd_9233_full_validation.pdf.gz | 77.3 KB | 表示 | |
| XML形式データ |  emd_9233_validation.xml.gz emd_9233_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9233 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9233 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9233 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9233 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9203C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10218 (タイトル: Thermoplasma acidophilum 20S / Data size: 1.5 TB EMPIAR-10218 (タイトル: Thermoplasma acidophilum 20S / Data size: 1.5 TBData #1: unaligned multi-frame micrographs of T20S (18mar31a) [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9233.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9233.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T20S sharpened masked map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.6616 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
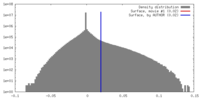
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9233_msk_1.map emd_9233_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
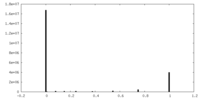
| 密度ヒストグラム |
-追加マップ: T20S unsharpened map
| ファイル | emd_9233_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T20S unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
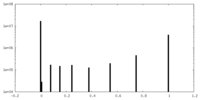
| 密度ヒストグラム |
-ハーフマップ: T20S even half map
| ファイル | emd_9233_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T20S even half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
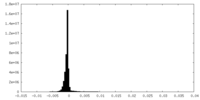
| 密度ヒストグラム |
-ハーフマップ: T20S odd half map
| ファイル | emd_9233_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T20S odd half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 20S proteasome
| 全体 | 名称: 20S proteasome |
|---|---|
| 要素 |
|
-超分子 #1: 20S proteasome
| 超分子 | 名称: 20S proteasome / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 700 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.21 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 20 mM Tris, 150 mM NaCl | |||||||||
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 75 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 詳細: Blotted for 2.5 seconds after a 30 second wait time.. | |||||||||
| 詳細 | 1.2/1.3 C-Flat grid, plasma cleaned |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 詳細 | Leginon |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 1173 / 平均露光時間: 6.259 sec. / 平均電子線量: 62.59 e/Å2 詳細: Images were collected in movie mode at 5 frames per second. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.02 µm / 最小 デフォーカス(補正後): 0.2 µm / 倍率(補正後): 75574 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 37000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)