+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9111 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis for cholesterol transport-like activity of the Hedgehog receptor Patched | ||||||||||||||||||
 マップデータ マップデータ | PTCH1 monomer | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Ligand-receptor interactions / Activation of SMO / neural plate axis specification / cell differentiation involved in kidney development / hedgehog receptor activity / response to chlorate / cell proliferation involved in metanephros development / smoothened binding / neural tube formation / hedgehog family protein binding ...Ligand-receptor interactions / Activation of SMO / neural plate axis specification / cell differentiation involved in kidney development / hedgehog receptor activity / response to chlorate / cell proliferation involved in metanephros development / smoothened binding / neural tube formation / hedgehog family protein binding / hindlimb morphogenesis / Hedgehog 'on' state / epidermal cell fate specification / spinal cord motor neuron differentiation / prostate gland development / Hedgehog 'off' state / somite development / patched binding / negative regulation of cell division / cellular response to cholesterol / dorsal/ventral neural tube patterning / smooth muscle tissue development / pattern specification process / pharyngeal system development / mammary gland epithelial cell differentiation / mammary gland duct morphogenesis / mammary gland development / cell fate determination / commissural neuron axon guidance / metanephric collecting duct development / dorsal/ventral pattern formation / regulation of growth / embryonic limb morphogenesis / negative regulation of multicellular organism growth / branching involved in ureteric bud morphogenesis / cholesterol binding / positive regulation of epidermal cell differentiation / dendritic growth cone / keratinocyte proliferation / spermatid development / positive regulation of cholesterol efflux / epidermis development / negative regulation of keratinocyte proliferation / negative regulation of osteoblast differentiation / embryonic organ development / axonal growth cone / negative regulation of smoothened signaling pathway / heart morphogenesis / response to mechanical stimulus / response to retinoic acid / negative regulation of stem cell proliferation / regulation of mitotic cell cycle / cyclin binding / epithelial cell proliferation / stem cell proliferation / caveola / neural tube closure / protein localization to plasma membrane / liver regeneration / animal organ morphogenesis / brain development / protein processing / cilium / negative regulation of epithelial cell proliferation / regulation of protein localization / apical part of cell / response to estradiol / glucose homeostasis / heparin binding / regulation of cell population proliferation / midbody / in utero embryonic development / response to xenobiotic stimulus / negative regulation of cell population proliferation / negative regulation of DNA-templated transcription / protein-containing complex binding / positive regulation of DNA-templated transcription / perinuclear region of cytoplasm / Golgi apparatus / negative regulation of transcription by RNA polymerase II / signal transduction / zinc ion binding / extracellular region / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||||||||
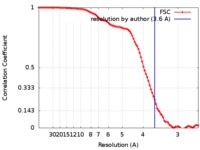
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zhang Y / Bulkley D / Xin Y / Roberts KJ / Asarnow DE / Sharma A / Myers BR / Cho W / Cheng Y / Beachy PA | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2018 ジャーナル: Cell / 年: 2018タイトル: Structural Basis for Cholesterol Transport-like Activity of the Hedgehog Receptor Patched. 著者: Yunxiao Zhang / David P Bulkley / Yao Xin / Kelsey J Roberts / Daniel E Asarnow / Ashutosh Sharma / Benjamin R Myers / Wonhwa Cho / Yifan Cheng / Philip A Beachy /  要旨: Hedgehog protein signals mediate tissue patterning and maintenance by binding to and inactivating their common receptor Patched, a 12-transmembrane protein that otherwise would suppress the activity ...Hedgehog protein signals mediate tissue patterning and maintenance by binding to and inactivating their common receptor Patched, a 12-transmembrane protein that otherwise would suppress the activity of the 7-transmembrane protein Smoothened. Loss of Patched function, the most common cause of basal cell carcinoma, permits unregulated activation of Smoothened and of the Hedgehog pathway. A cryo-EM structure of the Patched protein reveals striking transmembrane domain similarities to prokaryotic RND transporters. A central hydrophobic conduit with cholesterol-like contents courses through the extracellular domain and resembles that used by other RND proteins to transport substrates, suggesting Patched activity in cholesterol transport. Cholesterol activity in the inner leaflet of the plasma membrane is reduced by PTCH1 expression but rapidly restored by Hedgehog stimulation, suggesting that PTCH1 regulates Smoothened by controlling cholesterol availability. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9111.map.gz emd_9111.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9111-v30.xml emd-9111-v30.xml emd-9111.xml emd-9111.xml | 24.1 KB 24.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9111_fsc.xml emd_9111_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9111.png emd_9111.png | 50 KB | ||
| その他 |  emd_9111_additional_1.map.gz emd_9111_additional_1.map.gz emd_9111_additional_2.map.gz emd_9111_additional_2.map.gz emd_9111_half_map_1.map.gz emd_9111_half_map_1.map.gz emd_9111_half_map_2.map.gz emd_9111_half_map_2.map.gz | 2.8 MB 2.9 MB 55.5 MB 55.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9111 http://ftp.pdbj.org/pub/emdb/structures/EMD-9111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9111 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9111 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9111_validation.pdf.gz emd_9111_validation.pdf.gz | 449.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9111_full_validation.pdf.gz emd_9111_full_validation.pdf.gz | 448.7 KB | 表示 | |
| XML形式データ |  emd_9111_validation.xml.gz emd_9111_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_9111_validation.cif.gz emd_9111_validation.cif.gz | 23.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9111 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9111 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9111 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9111.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9111.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PTCH1 monomer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: PTCH1 dimer, C2 symmetry
| ファイル | emd_9111_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PTCH1 dimer, C2 symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
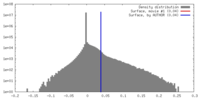
| 密度ヒストグラム |
-追加マップ: PTCH1 dimer, C1 symmetry
| ファイル | emd_9111_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | PTCH1 dimer, C1 symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Independent half map 2 used to calculate final monomer map
| ファイル | emd_9111_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Independent half map 2 used to calculate final monomer map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
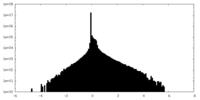
| 密度ヒストグラム |
-ハーフマップ: Independent half map 1 used to calculate final monomer map
| ファイル | emd_9111_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Independent half map 1 used to calculate final monomer map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Patched1 protein solubilized in amphipol
| 全体 | 名称: Patched1 protein solubilized in amphipol |
|---|---|
| 要素 |
|
-超分子 #1: Patched1 protein solubilized in amphipol
| 超分子 | 名称: Patched1 protein solubilized in amphipol / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: BacMam Homo sapiens (ヒト) / 組換細胞: HEK293 / 組換プラスミド: BacMam |
| 分子量 | 理論値: 145.2 KDa |
-分子 #1: Protein patched homolog 1
| 分子 | 名称: Protein patched homolog 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 145.357844 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MASAGNAAGA LGRQAGGGRR RRTGGPHRAA PDRDYLHRPS YCDAAFALEQ ISKGKATGRK APLWLRAKFQ RLLFKLGCYI QKNCGKFLV VGLLIFGAFA VGLKAANLET NVEELWVEVG GRVSRELNYT RQKIGEEAMF NPQLMIQTPK EEGANVLTTE A LLQHLDSA ...文字列: MASAGNAAGA LGRQAGGGRR RRTGGPHRAA PDRDYLHRPS YCDAAFALEQ ISKGKATGRK APLWLRAKFQ RLLFKLGCYI QKNCGKFLV VGLLIFGAFA VGLKAANLET NVEELWVEVG GRVSRELNYT RQKIGEEAMF NPQLMIQTPK EEGANVLTTE A LLQHLDSA LQASRVHVYM YNRQWKLEHL CYKSGELITE TGYMDQIIEY LYPCLIITPL DCFWEGAKLQ SGTAYLLGKP PL RWTNFDP LEFLEELKKI NYQVDSWEEM LNKAEVGHGY MDRPCLNPAD PDCPATAPNK NSTKPLDVAL VLNGGCQGLS RKY MHWQEE LIVGGTVKNA TGKLVSAHAL QTMFQLMTPK QMYEHFRGYD YVSHINWNED RAAAILEAWQ RTYVEVVHQS VAPN STQKV LPFTTTTLDD ILKSFSDVSV IRVASGYLLM LAYACLTMLR WDCSKSQGAV GLAGVLLVAL SVAAGLGLCS LIGIS FNAA TTQVLPFLAL GVGVDDVFLL AHAFSETGQN KRIPFEDRTG ECLKRTGASV ALTSISNVTA FFMAALIPIP ALRAFS LQA AVVVVFNFAM VLLIFPAILS MDLYRREDRR LDIFCCFTSP CVSRVIQVEP QAYTEPMQST VQLRTEYDPH THVYYTT AE PRSEISVQPV TVTQDNLSCQ SPESTSSTRD LLSQFSDSSL HCLEPPCTKW TLSSFAEKHY APFLLKPKAK VVVILLFL G LLGVSLYGTT RVRDGLDLTD IVPRETREYD FIAAQFKYFS FYNMYIVTQK ADYPNIQHLL YDLHKSFSNV KYVMLEENK QLPQMWLHYF RDWLQGLQDA FDSDWETGRI MPNNYKNGSD DGVLAYKLLV QTGSRDKPID ISQLTKQRLV DADGIINPSA FYIYLTAWV SNDPVAYAAS QANIRPHRPE WVHDKADYMP ETRLRIPAAE PIEYAQFPFY LNGLRDTSDF VEAIEKVRVI C NNYTSLGL SSYPNGYPFL FWEQYISLRH WLLLSISVVL ACTFLVCAVF LLNPWTAGII VMVLALMTVE LFGMMGLIGI KL SAVPVVI LIASVGIGVE FTVHVALAFL TAIGDKNHRA MLALEHMFAP VLDGAVSTLL GVLMLAGSEF DFIVRYFFAV LAI LTVLGV LNGLVLLPVL LSFFGPCPEV SPANGLNRLP TPSPEPPPSV VRFAVPPGHT NNGSDSSDSE YSSQTTVSGI SEEL RQYEA QQGAGGPAHQ VIVEATENPV FARSTVVHPD SRHQPPLTPR QQPHLDSGSL SPGRQGQQPR RDMDEKTTGW RGGHV VEGL AGELEQLRAR LEHHPQGQRE P |
-分子 #2: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 2 / コピー数: 4 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 23 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 実像数: 5236 / 平均露光時間: 8.0 sec. / 平均電子線量: 38.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6mg8: |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)