+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8f0p | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of VSD4-NaV1.7-NaVPas channel chimera bound to the hybrid inhibitor GNE-1305 | ||||||
 要素 要素 | Sodium channel protein PaFPC1,Sodium channel protein type 9 subunit alpha chimera | ||||||
 キーワード キーワード | MEMBRANE PROTEIN/INHIBITOR / Ion channel / small molecule / inhibitor / MEMBRANE PROTEIN-INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報action potential propagation / detection of mechanical stimulus involved in sensory perception / membrane depolarization during action potential / cardiac muscle cell action potential involved in contraction / voltage-gated sodium channel complex / node of Ranvier / Interaction between L1 and Ankyrins / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity / detection of temperature stimulus involved in sensory perception of pain ...action potential propagation / detection of mechanical stimulus involved in sensory perception / membrane depolarization during action potential / cardiac muscle cell action potential involved in contraction / voltage-gated sodium channel complex / node of Ranvier / Interaction between L1 and Ankyrins / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity / detection of temperature stimulus involved in sensory perception of pain / Phase 0 - rapid depolarisation / behavioral response to pain / neuronal action potential / axon terminus / sensory perception of pain / sodium ion transmembrane transport / post-embryonic development / circadian rhythm / response to toxic substance / Sensory perception of sweet, bitter, and umami (glutamate) taste / inflammatory response / axon / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) Periplaneta americana (ワモンゴキブリ) Periplaneta americana (ワモンゴキブリ) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.2 Å | ||||||
 データ登録者 データ登録者 | Kschonsak, M. / Jao, C.C. / Arthur, C.P. / Rohou, A.L. / Bergeron, P. / Ortwine, D. / McKerall, S.J. / Hackos, D.H. / Deng, L. / Chen, J. ...Kschonsak, M. / Jao, C.C. / Arthur, C.P. / Rohou, A.L. / Bergeron, P. / Ortwine, D. / McKerall, S.J. / Hackos, D.H. / Deng, L. / Chen, J. / Sutherlin, D. / Dragovich, P.S. / Volgraf, M. / Wright, M.R. / Payandeh, J. / Ciferri, C. / Tellis, J.C. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Cryo-EM reveals an unprecedented binding site for Na1.7 inhibitors enabling rational design of potent hybrid inhibitors. 著者: Marc Kschonsak / Christine C Jao / Christopher P Arthur / Alexis L Rohou / Philippe Bergeron / Daniel F Ortwine / Steven J McKerrall / David H Hackos / Lunbin Deng / Jun Chen / Tianbo Li / ...著者: Marc Kschonsak / Christine C Jao / Christopher P Arthur / Alexis L Rohou / Philippe Bergeron / Daniel F Ortwine / Steven J McKerrall / David H Hackos / Lunbin Deng / Jun Chen / Tianbo Li / Peter S Dragovich / Matthew Volgraf / Matthew R Wright / Jian Payandeh / Claudio Ciferri / John C Tellis /  要旨: The voltage-gated sodium (Na) channel Na1.7 has been identified as a potential novel analgesic target due to its involvement in human pain syndromes. However, clinically available Na channel-blocking ...The voltage-gated sodium (Na) channel Na1.7 has been identified as a potential novel analgesic target due to its involvement in human pain syndromes. However, clinically available Na channel-blocking drugs are not selective among the nine Na channel subtypes, Na1.1-Na1.9. Moreover, the two currently known classes of Na1.7 subtype-selective inhibitors (aryl- and acylsulfonamides) have undesirable characteristics that may limit their development. To this point understanding of the structure-activity relationships of the acylsulfonamide class of Na1.7 inhibitors, exemplified by the clinical development candidate , has been based solely on a single co-crystal structure of an arylsulfonamide inhibitor bound to voltage-sensing domain 4 (VSD4). To advance inhibitor design targeting the Na1.7 channel, we pursued high-resolution ligand-bound Na1.7-VSD4 structures using cryogenic electron microscopy (cryo-EM). Here, we report that engages the Na1.7-VSD4 through an unexpected binding mode orthogonal to the arylsulfonamide inhibitor class binding pose, which identifies a previously unknown ligand binding site in Na channels. This finding enabled the design of a novel hybrid inhibitor series that bridges the aryl- and acylsulfonamide binding pockets and allows for the generation of molecules with substantially differentiated structures and properties. Overall, our study highlights the power of cryo-EM methods to pursue challenging drug targets using iterative and high-resolution structure-guided inhibitor design. This work also underscores an important role of the membrane bilayer in the optimization of selective Na channel modulators targeting VSD4. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8f0p.cif.gz 8f0p.cif.gz | 246.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8f0p.ent.gz pdb8f0p.ent.gz | 191.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8f0p.json.gz 8f0p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/f0/8f0p https://data.pdbj.org/pub/pdb/validation_reports/f0/8f0p ftp://data.pdbj.org/pub/pdb/validation_reports/f0/8f0p ftp://data.pdbj.org/pub/pdb/validation_reports/f0/8f0p | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 184481.906 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: Chimeric construct of human Nav1.7 VSD4 and the NavPaS channel from American cockroach Periplaneta americana 由来: (組換発現)  Homo sapiens (ヒト), (組換発現) Homo sapiens (ヒト), (組換発現)  Periplaneta americana (ワモンゴキブリ) Periplaneta americana (ワモンゴキブリ)遺伝子: SCN9A, NENA / 細胞株 (発現宿主): 293 suspension / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: D0E0C2, UniProt: Q15858 Homo sapiens (ヒト) / 参照: UniProt: D0E0C2, UniProt: Q15858 |
|---|
-糖 , 2種, 4分子 
| #2: 多糖 | beta-D-mannopyranose-(1-3)-[beta-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-3)-2-acetamido-2- ...beta-D-mannopyranose-(1-3)-[beta-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-3)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-3)-2-acetamido-2-deoxy-beta-D-glucopyranose タイプ: oligosaccharide / 分子量: 910.823 Da / 分子数: 1 / 由来タイプ: 組換発現 |
|---|---|
| #3: 糖 |
-非ポリマー , 4種, 118分子 

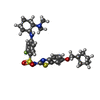




| #4: 化合物 | ChemComp-PEE / #5: 化合物 | ChemComp-Y01 / | #6: 化合物 | ChemComp-X7L / | #7: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: VSD4-NaV1.7-NaVPas channel chimera bound to the hybrid inhibitor GNE-1305 タイプ: COMPLEX 詳細: Chimeric construct of human Nav1.7 VSD4 and the NavPaS channel from American cockroach Periplaneta americana Entity ID: #1 / 由来: MULTIPLE SOURCES | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | |||||||||||||||
| 由来(天然) |
| |||||||||||||||
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) / 細胞: 293 suspension cells Homo sapiens (ヒト) / 細胞: 293 suspension cells | |||||||||||||||
| 緩衝液 | pH: 7.5 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 2 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: The sample was reconstituted into lipid nanodiscs (MSP1E3D1 in 3POPC:1POPE:1POPG) and was monodisperse. The sample was crosslinked with 0.05% glutaraldehyde for 10 minutes at RT, then quenched with 1M Tris pH7.0. | |||||||||||||||
| 試料支持 | 詳細: Grids incubated with a thiol reactive, self-assembling reaction mixture of 4mM monothiolalkane(C11)PEG6-OH (11-mercaptoundecyl) hexaethyleneglycol (SPT-0011P6, SensoPath Technologies, Inc., ...詳細: Grids incubated with a thiol reactive, self-assembling reaction mixture of 4mM monothiolalkane(C11)PEG6-OH (11-mercaptoundecyl) hexaethyleneglycol (SPT-0011P6, SensoPath Technologies, Inc., Bozeman, MT). Grids were incubated with this self-assembled monolayer (SAM) solution for 24 hours and afterwards rinsed with EtOH. グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: UltrAuFoil R0./1 | |||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277.15 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 500 nm |
| 撮影 | 平均露光時間: 3 sec. / 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: microgaphs with CTFfit of 6.0 A or better were selected タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2741402 詳細: template-matching particle picking with a 30A low-pass filtered template | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 1201168 詳細: A score threshold was applied so that only the best scoring particles are included in the 3D reconstruction at each cycle. 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj









