+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8793 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MERS S ectodomain trimer in complex with Fab of neutralizing antibody G4 | |||||||||
 マップデータ マップデータ | MERS S ectodomain trimer in complex with variable domain of neutralizing antibody G4 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endocytosis involved in viral entry into host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated virion attachment to host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / host cell plasma membrane / virion membrane / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.5 Å | |||||||||
 データ登録者 データ登録者 | Pallesen J / Ward AB | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Immunogenicity and structures of a rationally designed prefusion MERS-CoV spike antigen. 著者: Jesper Pallesen / Nianshuang Wang / Kizzmekia S Corbett / Daniel Wrapp / Robert N Kirchdoerfer / Hannah L Turner / Christopher A Cottrell / Michelle M Becker / Lingshu Wang / Wei Shi / Wing- ...著者: Jesper Pallesen / Nianshuang Wang / Kizzmekia S Corbett / Daniel Wrapp / Robert N Kirchdoerfer / Hannah L Turner / Christopher A Cottrell / Michelle M Becker / Lingshu Wang / Wei Shi / Wing-Pui Kong / Erica L Andres / Arminja N Kettenbach / Mark R Denison / James D Chappell / Barney S Graham / Andrew B Ward / Jason S McLellan /  要旨: Middle East respiratory syndrome coronavirus (MERS-CoV) is a lineage C betacoronavirus that since its emergence in 2012 has caused outbreaks in human populations with case-fatality rates of ∼36%. ...Middle East respiratory syndrome coronavirus (MERS-CoV) is a lineage C betacoronavirus that since its emergence in 2012 has caused outbreaks in human populations with case-fatality rates of ∼36%. As in other coronaviruses, the spike (S) glycoprotein of MERS-CoV mediates receptor recognition and membrane fusion and is the primary target of the humoral immune response during infection. Here we use structure-based design to develop a generalizable strategy for retaining coronavirus S proteins in the antigenically optimal prefusion conformation and demonstrate that our engineered immunogen is able to elicit high neutralizing antibody titers against MERS-CoV. We also determined high-resolution structures of the trimeric MERS-CoV S ectodomain in complex with G4, a stem-directed neutralizing antibody. The structures reveal that G4 recognizes a glycosylated loop that is variable among coronaviruses and they define four conformational states of the trimer wherein each receptor-binding domain is either tightly packed at the membrane-distal apex or rotated into a receptor-accessible conformation. Our studies suggest a potential mechanism for fusion initiation through sequential receptor-binding events and provide a foundation for the structure-based design of coronavirus vaccines. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8793.map.gz emd_8793.map.gz | 93.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8793-v30.xml emd-8793-v30.xml emd-8793.xml emd-8793.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8793_fsc.xml emd_8793_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8793.png emd_8793.png | 63.2 KB | ||
| その他 |  emd_8793_half_map_1.map.gz emd_8793_half_map_1.map.gz emd_8793_half_map_2.map.gz emd_8793_half_map_2.map.gz | 84.3 MB 84.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8793 http://ftp.pdbj.org/pub/emdb/structures/EMD-8793 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8793 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8793 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8793_validation.pdf.gz emd_8793_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8793_full_validation.pdf.gz emd_8793_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_8793_validation.xml.gz emd_8793_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8793 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8793 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8793 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8793 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8783C  8784C  8785C  8786C  8787C  8788C  8789C  8790C  8791C  8792C  5vyhC  5vzrC  5w9hC  5w9iC  5w9jC  5w9kC  5w9lC  5w9mC  5w9nC  5w9oC  5w9pC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8793.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8793.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MERS S ectodomain trimer in complex with variable domain of neutralizing antibody G4 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.02 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: MERS S ectodomain trimer in complex with variable...
| ファイル | emd_8793_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MERS S ectodomain trimer in complex with variable domain of neutralizing antibody G4 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
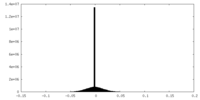
| 密度ヒストグラム |
-ハーフマップ: MERS S ectodomain trimer in complex with variable...
| ファイル | emd_8793_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MERS S ectodomain trimer in complex with variable domain of neutralizing antibody G4 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
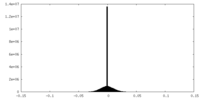
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MERS S ectodomain trimer in complex with Fab of neutralizing anti...
| 全体 | 名称: MERS S ectodomain trimer in complex with Fab of neutralizing antibody G4 |
|---|---|
| 要素 |
|
-超分子 #1: MERS S ectodomain trimer in complex with Fab of neutralizing anti...
| 超分子 | 名称: MERS S ectodomain trimer in complex with Fab of neutralizing antibody G4 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 600 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 名称: TBS |
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-35 / 撮影したグリッド数: 1 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.89 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)