+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8624 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo EM structure of anti-CRISPRs, AcrF1 and AcrF2, bound to type I-F crRNA-guided CRISPR surveillance complex | ||||||||||||
 マップデータ マップデータ | Cryo EM map of the Csy-Acr complex. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | CRISPR RNA-recognition-motif (RRM) / pseudo-helical / Type 1-F CRISPR / IMMUNE SYSTEM-RNA complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host CRISPR-cas system / symbiont-mediated suppression of host adaptive immune response / maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / virus-mediated perturbation of host defense response / RNA binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) / Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) /  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) / Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) /  Pseudomonas phage JBD30 (ファージ) / Pseudomonas phage JBD30 (ファージ) /  Pseudomonas phage D3112 (ファージ) Pseudomonas phage D3112 (ファージ) | ||||||||||||
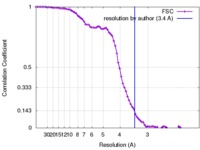
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | ||||||||||||
 データ登録者 データ登録者 | Chowdhury S / Carter J | ||||||||||||
| 資金援助 |  米国, 米国,  カナダ, 3件 カナダ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Structure Reveals Mechanisms of Viral Suppressors that Intercept a CRISPR RNA-Guided Surveillance Complex. 著者: Saikat Chowdhury / Joshua Carter / MaryClare F Rollins / Sarah M Golden / Ryan N Jackson / Connor Hoffmann / Lyn'Al Nosaka / Joseph Bondy-Denomy / Karen L Maxwell / Alan R Davidson / ...著者: Saikat Chowdhury / Joshua Carter / MaryClare F Rollins / Sarah M Golden / Ryan N Jackson / Connor Hoffmann / Lyn'Al Nosaka / Joseph Bondy-Denomy / Karen L Maxwell / Alan R Davidson / Elizabeth R Fischer / Gabriel C Lander / Blake Wiedenheft /   要旨: Genetic conflict between viruses and their hosts drives evolution and genetic innovation. Prokaryotes evolved CRISPR-mediated adaptive immune systems for protection from viral infection, and viruses ...Genetic conflict between viruses and their hosts drives evolution and genetic innovation. Prokaryotes evolved CRISPR-mediated adaptive immune systems for protection from viral infection, and viruses have evolved diverse anti-CRISPR (Acr) proteins that subvert these immune systems. The adaptive immune system in Pseudomonas aeruginosa (type I-F) relies on a 350 kDa CRISPR RNA (crRNA)-guided surveillance complex (Csy complex) to bind foreign DNA and recruit a trans-acting nuclease for target degradation. Here, we report the cryo-electron microscopy (cryo-EM) structure of the Csy complex bound to two different Acr proteins, AcrF1 and AcrF2, at an average resolution of 3.4 Å. The structure explains the molecular mechanism for immune system suppression, and structure-guided mutations show that the Acr proteins bind to residues essential for crRNA-mediated detection of DNA. Collectively, these data provide a snapshot of an ongoing molecular arms race between viral suppressors and the immune system they target. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8624.map.gz emd_8624.map.gz | 5.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8624-v30.xml emd-8624-v30.xml emd-8624.xml emd-8624.xml | 33.9 KB 33.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8624_fsc.xml emd_8624_fsc.xml | 8.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8624.png emd_8624.png | 175.4 KB | ||
| Filedesc metadata |  emd-8624.cif.gz emd-8624.cif.gz | 9 KB | ||
| その他 |  emd_8624_additional_1.map.gz emd_8624_additional_1.map.gz emd_8624_additional_2.map.gz emd_8624_additional_2.map.gz emd_8624_half_map_1.map.gz emd_8624_half_map_1.map.gz emd_8624_half_map_2.map.gz emd_8624_half_map_2.map.gz | 1.9 MB 31 MB 4.8 MB 4.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8624 http://ftp.pdbj.org/pub/emdb/structures/EMD-8624 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8624 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8624 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8624_validation.pdf.gz emd_8624_validation.pdf.gz | 662.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8624_full_validation.pdf.gz emd_8624_full_validation.pdf.gz | 662.2 KB | 表示 | |
| XML形式データ |  emd_8624_validation.xml.gz emd_8624_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_8624_validation.cif.gz emd_8624_validation.cif.gz | 18.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8624 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8624 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8624 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8624 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8624.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8624.map.gz / 形式: CCP4 / 大きさ: 35.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo EM map of the Csy-Acr complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Focused map of the "tail" region of Csy-Acr...
| ファイル | emd_8624_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map of the "tail" region of Csy-Acr complex, comprising of Cas5, Cas8 and Acr2 subunits. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened Cryo EM map of the Csy-Acr complex.
| ファイル | emd_8624_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened Cryo EM map of the Csy-Acr complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 of the Csy-Acr complex.
| ファイル | emd_8624_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 of the Csy-Acr complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1 of the Csy-Acr complex.
| ファイル | emd_8624_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 of the Csy-Acr complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Anti-CRISPRs AcrF1 and AcrF2 bound P. aeruginosa crRNA-guided CRI...
| 全体 | 名称: Anti-CRISPRs AcrF1 and AcrF2 bound P. aeruginosa crRNA-guided CRISPR surveillance(Csy)complex. |
|---|---|
| 要素 |
|
-超分子 #1: Anti-CRISPRs AcrF1 and AcrF2 bound P. aeruginosa crRNA-guided CRI...
| 超分子 | 名称: Anti-CRISPRs AcrF1 and AcrF2 bound P. aeruginosa crRNA-guided CRISPR surveillance(Csy)complex. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Cas5F, Cas6F, Cas7F, Cas8F and CRISPR RNA (crRNA) forms the Csy surveillance complex. Cas5F, Cas8F and 5'crRNA handle forms the "tail" of the complex, and 3' crRNA stem-loop along with Cas6F ...詳細: Cas5F, Cas6F, Cas7F, Cas8F and CRISPR RNA (crRNA) forms the Csy surveillance complex. Cas5F, Cas8F and 5'crRNA handle forms the "tail" of the complex, and 3' crRNA stem-loop along with Cas6F forms the "head". Six copies Cas7F along with crRNA spacer forms the "core" of the complex. |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) |
| 分子量 | 理論値: 450 KDa |
-分子 #1: CRISPR-associated protein Csy1
| 分子 | 名称: CRISPR-associated protein Csy1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 49.194168 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSPLPTPTW QELRQFIESF IQERLQGKLD KLQPDEDDKR QTLLATHRRE AWLADAARRV GQLQLVTHTL KPIHPDARGS NLHSLPQAP GQPGLAGSHE LGDRLVSDVV GNAAALDVFK FLSLQYQGKN LLNWLTEDSA EALQALSDNA EQAREWRQAF I GITTVKGA ...文字列: MTSPLPTPTW QELRQFIESF IQERLQGKLD KLQPDEDDKR QTLLATHRRE AWLADAARRV GQLQLVTHTL KPIHPDARGS NLHSLPQAP GQPGLAGSHE LGDRLVSDVV GNAAALDVFK FLSLQYQGKN LLNWLTEDSA EALQALSDNA EQAREWRQAF I GITTVKGA PASHSLAKQL YFPLPGSGYH LLAPLFPTSL VHHVHALLRE ARFGDAAKAA REARSRQESW PHGFSEYPNL AI QKFGGTK PQNISQLNNE RRGENWLLPS LPPNWQRQNV NAPMRHSSVF EHDFGRTPEV SRLTRTLQRF LAKTVHNNLA IRQ RRAQLV AQICDEALQY AARLRELEPG WSATPGCQLH DAEQLWLDPL RAQTDETFLQ RRLRGDWPAE VGNRFANWLN RAVS SDSQI LGSPEAAQWS QELSKELTMF KEILEDERD UniProtKB: CRISPR-associated protein Csy1 |
-分子 #2: CRISPR-associated protein Csy2
| 分子 | 名称: CRISPR-associated protein Csy2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 36.244074 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVTDPEALL LLPRLSIQNA NAISSPLTWG FPSPGAFTGF VHALQRRVGI SLDIELDGVG IVCHRFEAQI SQPAGKRTKV FNLTRNPLN RDGSTAAIVE EGRAHLEVSL LLGVHGDGLD DHPAQEIARQ VQEQAGAMRL AGGSILPWCN ERFPAPNAEL L MLGGSDEQ ...文字列: MSVTDPEALL LLPRLSIQNA NAISSPLTWG FPSPGAFTGF VHALQRRVGI SLDIELDGVG IVCHRFEAQI SQPAGKRTKV FNLTRNPLN RDGSTAAIVE EGRAHLEVSL LLGVHGDGLD DHPAQEIARQ VQEQAGAMRL AGGSILPWCN ERFPAPNAEL L MLGGSDEQ RRKNQRRLTR RLLPGFALVS REALLQQHLE TLRTTLPEAT TLDALLDLCR INFEPPATSS EEEASPPDAA WQ VRDKPGW LVPIPAGYNA LSPLYLPGEV RNARDRETPL RFVENLFGLG EWLSPHRVAA LSDLLWYHHA EPDKGLYRWS TPR FVEHAI A UniProtKB: CRISPR-associated protein Csy2 |
-分子 #3: CRISPR-associated protein Csy3
| 分子 | 名称: CRISPR-associated protein Csy3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 37.448078 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SKPILSTASV LAFERKLDPS DALMSAGAWA QRDASQEWPA VTVREKSVRG TISNRLKTKD RDPAKLDASI QSPNLQTVDV ANLPSDADT LKVRFTLRVL GGAGTPSACN DAAYRDKLLQ TVATYVNDQG FAELARRYAH NLANARFLWR NRVGAEAVEV R INHIRQGE ...文字列: SKPILSTASV LAFERKLDPS DALMSAGAWA QRDASQEWPA VTVREKSVRG TISNRLKTKD RDPAKLDASI QSPNLQTVDV ANLPSDADT LKVRFTLRVL GGAGTPSACN DAAYRDKLLQ TVATYVNDQG FAELARRYAH NLANARFLWR NRVGAEAVEV R INHIRQGE VARAWRFDAL AIGLRDFKAD AELDALAELI ASGLSGSGHV LLEVVAFARI GDGQEVFPSQ ELILDKGDKK GQ KSKTLYS VRDAAAIHSQ KIGNALRTID TWYPDEDGLG PIAVEPYGSV TSQGKAYRQP KQKLDFYTLL DNWVLRDEAP AVE QQHYVI ANLIRGGVFG EAEEK UniProtKB: CRISPR-associated protein Csy3 |
-分子 #4: Anti-CRISPR protein Acr30-35
| 分子 | 名称: Anti-CRISPR protein Acr30-35 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage JBD30 (ファージ) Pseudomonas phage JBD30 (ファージ) |
| 分子量 | 理論値: 8.693734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KFIKYLSTAH LNYMNIAVYE NGSKIKARVE NVVNGKSVGA RDFDSTEQLE SWFYGLPGSG LGRIENAMNE ISRRENP UniProtKB: Uncharacterized protein |
-分子 #5: Anti-CRISPR protein 30
| 分子 | 名称: Anti-CRISPR protein 30 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage D3112 (ファージ) Pseudomonas phage D3112 (ファージ) |
| 分子量 | 理論値: 10.760481 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHIAQ QHKDTVAACE AAEAIAIAKD QVWDGEGYTK YTFDDNSVLI QSGTTQYAMD ADDADSIKGY ADWLDDEARS AEASEIERL LESVEEE UniProtKB: Anti-CRISPR protein 30 |
-分子 #6: CRISPR-associated endonuclease Cas6/Csy4
| 分子 | 名称: CRISPR-associated endonuclease Cas6/Csy4 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌) Pseudomonas aeruginosa (strain UCBPP-PA14) (緑膿菌)株: UCBPP-PA14 |
| 分子量 | 理論値: 21.691848 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: FTMDHYLDIR LRPDPEFPPA QLMCVLFGKL HQALVAQGGD RIGVSFPDLD ESRSRLGERL RIHASADDLR ALLARPWLEG LRDHLQFGE PAVVPHPTPY RQVSRVQAKS NPERLRRRLM RRHDLSEEEA RKRIPDTVAR ALDLPFVTLR SQSTGQHFRL F IRHGPLQV ...文字列: FTMDHYLDIR LRPDPEFPPA QLMCVLFGKL HQALVAQGGD RIGVSFPDLD ESRSRLGERL RIHASADDLR ALLARPWLEG LRDHLQFGE PAVVPHPTPY RQVSRVQAKS NPERLRRRLM RRHDLSEEEA RKRIPDTVAR ALDLPFVTLR SQSTGQHFRL F IRHGPLQV TAEEGGFTCY GLSKGGFVPW F UniProtKB: CRISPR-associated endonuclease Cas6/Csy4 |
-分子 #7: CRISPR RNA (60-MER)
| 分子 | 名称: CRISPR RNA (60-MER) / タイプ: rna / ID: 7 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) Pseudomonas aeruginosa UCBPP-PA14 (緑膿菌) |
| 分子量 | 理論値: 19.249404 KDa |
| 配列 | 文字列: CUAAGAAAUU CACGGCGGGC UUGAUGUCCG CGUCUACCUG GUUCACUGCC GUAUAGGCAG GENBANK: GENBANK: CP000438.1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer was filtered before use. | ||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 5 sec. / 前処理 - 雰囲気: OTHER 詳細: Holey grid was coated with an amorphous carbon film. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 98 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER 詳細: Sample was applied to poly-L-lysine hydrobromide pre-treated amorphous carbon film coated over a holey grid. Excess sample was blotted with Whatman-1 filter paper and plunge frozen. Manual ...詳細: Sample was applied to poly-L-lysine hydrobromide pre-treated amorphous carbon film coated over a holey grid. Excess sample was blotted with Whatman-1 filter paper and plunge frozen. Manual plunge freezing was performed in a cold room.. | ||||||||||||
| 詳細 | Sample was free from any aggregation and monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 79.0 K |
| 詳細 | Objective astigmatism was corrected at nominal magnification of 29000X, using Thon rings visualized with a K2 camera. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-30 / 撮影したグリッド数: 2 / 実像数: 2261 / 平均露光時間: 6.0 sec. / 平均電子線量: 46.0 e/Å2 詳細: Images were collected in movie mode using Leginon automated data acquisition software. Movie frames were aligned using MotionCorr program. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Top scoring five models out of two hundred models generated using Rosetta were refined using Phenix and deposited as an ensemble atomic model for the final EM map. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-5uz9: |
-原子モデル構築 2
| 詳細 | The docked Cas6F structure in the "head" region of the complex was reduced to C-alpha backbone. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-5uz9: |
-原子モデル構築 3
| 詳細 | Initially two copies were fitted using Chimera and then backbones and side chains were fixed using Coot, followed by real space refinement in Phenix. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-5uz9: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X