+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7qhh | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Desensitized state of GluA1/2 AMPA receptor in complex with TARP-gamma 8 (TMD-LBD) | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | MEMBRANE PROTEIN / glutamate / AMPA receptor / TARPs | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Phase 2 - plateau phase / Phase 0 - rapid depolarisation / Cargo concentration in the ER / cellular response to amine stimulus / axonal spine / COPII-mediated vesicle transport / positive regulation of membrane potential / cellular response to ammonium ion / neurotransmitter receptor transport, postsynaptic endosome to lysosome / L-type voltage-gated calcium channel complex ...Phase 2 - plateau phase / Phase 0 - rapid depolarisation / Cargo concentration in the ER / cellular response to amine stimulus / axonal spine / COPII-mediated vesicle transport / positive regulation of membrane potential / cellular response to ammonium ion / neurotransmitter receptor transport, postsynaptic endosome to lysosome / L-type voltage-gated calcium channel complex / neurotransmitter receptor activity involved in regulation of postsynaptic cytosolic calcium ion concentration / LGI-ADAM interactions / myosin V binding / Trafficking of AMPA receptors / neuron spine / regulation of AMPA receptor activity / neurotransmitter receptor internalization / channel regulator activity / protein phosphatase 2B binding / response to arsenic-containing substance / cellular response to dsRNA / postsynaptic neurotransmitter receptor diffusion trapping / dendritic spine membrane / Synaptic adhesion-like molecules / glutamate-gated calcium ion channel activity / long-term synaptic depression / cellular response to peptide hormone stimulus / protein kinase A binding / spinal cord development / spine synapse / neuronal cell body membrane / dendritic spine neck / dendritic spine head / Activation of AMPA receptors / perisynaptic space / transmission of nerve impulse / AMPA glutamate receptor activity / regulation of postsynaptic membrane neurotransmitter receptor levels / Trafficking of GluR2-containing AMPA receptors / response to lithium ion / immunoglobulin binding / AMPA glutamate receptor complex / kainate selective glutamate receptor activity / cellular response to organic cyclic compound / adenylate cyclase binding / ionotropic glutamate receptor complex / extracellularly glutamate-gated ion channel activity / cellular response to glycine / excitatory synapse / calcium channel regulator activity / asymmetric synapse / regulation of receptor recycling / G-protein alpha-subunit binding / voltage-gated calcium channel activity / Unblocking of NMDA receptors, glutamate binding and activation / neuronal action potential / regulation of postsynaptic membrane potential / postsynaptic density, intracellular component / glutamate receptor binding / positive regulation of synaptic transmission / long-term memory / response to electrical stimulus / glutamate-gated receptor activity / presynaptic active zone membrane / beta-2 adrenergic receptor binding / response to fungicide / regulation of synaptic transmission, glutamatergic / somatodendritic compartment / cellular response to brain-derived neurotrophic factor stimulus / dendrite membrane / synapse assembly / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / cytoskeletal protein binding / ionotropic glutamate receptor signaling pathway / dendrite cytoplasm / positive regulation of synaptic transmission, glutamatergic / SNARE binding / response to cocaine / dendritic shaft / synaptic membrane / synaptic transmission, glutamatergic / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / PDZ domain binding / long-term synaptic potentiation / protein tetramerization / cellular response to amino acid stimulus / postsynaptic density membrane / ionotropic glutamate receptor binding / Schaffer collateral - CA1 synapse / modulation of chemical synaptic transmission / establishment of protein localization / regulation of synaptic plasticity / neuromuscular junction / response to organic cyclic compound / terminal bouton / receptor internalization / recycling endosome / response to toxic substance / cerebral cortex development / cellular response to growth factor stimulus 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||
 データ登録者 データ登録者 | Herguedas, B. / Kohegyi, B. / Dohrke, J.N. / Watson, J.F. / Zhang, D. / Ho, H. / Shaikh, S. / Lape, R. / Krieger, J.M. / Greger, I.H. | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Mechanisms underlying TARP modulation of the GluA1/2-γ8 AMPA receptor. 著者: Beatriz Herguedas / Bianka K Kohegyi / Jan-Niklas Dohrke / Jake F Watson / Danyang Zhang / Hinze Ho / Saher A Shaikh / Remigijus Lape / James M Krieger / Ingo H Greger /     要旨: AMPA-type glutamate receptors (AMPARs) mediate rapid signal transmission at excitatory synapses in the brain. Glutamate binding to the receptor's ligand-binding domains (LBDs) leads to ion channel ...AMPA-type glutamate receptors (AMPARs) mediate rapid signal transmission at excitatory synapses in the brain. Glutamate binding to the receptor's ligand-binding domains (LBDs) leads to ion channel activation and desensitization. Gating kinetics shape synaptic transmission and are strongly modulated by transmembrane AMPAR regulatory proteins (TARPs) through currently incompletely resolved mechanisms. Here, electron cryo-microscopy structures of the GluA1/2 TARP-γ8 complex, in both open and desensitized states (at 3.5 Å), reveal state-selective engagement of the LBDs by the large TARP-γ8 loop ('β1'), elucidating how this TARP stabilizes specific gating states. We further show how TARPs alter channel rectification, by interacting with the pore helix of the selectivity filter. Lastly, we reveal that the Q/R-editing site couples the channel constriction at the filter entrance to the gate, and forms the major cation binding site in the conduction path. Our results provide a mechanistic framework of how TARPs modulate AMPAR gating and conductance. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7qhh.cif.gz 7qhh.cif.gz | 393 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7qhh.ent.gz pdb7qhh.ent.gz | 287.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7qhh.json.gz 7qhh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7qhh_validation.pdf.gz 7qhh_validation.pdf.gz | 1.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7qhh_full_validation.pdf.gz 7qhh_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  7qhh_validation.xml.gz 7qhh_validation.xml.gz | 60.6 KB | 表示 | |
| CIF形式データ |  7qhh_validation.cif.gz 7qhh_validation.cif.gz | 84.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qh/7qhh https://data.pdbj.org/pub/pdb/validation_reports/qh/7qhh ftp://data.pdbj.org/pub/pdb/validation_reports/qh/7qhh ftp://data.pdbj.org/pub/pdb/validation_reports/qh/7qhh | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCSアンサンブル:
|
- 要素
要素
-Isoform Flip of Glutamate receptor ... , 2種, 4分子 ACBD
| #1: タンパク質 | 分子量: 102661.930 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 参照: UniProt: P19490 Homo sapiens (ヒト) / 参照: UniProt: P19490#2: タンパク質 | 分子量: 96247.055 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 参照: UniProt: P19491 Homo sapiens (ヒト) / 参照: UniProt: P19491 |
|---|
-タンパク質 , 1種, 2分子 JI
| #3: タンパク質 | 分子量: 43576.004 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 参照: UniProt: Q8VHW5 Homo sapiens (ヒト) / 参照: UniProt: Q8VHW5 |
|---|
-非ポリマー , 4種, 18分子 
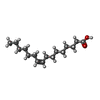

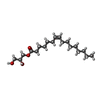



| #4: 化合物 | ChemComp-GLU / #5: 化合物 | ChemComp-PAM / #6: 化合物 | #7: 化合物 | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex between GluA1/2 AMPA receptor and auxiliary subunit TARP gamma8 タイプ: COMPLEX / 詳細: GluA2 and TARP8 are expressed as a tandem construct / Entity ID: #1-#3 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 0.490 MDa / 実験値: NO |
| 由来(天然) | 生物種:  |
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) / 細胞: HEK293 EXPI / プラスミド: PRK5 Homo sapiens (ヒト) / 細胞: HEK293 EXPI / プラスミド: PRK5 |
| 緩衝液 | pH: 8 詳細: 25 mM TRIS 150 mM NaCl 0.02 % GDN 87 uM CTZ 100 mM L-Glu |
| 試料 | 濃度: 2.5 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES 詳細: Sample was incubated with 87 uM CTZ for 30 minutes and 100 mM L-Glutamate was added before grid preparation |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K / 詳細: 3-4 second blots |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 2200 nm / 最小 デフォーカス(公称値): 340 nm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 4 sec. / 電子線照射量: 51 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 9664 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0267 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 105918 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL 詳細: First, rigid body fit was performed in Chimera using the coordinates of the TMD of 6QKC and the coordinates of the LBD in PDB code 3TKD. Manual model building was performed with Coot and ...詳細: First, rigid body fit was performed in Chimera using the coordinates of the TMD of 6QKC and the coordinates of the LBD in PDB code 3TKD. Manual model building was performed with Coot and Refinement was performed with Refmac. The TMD and LBD maps obtained independently aided model building but were not used for refinement. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.6→339.2 Å / Cor.coef. Fo:Fc: 0.664 / SU B: 23.138 / SU ML: 0.306 / ESU R: 0.153 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 81.995 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 13622 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj















