+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6igz | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of PSI-LHCI | ||||||||||||||||||||||||||||||||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | PLANT PROTEIN / PSI-LHCI | ||||||||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thylakoid membrane / photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting / chloroplast thylakoid lumen / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / photosystem II / chlorophyll binding ...thylakoid membrane / photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting / chloroplast thylakoid lumen / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / photosystem II / chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus / photosynthesis / chloroplast / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / metal ion binding 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||||||||||||||||||||
| 生物種 |  Bryopsis corticulans (ネザシハネモ) Bryopsis corticulans (ネザシハネモ) | ||||||||||||||||||||||||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.49 Å | ||||||||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Xiong, P. / Xiaochun, Q. | ||||||||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2019 ジャーナル: Nat Plants / 年: 2019タイトル: Structure of a green algal photosystem I in complex with a large number of light-harvesting complex I subunits. 著者: Xiaochun Qin / Xiong Pi / Wenda Wang / Guangye Han / Lixia Zhu / Mingmei Liu / Linpeng Cheng / Jian-Ren Shen / Tingyun Kuang / Sen-Fang Sui /   要旨: Photosystem I (PSI) is a highly efficient natural light-energy converter, and has diverse light-harvesting antennas associated with its core in different photosynthetic organisms. In green algae, an ...Photosystem I (PSI) is a highly efficient natural light-energy converter, and has diverse light-harvesting antennas associated with its core in different photosynthetic organisms. In green algae, an extremely large light-harvesting complex I (LHCI) captures and transfers energy to the PSI core. Here, we report the structure of PSI-LHCI from a green alga Bryopsis corticulans at 3.49 Å resolution, obtained by single-particle cryo-electron microscopy, which revealed 13 core subunits including subunits characteristic of both prokaryotes and eukaryotes, and 10 light-harvesting complex a (Lhca) antennas that form a double semi-ring and an additional Lhca dimer, including a novel four-transmembrane-helix Lhca. In total, 244 chlorophylls were identified, some of which were located at key positions for the fast energy transfer. These results provide a firm structural basis for unravelling the mechanisms of light-energy harvesting, transfer and quenching in the green algal PSI-LHCI, and important clues as to how PSI-LHCI has changed during evolution. | ||||||||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6igz.cif.gz 6igz.cif.gz | 1.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6igz.ent.gz pdb6igz.ent.gz | 1.1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6igz.json.gz 6igz.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6igz_validation.pdf.gz 6igz_validation.pdf.gz | 16.5 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6igz_full_validation.pdf.gz 6igz_full_validation.pdf.gz | 17.6 MB | 表示 | |
| XML形式データ |  6igz_validation.xml.gz 6igz_validation.xml.gz | 320.4 KB | 表示 | |
| CIF形式データ |  6igz_validation.cif.gz 6igz_validation.cif.gz | 377.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ig/6igz https://data.pdbj.org/pub/pdb/validation_reports/ig/6igz ftp://data.pdbj.org/pub/pdb/validation_reports/ig/6igz ftp://data.pdbj.org/pub/pdb/validation_reports/ig/6igz | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 18種, 20分子 ABCDEFGHKL1523486790
| #1: タンパク質 | 分子量: 83412.336 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ2*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ2*PLUS | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #2: タンパク質 | 分子量: 82164.461 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H003*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H003*PLUS | ||||||||||||||
| #3: タンパク質 | 分子量: 8880.307 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H005*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H005*PLUS | ||||||||||||||
| #4: タンパク質 | 分子量: 21733.045 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H006*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H006*PLUS | ||||||||||||||
| #5: タンパク質 | 分子量: 10096.458 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H007*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H007*PLUS | ||||||||||||||
| #6: タンパク質 | 分子量: 25129.158 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H008*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H008*PLUS | ||||||||||||||
| #7: タンパク質 | 分子量: 18226.600 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) Bryopsis corticulans (ネザシハネモ) | ||||||||||||||
| #8: タンパク質 | 分子量: 14400.360 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H009*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H009*PLUS | ||||||||||||||
| #11: タンパク質 | 分子量: 12487.672 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ4*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ4*PLUS | ||||||||||||||
| #12: タンパク質 | 分子量: 21338.602 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ5*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ5*PLUS | ||||||||||||||
| #13: タンパク質 | 分子量: 24458.012 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ6*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ6*PLUS#14: タンパク質 | | 分子量: 27808.264 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ7*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ7*PLUS#15: タンパク質 | | 分子量: 30409.689 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ8*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ8*PLUS#16: タンパク質 | 分子量: 26575.912 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ9*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ9*PLUS#17: タンパク質 | | 分子量: 29300.680 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H000*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H000*PLUS#18: タンパク質 | | 分子量: 28621.643 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H001*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H001*PLUS#19: タンパク質 | | 分子量: 24152.645 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H002*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H002*PLUS#20: タンパク質 | | 分子量: 26910.092 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H004*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H004*PLUS |
-タンパク質・ペプチド , 3種, 3分子 IJM
| #9: タンパク質・ペプチド | 分子量: 3859.615 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H010*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8H010*PLUS |
|---|---|
| #10: タンパク質・ペプチド | 分子量: 4744.679 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ3*PLUS Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A4V8GZZ3*PLUS |
| #21: タンパク質・ペプチド | 分子量: 3505.114 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A0D6E2L8 Bryopsis corticulans (ネザシハネモ) / 参照: UniProt: A0A0D6E2L8 |
-糖 , 2種, 3分子 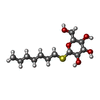


| #26: 糖 | | #28: 糖 | ChemComp-DGD / | |
|---|
-非ポリマー , 8種, 317分子 


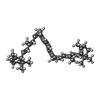











| #22: 化合物 | ChemComp-CLA / #23: 化合物 | #24: 化合物 | ChemComp-LHG / #25: 化合物 | ChemComp-8CT / ( #27: 化合物 | #29: 化合物 | ChemComp-CHL / #30: 化合物 | ChemComp-XAT / ( #31: 化合物 | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: PSI-LHCI / タイプ: COMPLEX / Entity ID: #1-#21 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:  Bryopsis corticulans (ネザシハネモ) Bryopsis corticulans (ネザシハネモ) |
| 緩衝液 | pH: 7.8 |
| 試料 | 濃度: 3 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 1.852 e/Å2 フィルム・検出器のモデル: FEI FALCON II (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.10.1_2155: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||
| CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.49 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 59525 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj



















