+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6000 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full virus map of Brome Mosaic Virus | |||||||||
 マップデータ マップデータ | full BMV map with combined data | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Bromovirus coat protein / Bromovirus coat protein / T=3 icosahedral viral capsid / host cell endoplasmic reticulum / viral nucleocapsid / ribonucleoprotein complex / structural molecule activity / RNA binding / Capsid protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Brome mosaic virus (ウイルス) Brome mosaic virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Wang Z / Hryc FC / Bammes B / Afonine PV / Jakana J / Chen DH / Liu XA / Baker ML / Kao C / Ludtke SJ ...Wang Z / Hryc FC / Bammes B / Afonine PV / Jakana J / Chen DH / Liu XA / Baker ML / Kao C / Ludtke SJ / Schmid MF / Adams PD / Chiu W | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2014 ジャーナル: Nat Commun / 年: 2014タイトル: An atomic model of brome mosaic virus using direct electron detection and real-space optimization. 著者: Zhao Wang / Corey F Hryc / Benjamin Bammes / Pavel V Afonine / Joanita Jakana / Dong-Hua Chen / Xiangan Liu / Matthew L Baker / Cheng Kao / Steven J Ludtke / Michael F Schmid / Paul D Adams / Wah Chiu /  要旨: Advances in electron cryo-microscopy have enabled structure determination of macromolecules at near-atomic resolution. However, structure determination, even using de novo methods, remains ...Advances in electron cryo-microscopy have enabled structure determination of macromolecules at near-atomic resolution. However, structure determination, even using de novo methods, remains susceptible to model bias and overfitting. Here we describe a complete workflow for data acquisition, image processing, all-atom modelling and validation of brome mosaic virus, an RNA virus. Data were collected with a direct electron detector in integrating mode and an exposure beyond the traditional radiation damage limit. The final density map has a resolution of 3.8 Å as assessed by two independent data sets and maps. We used the map to derive an all-atom model with a newly implemented real-space optimization protocol. The validity of the model was verified by its match with the density map and a previous model from X-ray crystallography, as well as the internal consistency of models from independent maps. This study demonstrates a practical approach to obtain a rigorously validated atomic resolution electron cryo-microscopy structure. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6000.map.gz emd_6000.map.gz | 203 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6000-v30.xml emd-6000-v30.xml emd-6000.xml emd-6000.xml | 9 KB 9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-6000_image.png EMD-6000_image.png | 967.4 KB | ||
| その他 |  emd_6000_additional_1.map.gz emd_6000_additional_1.map.gz emd_6000_additional_2.map.gz emd_6000_additional_2.map.gz emd_6000_additional_3.map.gz emd_6000_additional_3.map.gz emd_6000_additional_4.map.gz emd_6000_additional_4.map.gz emd_6000_additional_5.map.gz emd_6000_additional_5.map.gz | 392 KB 404.3 KB 404.5 KB 229.9 MB 229.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6000 http://ftp.pdbj.org/pub/emdb/structures/EMD-6000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6000 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6000_validation.pdf.gz emd_6000_validation.pdf.gz | 387.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6000_full_validation.pdf.gz emd_6000_full_validation.pdf.gz | 386.9 KB | 表示 | |
| XML形式データ |  emd_6000_validation.xml.gz emd_6000_validation.xml.gz | 7.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6000 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6000 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3j7lMC  3j7mMC  3j7nMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10010 (タイトル: Full virus map of Brome Mosaic Virus (micrographs and particle coordinates) EMPIAR-10010 (タイトル: Full virus map of Brome Mosaic Virus (micrographs and particle coordinates)Data size: 1.7 TB Data #1: Brome Mosaic Virus micrographs - non gain corrected [micrographs - multiframe] Data #2: Brome Mosaic Virus micrographs - gain corrected [micrographs - multiframe])  EMPIAR-10011 (タイトル: Full virus map of Brome Mosaic Virus (picked particles) EMPIAR-10011 (タイトル: Full virus map of Brome Mosaic Virus (picked particles)Data size: 23.1 Data #1: Brome Mosaic Virus boxed particles [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6000.map.gz / 形式: CCP4 / 大きさ: 276 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6000.map.gz / 形式: CCP4 / 大きさ: 276 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
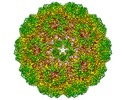
| 注釈 | full BMV map with combined data | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.99 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
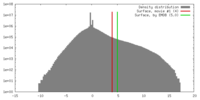
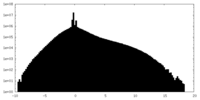
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 6000 additional 1.map
| ファイル | emd_6000_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
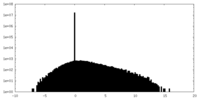
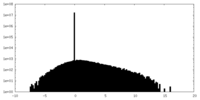
| 密度ヒストグラム |
-添付マップデータ: emd 6000 additional 2.map
| ファイル | emd_6000_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
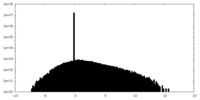
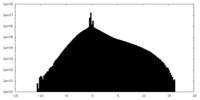
| 密度ヒストグラム |
-添付マップデータ: emd 6000 additional 3.map
| ファイル | emd_6000_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
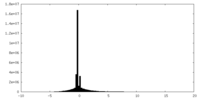
| 密度ヒストグラム |
-添付マップデータ: emd 6000 additional 4.map
| ファイル | emd_6000_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 6000 additional 5.map
| ファイル | emd_6000_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : brome mosaic virus
| 全体 | 名称:   brome mosaic virus (ウイルス) brome mosaic virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1000: brome mosaic virus
| 超分子 | 名称: brome mosaic virus / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 4.6 MDa |
-超分子 #1: Brome mosaic virus
| 超分子 | 名称: Brome mosaic virus / タイプ: virus / ID: 1 / NCBI-ID: 12302 / 生物種: Brome mosaic virus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 171.0 K / 装置: FEI VITROBOT MARK IV |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 日付 | 2013年1月10日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: DIRECT ELECTRON DE-12 (4k x 3k) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.8 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: MPSA, EMAN / 使用した粒子像数: 30000 |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)