+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5zz4 | ||||||
|---|---|---|---|---|---|---|---|
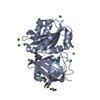
| タイトル | Crystal structure of bruton's tyrosine kinase in complex with inhibitor 2e | ||||||
 要素 要素 | Tyrosine-protein kinase BTK | ||||||
 キーワード キーワード | TRANSFERASE/TRANSFERASE INHIBITOR / protein kinase / inhibitor complex / non-covalent / TRANSFERASE-TRANSFERASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of B cell cytokine production / proteoglycan catabolic process / monocyte proliferation / positive regulation of interleukin-17A production / regulation of B cell apoptotic process / eosinophil homeostasis / positive regulation of type III hypersensitivity / B cell affinity maturation / positive regulation of synoviocyte proliferation / histamine secretion by mast cell ...regulation of B cell cytokine production / proteoglycan catabolic process / monocyte proliferation / positive regulation of interleukin-17A production / regulation of B cell apoptotic process / eosinophil homeostasis / positive regulation of type III hypersensitivity / B cell affinity maturation / positive regulation of synoviocyte proliferation / histamine secretion by mast cell / neutrophil homeostasis / cellular response to molecule of fungal origin / positive regulation of type I hypersensitivity / cellular response to interleukin-7 / MyD88 deficiency (TLR2/4) / MyD88-dependent toll-like receptor signaling pathway / IRAK4 deficiency (TLR2/4) / positive regulation of immunoglobulin production / MyD88:MAL(TIRAP) cascade initiated on plasma membrane / positive regulation of B cell differentiation / positive regulation of NLRP3 inflammasome complex assembly / phospholipase activator activity / negative regulation of interleukin-10 production / B cell activation / negative regulation of B cell proliferation / Fc-epsilon receptor signaling pathway / mesoderm development / phosphatidylinositol-3,4,5-trisphosphate binding / phospholipase binding / RHO GTPases Activate WASPs and WAVEs / positive regulation of B cell proliferation / positive regulation of phagocytosis / cell maturation / FCERI mediated Ca+2 mobilization / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / calcium-mediated signaling / apoptotic signaling pathway / FCGR3A-mediated phagocytosis / B cell receptor signaling pathway / non-specific protein-tyrosine kinase / non-membrane spanning protein tyrosine kinase activity / Regulation of actin dynamics for phagocytic cup formation / G beta:gamma signalling through BTK / cellular response to reactive oxygen species / peptidyl-tyrosine phosphorylation / positive regulation of interleukin-6 production / positive regulation of tumor necrosis factor production / G alpha (12/13) signalling events / DAP12 signaling / positive regulation of NF-kappaB transcription factor activity / T cell receptor signaling pathway / ER-Phagosome pathway / cytoplasmic vesicle / G alpha (q) signalling events / protein tyrosine kinase activity / adaptive immune response / Potential therapeutics for SARS / response to lipopolysaccharide / intracellular signal transduction / membrane raft / protein phosphorylation / innate immune response / perinuclear region of cytoplasm / ATP binding / identical protein binding / nucleus / metal ion binding / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2.9 Å 分子置換 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Kawahata, W. / Asami, T. / Irie, T. / Kiyoi, T. / Taniguchi, H. / Asamitsu, Y. / Inoue, T. / Miyake, T. / Sawa, M. | ||||||
 引用 引用 |  ジャーナル: J. Med. Chem. / 年: 2018 ジャーナル: J. Med. Chem. / 年: 2018タイトル: Design and Synthesis of Novel Amino-triazine Analogues as Selective Bruton's Tyrosine Kinase Inhibitors for Treatment of Rheumatoid Arthritis. 著者: Kawahata, W. / Asami, T. / Kiyoi, T. / Irie, T. / Taniguchi, H. / Asamitsu, Y. / Inoue, T. / Miyake, T. / Sawa, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5zz4.cif.gz 5zz4.cif.gz | 323.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5zz4.ent.gz pdb5zz4.ent.gz | 263.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5zz4.json.gz 5zz4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5zz4_validation.pdf.gz 5zz4_validation.pdf.gz | 1.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5zz4_full_validation.pdf.gz 5zz4_full_validation.pdf.gz | 1.7 MB | 表示 | |
| XML形式データ |  5zz4_validation.xml.gz 5zz4_validation.xml.gz | 60.4 KB | 表示 | |
| CIF形式データ |  5zz4_validation.cif.gz 5zz4_validation.cif.gz | 78.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zz/5zz4 https://data.pdbj.org/pub/pdb/validation_reports/zz/5zz4 ftp://data.pdbj.org/pub/pdb/validation_reports/zz/5zz4 ftp://data.pdbj.org/pub/pdb/validation_reports/zz/5zz4 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  3genS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 31178.926 Da / 分子数: 6 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: BTK, AGMX1, ATK, BPK / 発現宿主: Homo sapiens (ヒト) / 遺伝子: BTK, AGMX1, ATK, BPK / 発現宿主:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス)参照: UniProt: Q06187, non-specific protein-tyrosine kinase #2: 化合物 | ChemComp-9M3 / |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.34 Å3/Da / 溶媒含有率: 47.36 % |
|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: 0.1M imidazole(pH7.0), 0.1 M Potassium thiocyanate, 30% w/v PEG MME 2000, 4% Polypropylene glycol P 400 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-002 / 波長: 1.54 Å 回転陽極 / タイプ: RIGAKU MICROMAX-002 / 波長: 1.54 Å |
| 検出器 | タイプ: RIGAKU RAXIS IV++ / 検出器: IMAGE PLATE / 日付: 2012年7月12日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.54 Å / 相対比: 1 |
| 反射 | 解像度: 2.9→50 Å / Num. obs: 38992 / % possible obs: 100 % / 冗長度: 3.8 % / Rmerge(I) obs: 0.112 / Net I/σ(I): 5.4 |
| 反射 シェル | 解像度: 2.9→2.95 Å / Rmerge(I) obs: 0.25 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3GEN 解像度: 2.9→48.99 Å / Cor.coef. Fo:Fc: 0.923 / Cor.coef. Fo:Fc free: 0.835 / SU B: 23.697 / SU ML: 0.451 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.55 詳細: HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT U VALUES : REFINED INDIVIDUALLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 130.54 Å2 / Biso mean: 48.881 Å2 / Biso min: 16.72 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.9→48.99 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | Ens-ID: 1 / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.9→2.975 Å / Rfactor Rfree error: 0 / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj













