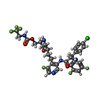
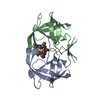
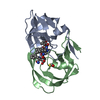
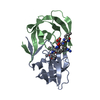
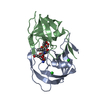
登録情報 データベース : PDB / ID : 5ivtタイトル Crystal Structure of HIV Protease complexed with [(1S)-1-[(S)-(4-chlorophenyl)-(3,5-difluorophenyl)methyl]-2-[[5-fluoro-4-[2-[(2R,5S)-5-(2,2,2-trifluoroethylcarbamoyloxymethyl)morpholin-4-ium-2-yl]ethyl]pyridin-1-ium-3-yl]amino]-2-oxo-ethyl]ammonium Protease キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 手法 / / / 解像度 : 1.15 Å データ登録者 Su, H.P. ジャーナル : Acs Med.Chem.Lett. / 年 : 2016タイトル : Discovery of MK-8718, an HIV Protease Inhibitor Containing a Novel Morpholine Aspartate Binding Group.著者: Bungard, C.J. / Williams, P.D. / Ballard, J.E. / Bennett, D.J. / Beaulieu, C. / Bahnck-Teets, C. / Carroll, S.S. / Chang, R.K. / Dubost, D.C. / Fay, J.F. / Diamond, T.L. / Greshock, T.J. / ... 著者 : Bungard, C.J. / Williams, P.D. / Ballard, J.E. / Bennett, D.J. / Beaulieu, C. / Bahnck-Teets, C. / Carroll, S.S. / Chang, R.K. / Dubost, D.C. / Fay, J.F. / Diamond, T.L. / Greshock, T.J. / Hao, L. / Holloway, M.K. / Felock, P.J. / Gesell, J.J. / Su, H.P. / Manikowski, J.J. / McKay, D.J. / Miller, M. / Min, X. / Molinaro, C. / Moradei, O.M. / Nantermet, P.G. / Nadeau, C. / Sanchez, R.I. / Satyanarayana, T. / Shipe, W.D. / Singh, S.K. / Truong, V.L. / Vijayasaradhi, S. / Wiscount, C.M. / Vacca, J.P. / Crane, S.N. / McCauley, J.A. 履歴 登録 2016年3月21日 登録サイト / 処理サイト 改定 1.0 2016年5月18日 Provider / タイプ 改定 1.1 2016年8月10日 Group / Database references改定 1.2 2024年3月6日 Group / Database references / Derived calculationsカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / diffrn_radiation_wavelength / pdbx_struct_oper_list Item / _database_2.pdbx_database_accession / _pdbx_struct_oper_list.symmetry_operation
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 1.15 Å
フーリエ合成 / 解像度: 1.15 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acs Med.Chem.Lett. / 年: 2016
ジャーナル: Acs Med.Chem.Lett. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ivt.cif.gz
5ivt.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ivt.ent.gz
pdb5ivt.ent.gz PDB形式
PDB形式 5ivt.json.gz
5ivt.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/iv/5ivt
https://data.pdbj.org/pub/pdb/validation_reports/iv/5ivt ftp://data.pdbj.org/pub/pdb/validation_reports/iv/5ivt
ftp://data.pdbj.org/pub/pdb/validation_reports/iv/5ivt リンク
リンク 集合体
集合体
 要素
要素
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 17-ID / 波長: 1 Å
/ ビームライン: 17-ID / 波長: 1 Å 解析
解析 フーリエ合成 / 解像度: 1.15→46.22 Å / Cor.coef. Fo:Fc: 0.9481 / Cor.coef. Fo:Fc free: 0.9556 / SU R Cruickshank DPI: 0.033 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.034 / SU Rfree Blow DPI: 0.034 / SU Rfree Cruickshank DPI: 0.033
フーリエ合成 / 解像度: 1.15→46.22 Å / Cor.coef. Fo:Fc: 0.9481 / Cor.coef. Fo:Fc free: 0.9556 / SU R Cruickshank DPI: 0.033 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.034 / SU Rfree Blow DPI: 0.034 / SU Rfree Cruickshank DPI: 0.033  ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj