+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5i56 | ||||||
|---|---|---|---|---|---|---|---|
| Title | Agonist-bound GluN1/GluN2A agonist binding domains with TCN201 | ||||||
 Components Components | (Glutamate receptor ionotropic, NMDA ...) x 2 | ||||||
 Keywords Keywords | TRANSPORT PROTEIN / RECEPTOR / NMDA receptor / Antagonist | ||||||
| Function / homology |  Function and homology information Function and homology informationneurotransmitter receptor transport, plasma membrane to endosome / regulation of response to alcohol / response to ammonium ion / receptor recycling / response to environmental enrichment / directional locomotion / positive regulation of Schwann cell migration / pons maturation / regulation of cell communication / EPHB-mediated forward signaling ...neurotransmitter receptor transport, plasma membrane to endosome / regulation of response to alcohol / response to ammonium ion / receptor recycling / response to environmental enrichment / directional locomotion / positive regulation of Schwann cell migration / pons maturation / regulation of cell communication / EPHB-mediated forward signaling / Assembly and cell surface presentation of NMDA receptors / response to hydrogen sulfide / auditory behavior / olfactory learning / conditioned taste aversion / dendritic branch / regulation of respiratory gaseous exchange / response to other organism / regulation of ARF protein signal transduction / protein localization to postsynaptic membrane / cellular response to magnesium ion / serotonin metabolic process / transmitter-gated monoatomic ion channel activity / positive regulation of inhibitory postsynaptic potential / response to methylmercury / suckling behavior / response to manganese ion / response to glycine / response to carbohydrate / propylene metabolic process / sleep / dendritic spine organization / cellular response to dsRNA / locomotion / regulation of NMDA receptor activity / cellular response to lipid / RAF/MAP kinase cascade / regulation of monoatomic cation transmembrane transport / Synaptic adhesion-like molecules / NMDA glutamate receptor activity / voltage-gated monoatomic cation channel activity / response to glycoside / NMDA selective glutamate receptor complex / glutamate binding / neurotransmitter receptor complex / ligand-gated sodium channel activity / response to morphine / glutamate receptor signaling pathway / regulation of axonogenesis / neuromuscular process / calcium ion transmembrane import into cytosol / regulation of dendrite morphogenesis / protein heterotetramerization / male mating behavior / regulation of synapse assembly / spinal cord development / response to amine / glycine binding / cellular response to zinc ion / startle response / parallel fiber to Purkinje cell synapse / positive regulation of reactive oxygen species biosynthetic process / dopamine metabolic process / monoatomic cation transmembrane transport / response to lithium ion / positive regulation of calcium ion transport into cytosol / cellular response to glycine / response to light stimulus / modulation of excitatory postsynaptic potential / regulation of postsynaptic membrane potential / action potential / associative learning / conditioned place preference / excitatory synapse / positive regulation of dendritic spine maintenance / monoatomic ion channel complex / social behavior / regulation of neuronal synaptic plasticity / monoatomic cation transport / glutamate receptor binding / positive regulation of protein targeting to membrane / Unblocking of NMDA receptors, glutamate binding and activation / positive regulation of excitatory postsynaptic potential / long-term memory / synaptic cleft / neuron development / phosphatase binding / prepulse inhibition / positive regulation of synaptic transmission, glutamatergic / multicellular organismal response to stress / postsynaptic density, intracellular component / response to fungicide / monoatomic cation channel activity / calcium ion homeostasis / glutamate-gated receptor activity / regulation of neuron apoptotic process / cell adhesion molecule binding / cellular response to manganese ion / glutamate-gated calcium ion channel activity / presynaptic active zone membrane Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.28 Å MOLECULAR REPLACEMENT / Resolution: 2.28 Å | ||||||
 Authors Authors | Mou, T.-C. / Sprang, S.R. / Hansen, K.B. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: Neuron / Year: 2016 Journal: Neuron / Year: 2016Title: Structural Basis for Negative Allosteric Modulation of GluN2A-Containing NMDA Receptors. Authors: Yi, F. / Mou, T.C. / Dorsett, K.N. / Volkmann, R.A. / Menniti, F.S. / Sprang, S.R. / Hansen, K.B. #1:  Journal: To Be Published Journal: To Be PublishedTitle: Structural basis for negative allosteric modulation of GluN2A-containing NMDA receptors Authors: Yi, F. / Mou, T.-C. / Dorsett, K.N. / Sprang, S.R. / Hansen, K.B. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5i56.cif.gz 5i56.cif.gz | 133.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5i56.ent.gz pdb5i56.ent.gz | 101.6 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5i56.json.gz 5i56.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/i5/5i56 https://data.pdbj.org/pub/pdb/validation_reports/i5/5i56 ftp://data.pdbj.org/pub/pdb/validation_reports/i5/5i56 ftp://data.pdbj.org/pub/pdb/validation_reports/i5/5i56 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  5i57C  5i58C  5i59C  5jtyC  4nf8S C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Glutamate receptor ionotropic, NMDA ... , 2 types, 2 molecules AB
| #1: Protein | Mass: 33340.031 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|---|
| #2: Protein | Mass: 31533.053 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
-Non-polymers , 4 types, 179 molecules 

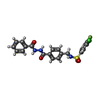




| #3: Chemical | ChemComp-GLY / |
|---|---|
| #4: Chemical | ChemComp-GLU / |
| #5: Chemical | ChemComp-67P / |
| #6: Water | ChemComp-HOH / |
-Details
| Has protein modification | Y |
|---|---|
| Sequence details | THE SEQUENCE CORRESPONDS TO THE NCBI REFERENCE SEQUENCE NP_036705.3 FOR GLUN2A. RESIDUE THR242 IN ...THE SEQUENCE CORRESPOND |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.33 Å3/Da / Density % sol: 47.63 % / Description: rod |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / Details: 0.2 M ammonium sulfate and 16-22% PEG 4000 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 19-ID / Wavelength: 0.98 Å / Beamline: 19-ID / Wavelength: 0.98 Å |
| Detector | Type: ADSC QUANTUM 315r / Detector: CCD / Date: Dec 6, 2015 |
| Radiation | Monochromator: Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.98 Å / Relative weight: 1 |
| Reflection | Resolution: 2.278→20.032 Å / Num. obs: 27522 / % possible obs: 96.7 % / Redundancy: 4.7 % / Biso Wilson estimate: 34.61 Å2 / Rsym value: 0.1 / Net I/σ(I): 10.42 |
| Reflection shell | Resolution: 2.28→2.36 Å / Redundancy: 4.2 % / Rmerge(I) obs: 0.49 / % possible all: 91.8 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4NF8 Resolution: 2.28→20.03 Å / SU ML: 0.26 / Cross valid method: FREE R-VALUE / σ(F): 1.35 / Phase error: 24.16
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.28→20.03 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj








