+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5199 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Atomic model of an infectious rotavirus particle | |||||||||
 マップデータ マップデータ | Infectious rotavirus particle. Center of the virus is at 0,0,0 and the map is a quadrant of the actual map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Rotavirus / Triple Layered Particle / Near Atomic Resolution / VP4 / VP7 / Double layered particle / de novo / Infectious / Virus | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral intermediate capsid / host cell endoplasmic reticulum lumen / host cell rough endoplasmic reticulum / permeabilization of host organelle membrane involved in viral entry into host cell / T=13 icosahedral viral capsid / T=2 icosahedral viral capsid / host cytoskeleton / viral inner capsid / viral outer capsid / viral nucleocapsid ...viral intermediate capsid / host cell endoplasmic reticulum lumen / host cell rough endoplasmic reticulum / permeabilization of host organelle membrane involved in viral entry into host cell / T=13 icosahedral viral capsid / T=2 icosahedral viral capsid / host cytoskeleton / viral inner capsid / viral outer capsid / viral nucleocapsid / host cell endoplasmic reticulum-Golgi intermediate compartment / receptor-mediated virion attachment to host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / structural molecule activity / RNA binding / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 | Rhesus Rotavirus (RRV) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Settembre EC / Chen JZ / Dormitzer PR / Grigorieff N / Harrison SC | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2011 ジャーナル: EMBO J / 年: 2011タイトル: Atomic model of an infectious rotavirus particle. 著者: Ethan C Settembre / James Z Chen / Philip R Dormitzer / Nikolaus Grigorieff / Stephen C Harrison /  要旨: Non-enveloped viruses of different types have evolved distinct mechanisms for penetrating a cellular membrane during infection. Rotavirus penetration appears to occur by a process resembling ...Non-enveloped viruses of different types have evolved distinct mechanisms for penetrating a cellular membrane during infection. Rotavirus penetration appears to occur by a process resembling enveloped-virus fusion: membrane distortion linked to conformational changes in a viral protein. Evidence for such a mechanism comes from crystallographic analyses of fragments of VP4, the rotavirus-penetration protein, and infectivity analyses of structure-based VP4 mutants. We describe here the structure of an infectious rotavirus particle determined by electron cryomicroscopy (cryoEM) and single-particle analysis at about 4.3 Å resolution. The cryoEM image reconstruction permits a nearly complete trace of the VP4 polypeptide chain, including the positions of most side chains. It shows how the two subfragments of VP4 (VP8(*) and VP5(*)) retain their association after proteolytic cleavage, reveals multiple structural roles for the β-barrel domain of VP5(*), and specifies interactions of VP4 with other capsid proteins. The virion model allows us to integrate structural and functional information into a coherent mechanism for rotavirus entry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5199.map.gz emd_5199.map.gz | 374.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5199-v30.xml emd-5199-v30.xml emd-5199.xml emd-5199.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5199_1.jpg emd_5199_1.jpg | 213.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5199 http://ftp.pdbj.org/pub/emdb/structures/EMD-5199 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5199 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5199 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5199.map.gz / 形式: CCP4 / 大きさ: 465.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5199.map.gz / 形式: CCP4 / 大きさ: 465.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Infectious rotavirus particle. Center of the virus is at 0,0,0 and the map is a quadrant of the actual map. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.23 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
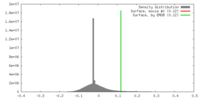
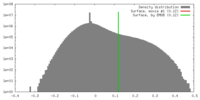
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rotavirus Triple Layered Particle
| 全体 | 名称: Rotavirus Triple Layered Particle |
|---|---|
| 要素 |
|
-超分子 #1000: Rotavirus Triple Layered Particle
| 超分子 | 名称: Rotavirus Triple Layered Particle / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: Full icosahedral Infectious Particle / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 100 MDa / 理論値: 100 MDa 手法: Calculation of Protein Mass alone, no genome included. |
-超分子 #1: Rhesus Rotavirus (RRV)
| 超分子 | 名称: Rhesus Rotavirus (RRV) / タイプ: virus / ID: 1 / Name.synonym: Rotavirus 詳細: The entire particle with genome was measured (the molecular weight is based on protein alone). 生物種: Rhesus Rotavirus (RRV) / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: Rotavirus |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| 分子量 | 実験値: 100 MDa / 理論値: 100 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: VP7 / 直径: 800 Å / T番号(三角分割数): 13 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 10 mM Tris-HCL |
| グリッド | 詳細: 200 mesh Carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 90 K / 装置: HOMEMADE PLUNGER 詳細: Vitrification instrument: manual plunger. in normal coldroom environment 手法: front blotting for 3s before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 温度 | 最低: 90 K / 最高: 90 K / 平均: 90 K |
| 詳細 | Cut-plate film holders to reduce electron back-scattering |
| 日付 | 2008年3月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7.0 µm / 実像数: 110 / 平均電子線量: 20 e/Å2 / カメラ長: 800 / 詳細: 4,187 particles in the dataset / Od range: 1.2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 56772 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)