+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EVA71 E096A native particle | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | native provirion intermediate / VIRUS | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / symbiont-mediated suppression of host gene expression ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / cytoplasmic vesicle membrane / endocytosis involved in viral entry into host cell / symbiont-mediated suppression of host gene expression / nucleoside-triphosphate phosphatase / channel activity / viral capsid / monoatomic ion transmembrane transport / RNA helicase activity / induction by virus of host autophagy / RNA-directed RNA polymerase / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / virus-mediated perturbation of host defense response / DNA-templated transcription / host cell nucleus / virion attachment to host cell / structural molecule activity / ATP hydrolysis activity / proteolysis / RNA binding / ATP binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Enterovirus A71 (エンテロウイルス) Enterovirus A71 (エンテロウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | ||||||||||||
 データ登録者 データ登録者 | Kingston NJ / Stonehouse NJ / Rowlands DJ / Hogle JM / Filman DJ / Snowden JSS | ||||||||||||
| 資金援助 |  米国, 米国,  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2024 ジャーナル: PLoS Pathog / 年: 2024タイトル: Mechanism of enterovirus VP0 maturation cleavage based on the structure of a stabilised assembly intermediate. 著者: Natalie J Kingston / Joseph S Snowden / Keith Grehan / Philippa K Hall / Eero V Hietanen / Tim C Passchier / Stephen J Polyak / David J Filman / James M Hogle / David J Rowlands / Nicola J Stonehouse /   要旨: Molecular details of genome packaging are little understood for the majority of viruses. In enteroviruses (EVs), cleavage of the structural protein VP0 into VP4 and VP2 is initiated by the ...Molecular details of genome packaging are little understood for the majority of viruses. In enteroviruses (EVs), cleavage of the structural protein VP0 into VP4 and VP2 is initiated by the incorporation of RNA into the assembling virion and is essential for infectivity. We have applied a combination of bioinformatic, molecular and structural approaches to generate the first high-resolution structure of an intermediate in the assembly pathway, termed a provirion, which contains RNA and intact VP0. We have demonstrated an essential role of VP0 E096 in VP0 cleavage independent of RNA encapsidation and generated a new model of capsid maturation, supported by bioinformatic analysis. This provides a molecular basis for RNA-dependence, where RNA induces conformational changes required for VP0 maturation, but that RNA packaging itself is not sufficient to induce maturation. These data have implications for understanding production of infectious virions and potential relevance for future vaccine and antiviral drug design. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50221.map.gz emd_50221.map.gz | 169.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50221-v30.xml emd-50221-v30.xml emd-50221.xml emd-50221.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_50221.png emd_50221.png | 128.6 KB | ||
| マスクデータ |  emd_50221_msk_1.map emd_50221_msk_1.map | 282.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50221.cif.gz emd-50221.cif.gz | 6.2 KB | ||
| その他 |  emd_50221_half_map_1.map.gz emd_50221_half_map_1.map.gz emd_50221_half_map_2.map.gz emd_50221_half_map_2.map.gz | 224.8 MB 224.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50221 http://ftp.pdbj.org/pub/emdb/structures/EMD-50221 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50221 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50221 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50221_validation.pdf.gz emd_50221_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50221_full_validation.pdf.gz emd_50221_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_50221_validation.xml.gz emd_50221_validation.xml.gz | 16.8 KB | 表示 | |
| CIF形式データ |  emd_50221_validation.cif.gz emd_50221_validation.cif.gz | 19.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50221 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50221 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50221 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50221 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9f6aM M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50221.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50221.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||||||||||||||||||
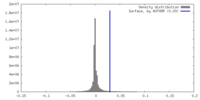
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50221_msk_1.map emd_50221_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
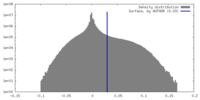
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterovirus A71
| 全体 | 名称:   Enterovirus A71 (エンテロウイルス) Enterovirus A71 (エンテロウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Enterovirus A71
| 超分子 | 名称: Enterovirus A71 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Mutant virus recovered from in vitro transcribed RNA NCBI-ID: 39054 / 生物種: Enterovirus A71 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|
-分子 #1: VP0
| 分子 | 名称: VP0 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterovirus A71 (エンテロウイルス) Enterovirus A71 (エンテロウイルス) |
| 分子量 | 理論値: 35.19523 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGSQVSTQRS GSHENSNSAT EGSTINYTTI NYYKDSYAAT AGKQSLKQDP DKFANPVKDV FTEMAAPLKS PSAEACGYSD RVAQLTIGN STITTQAAAN IIVGYGEWPS YCSDDDATAV DKPTRPDVSV NRFYTLDTKL WEKSSKGWYW KFPDVLTETG V FGQNAQFH ...文字列: MGSQVSTQRS GSHENSNSAT EGSTINYTTI NYYKDSYAAT AGKQSLKQDP DKFANPVKDV FTEMAAPLKS PSAEACGYSD RVAQLTIGN STITTQAAAN IIVGYGEWPS YCSDDDATAV DKPTRPDVSV NRFYTLDTKL WEKSSKGWYW KFPDVLTETG V FGQNAQFH YLYRSGFCIH VQCNASKFHQ GALLVAILPE YVIGTVAGGT GTEDSHPPYI QTQPGADGFE LQHPYVLDAG IP ISQLTVC PHQWINLRTN NCATIIVPYM NTLPFDSALN HCNFGLLVVP ISPLDFDQGA TPVIPITITL APMCSEFAGL RQA VTQ UniProtKB: Genome polyprotein |
-分子 #2: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterovirus A71 (エンテロウイルス) Enterovirus A71 (エンテロウイルス) |
| 分子量 | 理論値: 26.540332 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GFPTELKPGT NQFLTTDDGV SAPILPNFHP TPCIHIPGEV RNLLELCQVE TILEVNNVPT NATSLMERLR FPVSAQAGKG ELCAVFRAD PGRDGPWQST MLGQLCGYYT QWSGSLEVTF MFTGSFMATG KMLIAYTPPG GPLPKDRATA MLGTHVIWDF G LQSSVTLV ...文字列: GFPTELKPGT NQFLTTDDGV SAPILPNFHP TPCIHIPGEV RNLLELCQVE TILEVNNVPT NATSLMERLR FPVSAQAGKG ELCAVFRAD PGRDGPWQST MLGQLCGYYT QWSGSLEVTF MFTGSFMATG KMLIAYTPPG GPLPKDRATA MLGTHVIWDF G LQSSVTLV IPWISNTHYR AHARDGVFDY YTTGLVSIWY QTNYVVPIGA PNTAYIIALA AAQKNFTMKL CKDTSHILQT AS IQ UniProtKB: Genome polyprotein |
-分子 #3: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: picornain 2A |
|---|---|
| 由来(天然) | 生物種:   Enterovirus A71 (エンテロウイルス) Enterovirus A71 (エンテロウイルス) |
| 分子量 | 理論値: 32.781805 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GDRVADMIES SIGNSVSRAL TQALPAPTGQ NTQVSSHRLD TGEVPALQAA EIGASSNTSD ESMIETRCVL NSHSTAETTL DSFFSRAGL VGEIDLPLEG TTNPNGYANW DIDITGYAQM RRKVELFTYM RFDAEFTFVA CTPTGQVVPQ LLQYMFVPPG A PKPESRES ...文字列: GDRVADMIES SIGNSVSRAL TQALPAPTGQ NTQVSSHRLD TGEVPALQAA EIGASSNTSD ESMIETRCVL NSHSTAETTL DSFFSRAGL VGEIDLPLEG TTNPNGYANW DIDITGYAQM RRKVELFTYM RFDAEFTFVA CTPTGQVVPQ LLQYMFVPPG A PKPESRES LAWQTATNPS VFVKLTDPPA QVSVPFMSPA SAYQWFYDGY PTFGEHKQEK DLEYGACPNN MMGTFSVRTV GS SKSKYAL VVRIYMRMKH VRAWIPRPMR NQNYLFKANP NYAGDSIKPT GTSRNAITTL UniProtKB: Genome polyprotein |
-分子 #4: SPHINGOSINE
| 分子 | 名称: SPHINGOSINE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: SPH |
|---|---|
| 分子量 | 理論値: 299.492 Da |
| Chemical component information |  ChemComp-SPH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 31.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.3 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 1182 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)