+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4uvt | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of human tankyrase 2 in complex with 5-amino-4- methyl-1,2-dihydroisoquinolin-1-one | ||||||
 要素 要素 | TANKYRASE-2 | ||||||
 キーワード キーワード | TRANSFERASE / PROTEIN-LIGAND COMPLEX / DIPHTHERIA TOXIN LIKE FOLD / ADP- RIBOSYLATION / TRANSFERASE-TRANSFERASE INHIBITOR COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報XAV939 stabilizes AXIN / NAD+ ADP-ribosyltransferase / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / protein poly-ADP-ribosylation / pericentriolar material / NAD+-protein ADP-ribosyltransferase activity / positive regulation of telomere capping / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの ...XAV939 stabilizes AXIN / NAD+ ADP-ribosyltransferase / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / protein poly-ADP-ribosylation / pericentriolar material / NAD+-protein ADP-ribosyltransferase activity / positive regulation of telomere capping / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / NAD+ ADP-ribosyltransferase activity / positive regulation of telomere maintenance via telomerase / nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN / Wnt signaling pathway / Regulation of PTEN stability and activity / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / nuclear envelope / chromosome, telomeric region / Ub-specific processing proteases / Golgi membrane / perinuclear region of cytoplasm / enzyme binding / nucleus / metal ion binding / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.95 Å 分子置換 / 解像度: 1.95 Å | ||||||
 データ登録者 データ登録者 | Narwal, M. / Haikarainen, T. / Lehtio, L. | ||||||
 引用 引用 |  ジャーナル: Bioorg.Med.Chem. / 年: 2015 ジャーナル: Bioorg.Med.Chem. / 年: 2015タイトル: Exploration of the Nicotinamide-Binding Site of the Tankyrases, Identifying 3-Arylisoquinolin-1-Ones as Potent and Selective Inhibitors in Vitro. 著者: Paine, H.A. / Nathubhai, A. / Woon, E.C.Y. / Sunderland, P.T. / Wood, P.J. / Mahon, M.F. / Lloyd, M.D. / Thompson, A.S. / Haikarainen, T. / Narwal, M. / Lehtio, L. / Threadgill, M.D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4uvt.cif.gz 4uvt.cif.gz | 108.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4uvt.ent.gz pdb4uvt.ent.gz | 82.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4uvt.json.gz 4uvt.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4uvt_validation.pdf.gz 4uvt_validation.pdf.gz | 490.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4uvt_full_validation.pdf.gz 4uvt_full_validation.pdf.gz | 495.2 KB | 表示 | |
| XML形式データ |  4uvt_validation.xml.gz 4uvt_validation.xml.gz | 22.7 KB | 表示 | |
| CIF形式データ |  4uvt_validation.cif.gz 4uvt_validation.cif.gz | 32.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uv/4uvt https://data.pdbj.org/pub/pdb/validation_reports/uv/4uvt ftp://data.pdbj.org/pub/pdb/validation_reports/uv/4uvt ftp://data.pdbj.org/pub/pdb/validation_reports/uv/4uvt | HTTPS FTP |
-関連構造データ
| 関連構造データ |  4uvlC  4uvnC  4uvoC  4uvpC  4uvsC  4uvuC  4uvvC  4uvwC  4uvxC  4uvyC  4uvzC  3kr7S C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AC
| #1: タンパク質 | 分子量: 27299.764 Da / 分子数: 2 / 断片: C-TERMINAL FRAGMENT, UNP RESIDUES 946-1162 / 由来タイプ: 組換発現 / 由来: (組換発現)  HOMO SAPIENS (ヒト) / プラスミド: PNIC28-BSA4 / 発現宿主: HOMO SAPIENS (ヒト) / プラスミド: PNIC28-BSA4 / 発現宿主:  |
|---|
-非ポリマー , 6種, 379分子 



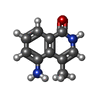






| #2: 化合物 | | #3: 化合物 | ChemComp-SO4 / #4: 化合物 | ChemComp-GOL / | #5: 化合物 | #6: 化合物 | #7: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 49 % / 解説: NONE |
|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: 0.2 M LISO4, 0.1 M TRIS HCL, 22% PEG3350, PH 8.5, VAPOR DIFFUSION, SITTING DROP, TEMPERATURE 277K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID23-1 / 波長: 1.07227 / ビームライン: ID23-1 / 波長: 1.07227 |
| 検出器 | タイプ: ADSC CCD / 検出器: CCD / 日付: 2011年4月8日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.07227 Å / 相対比: 1 |
| 反射 | 解像度: 1.95→50 Å / Num. obs: 38605 / % possible obs: 99.3 % / Observed criterion σ(I): -3 / 冗長度: 7.2 % / Rmerge(I) obs: 0.07 / Net I/σ(I): 18.62 |
| 反射 シェル | 解像度: 1.95→2 Å / 冗長度: 7.4 % / Rmerge(I) obs: 0.6 / Mean I/σ(I) obs: 3.37 / % possible all: 98.9 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 3KR7 解像度: 1.95→48.81 Å / Cor.coef. Fo:Fc: 0.962 / Cor.coef. Fo:Fc free: 0.939 / SU B: 2.974 / SU ML: 0.087 / 交差検証法: THROUGHOUT / ESU R: 0.141 / ESU R Free: 0.136 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 22.054 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.95→48.81 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー






















 PDBj
PDBj







