+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4990 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
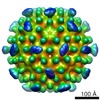
| タイトル | T=4 MS2 Virus-like-particle | |||||||||
 マップデータ マップデータ | 0.04 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MS2 / T=4 / BACTERIOPHAGE / VLP / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of viral translation / T=3 icosahedral viral capsid / regulation of translation / structural molecule activity / RNA binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Escherichia phage MS2 (ファージ) / Escherichia phage MS2 (ファージ) /  Escherichia virus MS2 (ウイルス) Escherichia virus MS2 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.0 Å | |||||||||
 データ登録者 データ登録者 | de Martin Garrido N / Ramlaul K | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Microbiol / 年: 2020 ジャーナル: Mol Microbiol / 年: 2020タイトル: Bacteriophage MS2 displays unreported capsid variability assembling T = 4 and mixed capsids. 著者: Natàlia de Martín Garrido / Michael A Crone / Kailash Ramlaul / Paul A Simpson / Paul S Freemont / Christopher H S Aylett /  要旨: Bacteriophage MS2 is a positive-sense, single-stranded RNA virus encapsulated in an asymmetric T = 3 pseudo-icosahedral capsid. It infects Escherichia coli through the F-pilus, in which it binds ...Bacteriophage MS2 is a positive-sense, single-stranded RNA virus encapsulated in an asymmetric T = 3 pseudo-icosahedral capsid. It infects Escherichia coli through the F-pilus, in which it binds through a maturation protein incorporated into its capsid. Cryogenic electron microscopy has previously shown that its genome is highly ordered within virions, and that it regulates the assembly process of the capsid. In this study, we have assembled recombinant MS2 capsids with non-genomic RNA containing the capsid incorporation sequence, and investigated the structures formed, revealing that T = 3, T = 4 and mixed capsids between these two triangulation numbers are generated, and resolving structures of T = 3 and T = 4 capsids to 4 Å and 6 Å respectively. We conclude that the basic MS2 capsid can form a mix of T = 3 and T = 4 structures, supporting a role for the ordered genome in favouring the formation of functional T = 3 virions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4990.map.gz emd_4990.map.gz | 190.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4990-v30.xml emd-4990-v30.xml emd-4990.xml emd-4990.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4990.png emd_4990.png | 180.4 KB | ||
| Filedesc metadata |  emd-4990.cif.gz emd-4990.cif.gz | 5.9 KB | ||
| その他 |  emd_4990_half_map_1.map.gz emd_4990_half_map_1.map.gz emd_4990_half_map_2.map.gz emd_4990_half_map_2.map.gz | 165.9 MB 168.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4990 http://ftp.pdbj.org/pub/emdb/structures/EMD-4990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4990 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4990 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4990_validation.pdf.gz emd_4990_validation.pdf.gz | 874 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4990_full_validation.pdf.gz emd_4990_full_validation.pdf.gz | 873.6 KB | 表示 | |
| XML形式データ |  emd_4990_validation.xml.gz emd_4990_validation.xml.gz | 15.5 KB | 表示 | |
| CIF形式データ |  emd_4990_validation.cif.gz emd_4990_validation.cif.gz | 18.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4990 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4990 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4990 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4990 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4990.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4990.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 0.04 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0277 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: MS2 bacteriophage T=4 virus-like particle half-map 1
| ファイル | emd_4990_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MS2 bacteriophage T=4 virus-like particle half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
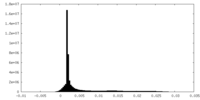
| 密度ヒストグラム |
-ハーフマップ: MS2 bacteriophage T=4 virus-like particle half-map 2
| ファイル | emd_4990_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MS2 bacteriophage T=4 virus-like particle half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
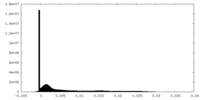
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia virus MS2
| 全体 | 名称:  Escherichia virus MS2 (ウイルス) Escherichia virus MS2 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia virus MS2
| 超分子 | 名称: Escherichia virus MS2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 329852 / 生物種: Escherichia virus MS2 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage MS2 (ファージ) Escherichia phage MS2 (ファージ) |
| 分子量 | 理論値: 13.869659 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASNFTQFVL VDNGGTGDVT VAPSNFANGV AEWISSNSRS QAYKVTCSVR QSSAQNRKYT IKVEVPKVAT QTVGGVELPV AAWRSYLNM ELTIPIFATN SDCELIVKAM QGLLKDGNPI PSAIAANSGI Y UniProtKB: Capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 10 mM Tris-HCl pH 7.5, 1 mM EDTA, 100 mM NaCl |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 278 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON I (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 4 / 実像数: 2129 / 平均露光時間: 4.0 sec. / 平均電子線量: 80.0 e/Å2 詳細: Frames were obtained with a beam-splitter Images were recorded at both 100kx and 150kx and resampled in Fourier space to 100kx for refinement |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): -2.0 µm / 最小 デフォーカス(公称値): -0.3 µm / 倍率(公称値): 100000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)