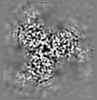
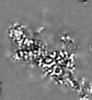
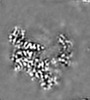
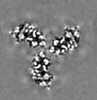
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the HIV-1 WITO IDL Env trimer in complex with PGT122 Fab | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CD4 / HIV-1 / SOSIP / Vaccine / gp120 / gp41 / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virus-mediated perturbation of host defense response => GO:0019049 / : / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / membrane => GO:0016020 / viral protein processing / fusion of virus membrane with host plasma membrane ...virus-mediated perturbation of host defense response => GO:0019049 / : / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / membrane => GO:0016020 / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / structural molecule activity / virion membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2024 ジャーナル: Cell Rep / 年: 2024タイトル: Design of soluble HIV-1 envelope trimers free of covalent gp120-gp41 bonds with prevalent native-like conformation. 著者: Peng Zhang / Jason Gorman / Yaroslav Tsybovsky / Maolin Lu / Qingbo Liu / Vinay Gopan / Mamta Singh / Yin Lin / Huiyi Miao / Yuna Seo / Alice Kwon / Adam S Olia / Gwo-Yu Chuang / Hui Geng / ...著者: Peng Zhang / Jason Gorman / Yaroslav Tsybovsky / Maolin Lu / Qingbo Liu / Vinay Gopan / Mamta Singh / Yin Lin / Huiyi Miao / Yuna Seo / Alice Kwon / Adam S Olia / Gwo-Yu Chuang / Hui Geng / Yen-Ting Lai / Tongqing Zhou / John R Mascola / Walther Mothes / Peter D Kwong / Paolo Lusso /   要旨: Soluble HIV-1 envelope (Env) trimers may serve as effective vaccine immunogens. The widely utilized SOSIP trimers have been paramount for structural studies, but the disulfide bond they feature ...Soluble HIV-1 envelope (Env) trimers may serve as effective vaccine immunogens. The widely utilized SOSIP trimers have been paramount for structural studies, but the disulfide bond they feature between gp120 and gp41 constrains intersubunit mobility and may alter antigenicity. Here, we report an alternative strategy to generate stabilized soluble Env trimers free of covalent gp120-gp41 bonds. Stabilization was achieved by introducing an intrasubunit disulfide bond between the inner and outer domains of gp120, defined as interdomain lock (IDL). Correctly folded IDL trimers displaying a native-like antigenic profile were produced for HIV-1 Envs of different clades. Importantly, the IDL design abrogated CD4 binding while not affecting recognition by potent neutralizing antibodies to the CD4-binding site. By cryoelectron microscopy, IDL trimers were shown to adopt a closed prefusion configuration, while single-molecule fluorescence resonance energy transfer documented a high prevalence of native-like conformation. Thus, IDL trimers may be promising candidates as vaccine immunogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44491.map.gz emd_44491.map.gz | 156 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44491-v30.xml emd-44491-v30.xml emd-44491.xml emd-44491.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_44491.png emd_44491.png | 87.1 KB | ||
| マスクデータ |  emd_44491_msk_1.map emd_44491_msk_1.map | 166.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44491.cif.gz emd-44491.cif.gz | 7.2 KB | ||
| その他 |  emd_44491_additional_1.map.gz emd_44491_additional_1.map.gz emd_44491_additional_2.map.gz emd_44491_additional_2.map.gz emd_44491_half_map_1.map.gz emd_44491_half_map_1.map.gz emd_44491_half_map_2.map.gz emd_44491_half_map_2.map.gz | 32.7 MB 8 MB 154.4 MB 154.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44491 http://ftp.pdbj.org/pub/emdb/structures/EMD-44491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44491 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44491_validation.pdf.gz emd_44491_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44491_full_validation.pdf.gz emd_44491_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_44491_validation.xml.gz emd_44491_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_44491_validation.cif.gz emd_44491_validation.cif.gz | 17.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44491 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44491 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44491 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44491 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44491.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44491.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||
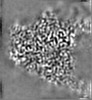
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0733 Å | ||||||||||||||||||||||||||||||||||||
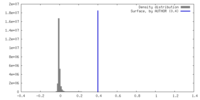
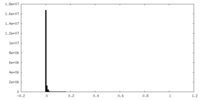
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_44491_msk_1.map emd_44491_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
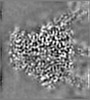
| 投影像・断面図 |
| ||||||||||||
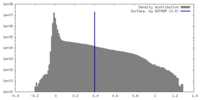
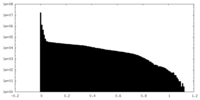
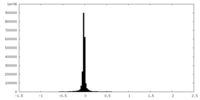
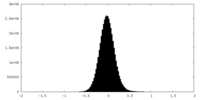
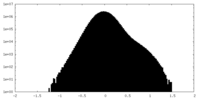
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_44491_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
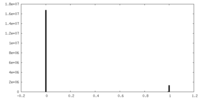
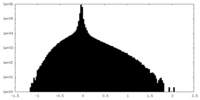
| 密度ヒストグラム |
-追加マップ: resolve map
| ファイル | emd_44491_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | resolve map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
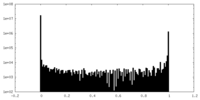
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_44491_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
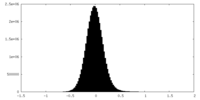
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_44491_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
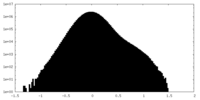
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM Structure of the HIV-1 WITO IDL Env trimer in complex wit...
| 全体 | 名称: Cryo-EM Structure of the HIV-1 WITO IDL Env trimer in complex with PGT122 Fab |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM Structure of the HIV-1 WITO IDL Env trimer in complex wit...
| 超分子 | 名称: Cryo-EM Structure of the HIV-1 WITO IDL Env trimer in complex with PGT122 Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.381771 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVTLGAVFLG FLGAAGSTMG AASLTLTVQA RLLLSGIVQQ QSNLLRAPEA QQHMLQLTVW GIKQLQARVL AIERYLKDQQ LLGIWGCSG KLICTTTVPW NTSWSNKSYD YIWNNMTWMQ WEREIDNYTG FIYTLIEESQ NQQEKNELEL LELD UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 53.827133 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AEQLWVTVYY GVPVWREANT TLFCASDAKA YDTEVHNVWA THACVPTDPN PQEVVMGNVT EDFNMWKNNM VEQMHEDIIS LWCQSLKPC VKLTPLCVTL HCTNVTISST NGSTANVTMR EEMKNCSFNT TTVIRDKIQK EYALFYKLDI VPIEGKNTNT S YRLINCNT ...文字列: AEQLWVTVYY GVPVWREANT TLFCASDAKA YDTEVHNVWA THACVPTDPN PQEVVMGNVT EDFNMWKNNM VEQMHEDIIS LWCQSLKPC VKLTPLCVTL HCTNVTISST NGSTANVTMR EEMKNCSFNT TTVIRDKIQK EYALFYKLDI VPIEGKNTNT S YRLINCNT SVITQACPKV SFEPIPIHYC APAGFAILKC NNKTFNGKGP CRNVSTVQCT HGIKPVVSTQ LLLNGSLAEE DI IIRSENF TNNGKNIIVQ LKEPVKINCT RPGNNTRRSI NIGPGRAFYA TGAIIGDIRK AHCNISTEQW NNTLTQIVDK LRE QFGN(UNK)K TIIFNQSSGG DPEVVMHTFN CGGEFFYCNS TQLFNSTWFN NGTSTWNSTA DNITLPCRIK QVINMWQEVG GCGAMYAPP IRGQIDCSSN ITGLILTRDG GSNSSQNETF RPGGGNMKDN WRSELYKYKV VKIEPLGIAP TRAKRRVVQR R RRRR UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: PGT122 heavy chain
| 分子 | 名称: PGT122 heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.838731 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVHLQESGPG LVKPSETLSL TCNVSGTLVR DNYWSWIRQP LGKQPEWIGY VHDSGDTNYN PSLKSRVHLS LDKSKNLVSL RLTGVTAAD SAIYYCATTK HGRRIYGVVA FKEWFTYFYM DVWGKGTSVT VSS |
-分子 #4: PGT122 light chain
| 分子 | 名称: PGT122 light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.423534 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TFVSVAPGQT ARITCGEESL GSRSVIWYQQ RPGQAPSLII YNNNDRPSGI PDRFSGSPGS TFGTTATLTI TSVEAGDEAD YYCHIWDSR RPTNWVFGEG TTLIVL |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 45 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #9: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 9 / コピー数: 1 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS |
| グリッド | モデル: C-flat-1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 70.77 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.7 µm / 最小 デフォーカス(公称値): 0.1 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-9bf6: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)