| 登録情報 | データベース: EMDB / ID: EMD-44425
|
|---|
| タイトル | HCN1 M305L with propofol |
|---|
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: HCN1
- タンパク質・ペプチド: Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 1
- リガンド: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
- リガンド: 2,6-BIS(1-METHYLETHYL)PHENOL
|
|---|
 キーワード キーワード | Inhibitor / complex / membrane protein / nanodisc / transport protein |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
intracellular cAMP-activated cation channel activity involved in regulation of presynaptic membrane potential / HCN channels / general adaptation syndrome, behavioral process / HCN channel complex / retinal cone cell development / regulation of membrane depolarization / intracellularly cAMP-activated cation channel activity / apical protein localization / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity ...intracellular cAMP-activated cation channel activity involved in regulation of presynaptic membrane potential / HCN channels / general adaptation syndrome, behavioral process / HCN channel complex / retinal cone cell development / regulation of membrane depolarization / intracellularly cAMP-activated cation channel activity / apical protein localization / voltage-gated monoatomic cation channel activity / voltage-gated sodium channel activity / voltage-gated potassium channel activity / potassium channel activity / sodium ion transmembrane transport / neuronal action potential / cAMP binding / cellular response to cAMP / presynaptic active zone membrane / potassium ion transmembrane transport / regulation of membrane potential / postsynaptic membrane / protein homotetramerization / axon / glutamatergic synapse / dendrite / identical protein binding / plasma membrane類似検索 - 分子機能 Ion transport N-terminal / Ion transport protein N-terminal / Potassium channel, voltage-dependent, EAG/ELK/ERG / Cyclic nucleotide-binding domain signature 1. / Cyclic nucleotide-binding, conserved site / Cyclic nucleotide-monophosphate binding domain / Cyclic nucleotide-binding domain / cAMP/cGMP binding motif profile. / Cyclic nucleotide-binding domain / Cyclic nucleotide-binding domain superfamily ...Ion transport N-terminal / Ion transport protein N-terminal / Potassium channel, voltage-dependent, EAG/ELK/ERG / Cyclic nucleotide-binding domain signature 1. / Cyclic nucleotide-binding, conserved site / Cyclic nucleotide-monophosphate binding domain / Cyclic nucleotide-binding domain / cAMP/cGMP binding motif profile. / Cyclic nucleotide-binding domain / Cyclic nucleotide-binding domain superfamily / RmlC-like jelly roll fold / Ion transport domain / Ion transport protein類似検索 - ドメイン・相同性 Potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channel 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
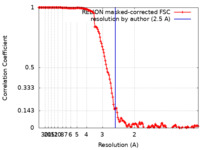
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å |
|---|
 データ登録者 データ登録者 | Kim ED / Nimigean CM |
|---|
| 資金援助 |  米国, 2件 米国, 2件 | Organization | Grant number | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM124451 |  米国 米国 | | National Institutes of Health/National Institute of Neurological Disorders and Stroke (NIH/NINDS) | NS137561 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024
タイトル: Propofol rescues voltage-dependent gating of HCN1 channel epilepsy mutants
著者: Kim ED / Wu X / Lee S / Tibbs GR / Cunningham KP / Di Zanni E / Perez ME / Goldstein PA / Accardi A / Larsson HP / Nimigean CM |
|---|
| 履歴 | | 登録 | 2024年4月7日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2024年7月31日 | - |
|---|
| マップ公開 | 2024年7月31日 | - |
|---|
| 更新 | 2024年7月31日 | - |
|---|
| 現状 | 2024年7月31日 | 処理サイト: RCSB / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
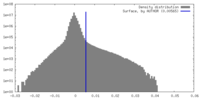
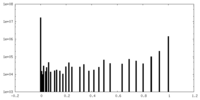
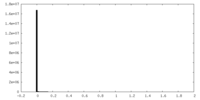
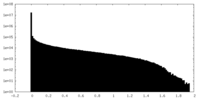
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Nature / 年: 2024
ジャーナル: Nature / 年: 2024 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_44425.map.gz
emd_44425.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-44425-v30.xml
emd-44425-v30.xml emd-44425.xml
emd-44425.xml EMDBヘッダ
EMDBヘッダ emd_44425_fsc.xml
emd_44425_fsc.xml FSCデータファイル
FSCデータファイル emd_44425.png
emd_44425.png emd_44425_msk_1.map
emd_44425_msk_1.map マスクマップ
マスクマップ emd-44425.cif.gz
emd-44425.cif.gz emd_44425_additional_1.map.gz
emd_44425_additional_1.map.gz emd_44425_half_map_1.map.gz
emd_44425_half_map_1.map.gz emd_44425_half_map_2.map.gz
emd_44425_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-44425
http://ftp.pdbj.org/pub/emdb/structures/EMD-44425 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44425
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44425 emd_44425_validation.pdf.gz
emd_44425_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_44425_full_validation.pdf.gz
emd_44425_full_validation.pdf.gz emd_44425_validation.xml.gz
emd_44425_validation.xml.gz emd_44425_validation.cif.gz
emd_44425_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44425
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44425 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44425
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44425 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_44425.map.gz / 形式: CCP4 / 大きさ: 294.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_44425.map.gz / 形式: CCP4 / 大きさ: 294.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_44425_msk_1.map
emd_44425_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)