+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A designed tetrahedral protein scaffold - DARP14 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | scaffold / tetrahedral / darpin / symmetrical / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Suder DS / Gonen S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2024 ジャーナル: Int J Mol Sci / 年: 2024タイトル: Mitigating the Blurring Effect of CryoEM Averaging on a Flexible and Highly Symmetric Protein Complex through Sub-Particle Reconstruction. 著者: Diana S Suder / Shane Gonen /  要旨: Many macromolecules are inherently flexible as a feature of their structure and function. During single-particle CryoEM processing, flexible protein regions can be detrimental to high-resolution ...Many macromolecules are inherently flexible as a feature of their structure and function. During single-particle CryoEM processing, flexible protein regions can be detrimental to high-resolution reconstruction as signals from thousands of particles are averaged together. This "blurring" effect can be difficult to overcome and is possibly more pronounced when averaging highly symmetric complexes. Approaches to mitigating flexibility during CryoEM processing are becoming increasingly critical as the technique advances and is applied to more dynamic proteins and complexes. Here, we detail the use of sub-particle averaging and signal subtraction techniques to precisely target and resolve flexible DARPin protein attachments on a designed tetrahedrally symmetric protein scaffold called DARP14. Particles are first aligned as full complexes, and then the symmetry is reduced by alignment and focused refinement of the constituent subunits. The final reconstructions we obtained were vastly improved over the fully symmetric reconstructions, with observable secondary structure and side-chain placement. Additionally, we were also able to reconstruct the core region of the scaffold to 2.7 Å. The data processing protocol outlined here is applicable to other dynamic and symmetric protein complexes, and our improved maps could allow for new structure-guided variant designs of DARP14. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43157.map.gz emd_43157.map.gz | 9.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43157-v30.xml emd-43157-v30.xml emd-43157.xml emd-43157.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43157_fsc.xml emd_43157_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43157.png emd_43157.png | 94.4 KB | ||
| Filedesc metadata |  emd-43157.cif.gz emd-43157.cif.gz | 5.3 KB | ||
| その他 |  emd_43157_half_map_1.map.gz emd_43157_half_map_1.map.gz emd_43157_half_map_2.map.gz emd_43157_half_map_2.map.gz | 77.4 MB 77.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43157 http://ftp.pdbj.org/pub/emdb/structures/EMD-43157 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43157 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43157 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43157_validation.pdf.gz emd_43157_validation.pdf.gz | 700 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43157_full_validation.pdf.gz emd_43157_full_validation.pdf.gz | 699.5 KB | 表示 | |
| XML形式データ |  emd_43157_validation.xml.gz emd_43157_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_43157_validation.cif.gz emd_43157_validation.cif.gz | 23.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43157 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43157 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43157 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43157 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43157.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43157.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.9825 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
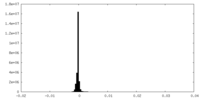
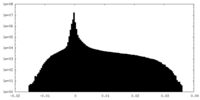
-ハーフマップ: #1
| ファイル | emd_43157_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
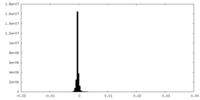
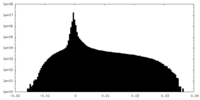
-ハーフマップ: #2
| ファイル | emd_43157_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A12B12 tetrahedral complex of a designed protein scaffold called ...
| 全体 | 名称: A12B12 tetrahedral complex of a designed protein scaffold called DARP14 |
|---|---|
| 要素 |
|
-超分子 #1: A12B12 tetrahedral complex of a designed protein scaffold called ...
| 超分子 | 名称: A12B12 tetrahedral complex of a designed protein scaffold called DARP14 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #2: Subunit A in a designed protein scaffold called DARP14
| 超分子 | 名称: Subunit A in a designed protein scaffold called DARP14 タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #3: Subunit B in a designed protein scaffold called DARP14
| 超分子 | 名称: Subunit B in a designed protein scaffold called DARP14 タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Subunit A
| 分子 | 名称: Subunit A / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO / EC番号: corrinoid adenosyltransferase |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.775438 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KDSPIIEANG TLDELTSFIG EAKHYVDEEM KGILEEIQND IYKIMGEIGS KGKIEGISEE RIAWLLKLIL RYMEMVNLKS FVLPGGTLE SAKLDVCRTI ARRALRKVLT VTREFGIGAE AAAYLLALSD LLFLLARVIE IE |
-分子 #2: Subunit B
| 分子 | 名称: Subunit B / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 12.630332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PHLVIEATAN LRLETSPGEL LEQANKALFA SGQFGEADIK SRFVTLEAYR QGTAAVERAY LHACLSILDG RDIATRTLLG ASLCAVLAE AVAGGGEEGV QVSVEVREME RLSYAKRVV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Phenix real space refine |
|---|---|
| 精密化 | 空間: REAL |
| 得られたモデル |  PDB-8vdz: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X