+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 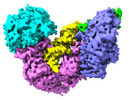 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Spo11 core complex with gapped DNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Spo11 / Rec102 / Rec104 / Ski8 / DNA binding / Cross over. / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報meiotic DNA double-strand break processing / Ski complex / mRNA decay by 3' to 5' exoribonuclease / protein-DNA complex assembly / meiotic DNA double-strand break formation / nuclear-transcribed mRNA catabolic process, 3'-5' exonucleolytic nonsense-mediated decay / Cdc73/Paf1 complex / nuclear-transcribed mRNA catabolic process, non-stop decay / : / DNA end binding ...meiotic DNA double-strand break processing / Ski complex / mRNA decay by 3' to 5' exoribonuclease / protein-DNA complex assembly / meiotic DNA double-strand break formation / nuclear-transcribed mRNA catabolic process, 3'-5' exonucleolytic nonsense-mediated decay / Cdc73/Paf1 complex / nuclear-transcribed mRNA catabolic process, non-stop decay / : / DNA end binding / synaptonemal complex assembly / homologous chromosome pairing at meiosis / reciprocal meiotic recombination / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase (ATP-hydrolysing) / nuclear chromosome / sporulation resulting in formation of a cellular spore / mRNA catabolic process / condensed nuclear chromosome / protein-containing complex assembly / defense response to virus / chromatin binding / DNA binding / nucleoplasm / ATP binding / nucleus / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Yu Y / Patel DJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of the yeast Spo11 core complex bound to DNA 著者: Yu Y / Wang JC / Liu KX / Zheng Z / Patel DJ / Keeney S | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
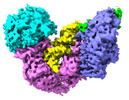
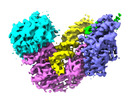
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42497.map.gz emd_42497.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42497-v30.xml emd-42497-v30.xml emd-42497.xml emd-42497.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_42497.png emd_42497.png | 156.6 KB | ||
| Filedesc metadata |  emd-42497.cif.gz emd-42497.cif.gz | 6.8 KB | ||
| その他 |  emd_42497_half_map_1.map.gz emd_42497_half_map_1.map.gz emd_42497_half_map_2.map.gz emd_42497_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42497 http://ftp.pdbj.org/pub/emdb/structures/EMD-42497 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42497 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42497 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42497_validation.pdf.gz emd_42497_validation.pdf.gz | 774.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42497_full_validation.pdf.gz emd_42497_full_validation.pdf.gz | 774.1 KB | 表示 | |
| XML形式データ |  emd_42497_validation.xml.gz emd_42497_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_42497_validation.cif.gz emd_42497_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42497 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42497 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42497 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42497 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8urqMC  8uruC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42497.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42497.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.064 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map A
| ファイル | emd_42497_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
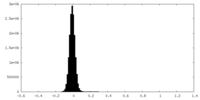
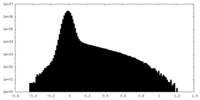
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map B
| ファイル | emd_42497_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
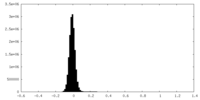
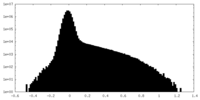
| 注釈 | Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Spo11 core complex bound to gapped DNA
| 全体 | 名称: Spo11 core complex bound to gapped DNA |
|---|---|
| 要素 |
|
-超分子 #1: Spo11 core complex bound to gapped DNA
| 超分子 | 名称: Spo11 core complex bound to gapped DNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 140 KDa |
-分子 #1: Meiotic recombination protein REC104
| 分子 | 名称: Meiotic recombination protein REC104 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 20.763146 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSIEEEDTNK ITCTQDFLHQ YFVTERVSIQ FGLNNKTVKR INKDEFDKAV NCIMSWTNYP KPGLKRTAST YLLSNSFKKS ATVSLPFIL GDPVCMPKRV ESNNNDTCLL YSDTLYDDPL IQRNDQAGDE IEDEFSFTLL RSEVNEIRPI SSSSTAQILQ S DYSALMYE RQASNGSIFQ FSSP UniProtKB: Meiotic recombination protein REC104 |
-分子 #2: Meiotic recombination protein REC102
| 分子 | 名称: Meiotic recombination protein REC102 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.263717 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MARDITFLTV FLESCGAVNN DEAGKLLSAW TSTVRIEGPE STDSNSLYIP LLPPGMLKIK LNFKMNDRLV TEEQELFTKL REIVGSSIR FWEEQLFYQV QDVSTIENHV ILSLKCTILT DAQISTFISK PRELHTHAKG YPEIYYLSEL STTVNFFSKE G NYVEISQV ...文字列: MARDITFLTV FLESCGAVNN DEAGKLLSAW TSTVRIEGPE STDSNSLYIP LLPPGMLKIK LNFKMNDRLV TEEQELFTKL REIVGSSIR FWEEQLFYQV QDVSTIENHV ILSLKCTILT DAQISTFISK PRELHTHAKG YPEIYYLSEL STTVNFFSKE G NYVEISQV IPHFNEYFSS LIVSQLEFEY PMVFSMISRL RLKWQQSSLA PISYALTSNS VLLPIMLNMI AQDKSSTTAY QI LCRRRGP PIQNFQIFSL PAVTYNK UniProtKB: Meiotic recombination protein REC102 |
-分子 #3: Meiosis-specific protein SPO11
| 分子 | 名称: Meiosis-specific protein SPO11 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 50.020109 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALEGLRKKY KTRQELVKAL TPKRRSIHLN SNGHSNGTPC SNADVLAHIK HFLSLAANSL EQHQQPISIV FQNKKKKGDT NSPDIHTTL DFPLNGPHLS THQFKLKRCA ILLNLLKVVM EKLPLGKNTT VRDIFYSNVE LFQRQANVVQ WLDVIRFNFK L SPRKSLNI ...文字列: MALEGLRKKY KTRQELVKAL TPKRRSIHLN SNGHSNGTPC SNADVLAHIK HFLSLAANSL EQHQQPISIV FQNKKKKGDT NSPDIHTTL DFPLNGPHLS THQFKLKRCA ILLNLLKVVM EKLPLGKNTT VRDIFYSNVE LFQRQANVVQ WLDVIRFNFK L SPRKSLNI IPAQKGLVYS PFPIDIYDNI LTCENEPKMQ KQTIFSGKPC LIPFFQDDAV IKLGTTSMCN IVIVEKEAVF TK LVNNYHK LSTNTMLITG KGFPDFLTRL FLKKLEQYCS NLISDCSIFT DADPYGISIA LNYTHSNERN AYICTMANYK GIR ITQVLA QNNEVHNKSI QLLSLNQRDY SLAKNLIASL TANSWDIATS PLKNVVIECQ REIFFQKKAE MNEIDAGIFK YKSR HHHHH HHHHHGDYKD DDDKDYKDDD DKDYKDDDDK UniProtKB: Meiosis-specific protein SPO11 |
-分子 #4: Antiviral protein SKI8
| 分子 | 名称: Antiviral protein SKI8 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 44.313555 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKVFIATAN AGKAHDADIF SVSACNSFTV SCSGDGYLKV WDNKLLDNEN PKDKSYSHFV HKSGLHHVDV LQTIERDAFE LCLVATTSF SGDLLFYRIT REDETKKVIF EKLDLLDSDM KKHSFWALKW GASNDRLLSH RLVATDVKGT TYIWKFHPFA D ESNSLTLN ...文字列: MSKVFIATAN AGKAHDADIF SVSACNSFTV SCSGDGYLKV WDNKLLDNEN PKDKSYSHFV HKSGLHHVDV LQTIERDAFE LCLVATTSF SGDLLFYRIT REDETKKVIF EKLDLLDSDM KKHSFWALKW GASNDRLLSH RLVATDVKGT TYIWKFHPFA D ESNSLTLN WSPTLELQGT VESPMTPSQF ATSVDISERG LIATGFNNGT VQISELSTLR PLYNFESQHS MINNSNSIRS VK FSPQGSL LAIAHDSNSF GCITLYETEF GERIGSLSVP THSSQASLGE FAHSSWVMSL SFNDSGETLC SAGWDGKLRF WDV KTKERI TTLNMHCDDI EIEEDILAVD EHGDSLAEPG VFDVKFLKKG WRSGMGADLN ESLCCVCLDR SIRWFREAGG K UniProtKB: Antiviral protein SKI8 |
-分子 #5: gapped DNA
| 分子 | 名称: gapped DNA / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.943064 KDa |
| 配列 | 文字列: (DT)(DA)(DG)(DG)(DC)(DC)(DG)(DT)(DC)(DG) (DG)(DC)(DT)(DA)(DC)(DT)(DA)(DA)(DA)(DA) (DG)(DT)(DA)(DG)(DC)(DC)(DG)(DA)(DC) (DG)(DG)(DC)(DC)(DG)(DG)(DA)(DT)(DT)(DA) (DG) (DC)(DA)(DA)(DT)(DG) ...文字列: (DT)(DA)(DG)(DG)(DC)(DC)(DG)(DT)(DC)(DG) (DG)(DC)(DT)(DA)(DC)(DT)(DA)(DA)(DA)(DA) (DG)(DT)(DA)(DG)(DC)(DC)(DG)(DA)(DC) (DG)(DG)(DC)(DC)(DG)(DG)(DA)(DT)(DT)(DA) (DG) (DC)(DA)(DA)(DT)(DG)(DT)(DA)(DA) (DT)(DC)(DG)(DT)(DC)(DT)(DT)(DA)(DA)(DG) (DA)(DC) (DG)(DA)(DT)(DT)(DA)(DC)(DA) (DT)(DT)(DG)(DC) |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 25 mM HEPES, pH 7.4, 300 mM NaCl, 5 mM EDTA, 2 mM DTT |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 3.0 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 548674 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X