+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | ssRNA phage PhiCb5 virion | |||||||||
 マップデータ マップデータ | map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ssRNA phage / VIRUS | |||||||||
| 機能・相同性 | Phage phiCb5, coat protein / Assembly protein / Phage maturation protein / virion attachment to host cell pilus / virion component / viral capsid / Maturation protein / Coat protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Caulobacter vibrioides (バクテリア) / Caulobacter vibrioides (バクテリア) /  Caulobacter phage phiCb5 (ファージ) Caulobacter phage phiCb5 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Wang Y / Zhang J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: Structural mechanisms of Tad pilus assembly and its interaction with an RNA virus. 著者: Yuhang Wang / Matthew Theodore / Zhongliang Xing / Utkarsh Narsaria / Zihao Yu / Lanying Zeng / Junjie Zhang /  要旨: Tad (tight adherence) pili, part of the type IV pili family, are crucial for mechanosensing, surface adherence, bacteriophage (phage) adsorption, and cell-cycle regulation. Unlike other type IV ... Tad (tight adherence) pili, part of the type IV pili family, are crucial for mechanosensing, surface adherence, bacteriophage (phage) adsorption, and cell-cycle regulation. Unlike other type IV pilins, Tad pilins lack the typical globular β sheet domain responsible for pilus assembly and phage binding. The mechanisms of Tad pilus assembly and its interaction with phage ΦCb5 have been elusive. Using cryo-electron microscopy, we unveiled the Tad pilus assembly mechanism, featuring a unique network of hydrogen bonds at its core. We then identified the Tad pilus binding to the ΦCb5 maturation protein (Mat) through its β region. Notably, the amino terminus of ΦCb5 Mat is exposed outside the capsid and phage/pilus interface, enabling the attachment of fluorescent and affinity tags. These engineered ΦCb5 virions can be efficiently assembled and purified in , maintaining infectivity against , which presents promising applications, including RNA delivery and phage display. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42163.map.gz emd_42163.map.gz | 456.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42163-v30.xml emd-42163-v30.xml emd-42163.xml emd-42163.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42163_fsc.xml emd_42163_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42163.png emd_42163.png | 68.4 KB | ||
| Filedesc metadata |  emd-42163.cif.gz emd-42163.cif.gz | 5.5 KB | ||
| その他 |  emd_42163_half_map_1.map.gz emd_42163_half_map_1.map.gz emd_42163_half_map_2.map.gz emd_42163_half_map_2.map.gz | 475.3 MB 475.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42163 http://ftp.pdbj.org/pub/emdb/structures/EMD-42163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42163 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42163_validation.pdf.gz emd_42163_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42163_full_validation.pdf.gz emd_42163_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_42163_validation.xml.gz emd_42163_validation.xml.gz | 26.4 KB | 表示 | |
| CIF形式データ |  emd_42163_validation.cif.gz emd_42163_validation.cif.gz | 34.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42163 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42163 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42163.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42163.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half
| ファイル | emd_42163_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
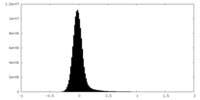
| 密度ヒストグラム |
-ハーフマップ: half
| ファイル | emd_42163_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
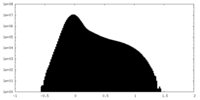
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A complex of phiCb5 maturation protein and phiCb5 shell
| 全体 | 名称: A complex of phiCb5 maturation protein and phiCb5 shell |
|---|---|
| 要素 |
|
-超分子 #1: A complex of phiCb5 maturation protein and phiCb5 shell
| 超分子 | 名称: A complex of phiCb5 maturation protein and phiCb5 shell タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: A complex of phiCb5 maturation protein and phiCb5 shell |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides (バクテリア) Caulobacter vibrioides (バクテリア) |
| 分子量 | 理論値: 2.1 MDa |
-分子 #1: Coat protein
| 分子 | 名称: Coat protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 178 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Caulobacter phage phiCb5 (ファージ) Caulobacter phage phiCb5 (ファージ) |
| 分子量 | 理論値: 13.498981 KDa |
| 配列 | 文字列: ALGDTLTITL GGSGGTAKVL RKINQDGYTS EYYLPETSSS FRAKVRHTKE SVKPNQVQYE RHNVEFTETV YASGSTPEFV RQAYVVIRH KVGDVSATVS DLGEALSFYL NEALYGKLIG WES UniProtKB: Coat protein |
-分子 #2: Maturation protein
| 分子 | 名称: Maturation protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Caulobacter phage phiCb5 (ファージ) Caulobacter phage phiCb5 (ファージ) |
| 分子量 | 理論値: 40.725336 KDa |
| 配列 | 文字列: MARIRNRSSI ASSGMSTFYL FGTPIVNEEI IVRNTEWCSD VIGNPGDNPL DIHKQEWTIK PLSGQIIFGS GTYRSLQCPP EYCRGASLS HLSLPSQSGL GTTALARTNP SRPAFNLPAF IGELRDLPRM FKIAGDTMLR KGANAFLSYQ FGWKPLISDI S KALDFSAT ...文字列: MARIRNRSSI ASSGMSTFYL FGTPIVNEEI IVRNTEWCSD VIGNPGDNPL DIHKQEWTIK PLSGQIIFGS GTYRSLQCPP EYCRGASLS HLSLPSQSGL GTTALARTNP SRPAFNLPAF IGELRDLPRM FKIAGDTMLR KGANAFLSYQ FGWKPLISDI S KALDFSAT VRTRSDEWHR LYSNGGLKRR INLGVDIEQK KENDVVLHSS NGFVVASHTV ITVRKTWATV RWRPDAGSLP PI TKSSSEK HARALLGLGV GGLIEGAWQL MPWSWMVDWF GNVGTFLQAS NNTIGASPGL VNIMTTTTTN HQFSVKRDLS DGW IKGGDC SATVTSKARS QSSGPTITAS IPNLSGRQLS ILGALGIQRV PRHLLR UniProtKB: Maturation protein |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 236 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20mM tris, 2mM MgCl2, 3mM CaCl2 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-2/1 | ||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 25.0 µm / 最小 デフォーカス(公称値): 5.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)