+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
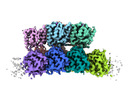
| タイトル | Structure of activated SAVED-CHAT filament | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SAVED-CHAT / IMMUNE SYSTEM | |||||||||
| 機能・相同性 | SMODS-associated and fused to various effectors / SMODS-associated and fused to various effectors sensor domain / CHAT domain / CHAT domain / CHAT domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Haliangium ochraceum (バクテリア) / synthetic construct (人工物) Haliangium ochraceum (バクテリア) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Bravo JPK / Taylor DW | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Type III-B CRISPR-Cas cascade of proteolytic cleavages. 著者: Jurre A Steens / Jack P K Bravo / Carl Raymund P Salazar / Caglar Yildiz / Afonso M Amieiro / Stephan Köstlbacher / Stijn H P Prinsen / Ane S Andres / Constantinos Patinios / Andreas Bardis ...著者: Jurre A Steens / Jack P K Bravo / Carl Raymund P Salazar / Caglar Yildiz / Afonso M Amieiro / Stephan Köstlbacher / Stijn H P Prinsen / Ane S Andres / Constantinos Patinios / Andreas Bardis / Arjan Barendregt / Richard A Scheltema / Thijs J G Ettema / John van der Oost / David W Taylor / Raymond H J Staals /   要旨: The generation of cyclic oligoadenylates and subsequent allosteric activation of proteins that carry sensory domains is a distinctive feature of type III CRISPR-Cas systems. In this work, we ...The generation of cyclic oligoadenylates and subsequent allosteric activation of proteins that carry sensory domains is a distinctive feature of type III CRISPR-Cas systems. In this work, we characterize a set of associated genes of a type III-B system from that contains two caspase-like proteases, SAVED-CHAT and PCaspase (prokaryotic caspase), co-opted from a cyclic oligonucleotide-based antiphage signaling system (CBASS). Cyclic tri-adenosine monophosphate (AMP)-induced oligomerization of SAVED-CHAT activates proteolytic activity of the CHAT domains, which specifically cleave and activate PCaspase. Subsequently, activated PCaspase cleaves a multitude of proteins, which results in a strong interference phenotype in vivo in Taken together, our findings reveal how a CRISPR-Cas-based detection of a target RNA triggers a cascade of caspase-associated proteolytic activities. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41358.map.gz emd_41358.map.gz | 256.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41358-v30.xml emd-41358-v30.xml emd-41358.xml emd-41358.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41358.png emd_41358.png | 89.7 KB | ||
| マスクデータ |  emd_41358_msk_1.map emd_41358_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41358.cif.gz emd-41358.cif.gz | 5.8 KB | ||
| その他 |  emd_41358_additional_1.map.gz emd_41358_additional_1.map.gz emd_41358_half_map_1.map.gz emd_41358_half_map_1.map.gz emd_41358_half_map_2.map.gz emd_41358_half_map_2.map.gz | 483.5 MB 474.4 MB 474.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41358 http://ftp.pdbj.org/pub/emdb/structures/EMD-41358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41358 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41358_validation.pdf.gz emd_41358_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41358_full_validation.pdf.gz emd_41358_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_41358_validation.xml.gz emd_41358_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_41358_validation.cif.gz emd_41358_validation.cif.gz | 22.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41358 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41358 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41358 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tl0MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41358.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41358.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.94 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41358_msk_1.map emd_41358_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
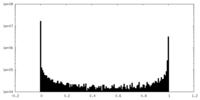
| 密度ヒストグラム |
-追加マップ: Sharpened map
| ファイル | emd_41358_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
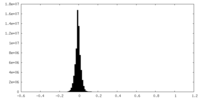
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41358_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
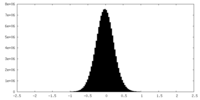
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41358_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
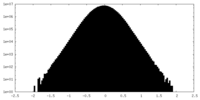
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Active SAVED-CHAT filament
| 全体 | 名称: Active SAVED-CHAT filament |
|---|---|
| 要素 |
|
-超分子 #1: Active SAVED-CHAT filament
| 超分子 | 名称: Active SAVED-CHAT filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Haliangium ochraceum (バクテリア) Haliangium ochraceum (バクテリア) |
| 分子量 | 理論値: 1.33 MDa |
-分子 #1: CHAT domain-containing protein
| 分子 | 名称: CHAT domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Haliangium ochraceum (バクテリア) Haliangium ochraceum (バクテリア) |
| 分子量 | 理論値: 54.378332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: IQCILVLDLS IDNAITACSV TPHLPRAARR VELHLNDFGA ERAPYGGASD RRTWRCWMQA VDAMLADARA QLGAEVEFTH YYLAGRAAL PVFAYLGLRL GKQANITTVN RRDDGCWDVV PCQRPASSAA SGVSPSARFF DEVRGLDTDE RSSESGMVAV W VSTQRDVD ...文字列: IQCILVLDLS IDNAITACSV TPHLPRAARR VELHLNDFGA ERAPYGGASD RRTWRCWMQA VDAMLADARA QLGAEVEFTH YYLAGRAAL PVFAYLGLRL GKQANITTVN RRDDGCWDVV PCQRPASSAA SGVSPSARFF DEVRGLDTDE RSSESGMVAV W VSTQRDVD RGLLRAFARA RGDRDLAGIV SLRARPAAGD DTGDMRLLEG ADGPDAAREL VNCFRSIPNQ YPRSSGLMVF VS GPVTLAA MVGRAINPRI HGPVWWPYFR GGEYEPALEY PWPLISGPPR ILIATANAPE GENPTLDVEA ELKHLEEALA EPR KRKLCE VQRCPAATVS DITSALRSFK PHILHFIGHG TALGVYLRSA EHDGAQFVRG EDFQQMIATS LRQKDREMHL VVLN ACCTH ELAKALTEQV SCTIGTDIEV YDSASIHFAA RFYDHLVHGT SVHYAFNAAV DECRAHSTSG QEVFCLHPAA ERAPA SATP PVRADELVFF SP UniProtKB: CHAT domain-containing protein |
-分子 #2: RNA (5'-R(*AP*AP*A)-3')
| 分子 | 名称: RNA (5'-R(*AP*AP*A)-3') / タイプ: rna / ID: 2 / コピー数: 7 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 942.66 Da |
| 配列 | 文字列: AAA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 38548 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー



 Z
Z Y
Y X
X