+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the wild-type AtMSL10 in saposin | |||||||||
 マップデータ マップデータ | NU-refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channels / mechanosensitive channels / heptamer / Arabidopsis thaliana / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報programmed cell death in response to reactive oxygen species / leaf senescence / detection of mechanical stimulus / mechanosensitive monoatomic ion channel activity / monoatomic anion transport / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Zhang J / Yuan P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Open structure and gating of the Arabidopsis mechanosensitive ion channel MSL10. 著者: Jingying Zhang / Grigory Maksaev / Peng Yuan /  要旨: Plants are challenged by drastically different osmotic environments during growth and development. Adaptation to these environments often involves mechanosensitive ion channels that can detect and ...Plants are challenged by drastically different osmotic environments during growth and development. Adaptation to these environments often involves mechanosensitive ion channels that can detect and respond to mechanical force. In the model plant Arabidopsis thaliana, the mechanosensitive channel MSL10 plays a crucial role in hypo-osmotic shock adaptation and programmed cell death induction, but the molecular basis of channel function remains poorly understood. Here, we report a structural and electrophysiological analysis of MSL10. The cryo-electron microscopy structures reveal a distinct heptameric channel assembly. Structures of the wild-type channel in detergent and lipid environments, and in the absence of membrane tension, capture an open conformation. Furthermore, structural analysis of a non-conductive mutant channel demonstrates that reorientation of phenylalanine side chains alone, without main chain rearrangements, may generate the hydrophobic gate. Together, these results reveal a distinct gating mechanism and advance our understanding of mechanotransduction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41166.map.gz emd_41166.map.gz | 51.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41166-v30.xml emd-41166-v30.xml emd-41166.xml emd-41166.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41166.png emd_41166.png | 132 KB | ||
| Filedesc metadata |  emd-41166.cif.gz emd-41166.cif.gz | 6.3 KB | ||
| その他 |  emd_41166_additional_1.map.gz emd_41166_additional_1.map.gz emd_41166_additional_2.map.gz emd_41166_additional_2.map.gz emd_41166_half_map_1.map.gz emd_41166_half_map_1.map.gz emd_41166_half_map_2.map.gz emd_41166_half_map_2.map.gz | 91.6 MB 51.2 MB 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41166 http://ftp.pdbj.org/pub/emdb/structures/EMD-41166 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41166 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41166 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_41166_validation.pdf.gz emd_41166_validation.pdf.gz | 792.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_41166_full_validation.pdf.gz emd_41166_full_validation.pdf.gz | 792.1 KB | 表示 | |
| XML形式データ |  emd_41166_validation.xml.gz emd_41166_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_41166_validation.cif.gz emd_41166_validation.cif.gz | 15.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41166 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41166 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41166 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-41166 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41166.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41166.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | NU-refinement | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: deepEMhancer
| ファイル | emd_41166_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | deepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
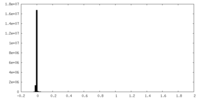
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_41166_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
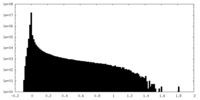
| 密度ヒストグラム |
-ハーフマップ: half A
| ファイル | emd_41166_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half_A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
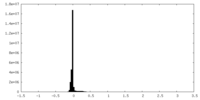
| 密度ヒストグラム |
-ハーフマップ: half B
| ファイル | emd_41166_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half_B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
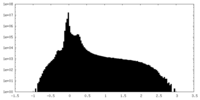
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AtMSL10 in saposin
| 全体 | 名称: AtMSL10 in saposin |
|---|---|
| 要素 |
|
-超分子 #1: AtMSL10 in saposin
| 超分子 | 名称: AtMSL10 in saposin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Mechanosensitive ion channel protein 10
| 分子 | 名称: Mechanosensitive ion channel protein 10 / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 83.955562 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MAEQKSSNGG GGGGDVVINV PVEEASRRSK EMASPESEKG VPFSKSPSPE ISKLVGSPNK PPRAPNQNNV GLTQRKSFAR SVYSKPKSR FVDPSCPVDT SILEEEVREQ LGAGFSFSRA SPNNKSNRSV GSPAPVTPSK VVVEKDEDEE IYKKVKLNRE M RSKISTLA ...文字列: MAEQKSSNGG GGGGDVVINV PVEEASRRSK EMASPESEKG VPFSKSPSPE ISKLVGSPNK PPRAPNQNNV GLTQRKSFAR SVYSKPKSR FVDPSCPVDT SILEEEVREQ LGAGFSFSRA SPNNKSNRSV GSPAPVTPSK VVVEKDEDEE IYKKVKLNRE M RSKISTLA LIESAFFVVI LSALVASLTI NVLKHHTFWG LEVWKWCVLV MVIFSGMLVT NWFMRLIVFL IETNFLLRRK VL YFVHGLK KSVQVFIWLC LILVAWILLF NHDVKRSPAA TKVLKCITRT LISILTGAFF WLVKTLLLKI LAANFNVNNF FDR IQDSVF HQYVLQTLSG LPLMEEAERV GREPSTGHLS FATVVKKGTV KEKKVIDMGK VHKMKREKVS AWTMRVLMEA VRTS GLSTI SDTLDETAYG EGKEQADREI TSEMEALAAA YHVFRNVAQP FFNYIEEEDL LRFMIKEEVD LVFPLFDGAA ETGRI TRKA FTEWVVKVYT SRRALAHSLN DTKTAVKQLN KLVTAILMVV TVVIWLLLLE VATTKVLLFF STQLVALAFI IGSTCK NLF ESIVFVFVMH PYDVGDRCVV DGVAMLVEEM NLLTTVFLKL NNEKVYYPNA VLATKPISNY FRSPNMGETV EFSISFS TP VSKIAHLKER IAEYLEQNPQ HWAPVHSVVV KEIENMNKLK MALYSDHTIT FQENRERNLR RTELSLAIKR MLEDLHID Y TLLPQDINLT KKNSLEVLFQ UniProtKB: Mechanosensitive ion channel protein 10 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 20 mM Tris-HCl and 150 mM NaCl | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 2120 / 平均電子線量: 54.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 59000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X