+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 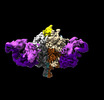 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5 | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | vaccine / GPC / GP1 / GP2 / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / receptor-mediated endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Lassa virus Josiah (ウイルス) / Lassa virus Josiah (ウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cleavage-intermediate Lassa virus trimer elicits neutralizing responses, identifies neutralizing nanobodies, and reveals an apex-situated site-of-vulnerability. 著者: Jason Gorman / Crystal Sao-Fong Cheung / Zhijian Duan / Li Ou / Maple Wang / Xuejun Chen / Cheng Cheng / Andrea Biju / Yaping Sun / Pengfei Wang / Yongping Yang / Baoshan Zhang / Jeffrey C ...著者: Jason Gorman / Crystal Sao-Fong Cheung / Zhijian Duan / Li Ou / Maple Wang / Xuejun Chen / Cheng Cheng / Andrea Biju / Yaping Sun / Pengfei Wang / Yongping Yang / Baoshan Zhang / Jeffrey C Boyington / Tatsiana Bylund / Sam Charaf / Steven J Chen / Haijuan Du / Amy R Henry / Tracy Liu / Edward K Sarfo / Chaim A Schramm / Chen-Hsiang Shen / Tyler Stephens / I-Ting Teng / John-Paul Todd / Yaroslav Tsybovsky / Raffaello Verardi / Danyi Wang / Shuishu Wang / Zhantong Wang / Cheng-Yan Zheng / Tongqing Zhou / Daniel C Douek / John R Mascola / David D Ho / Mitchell Ho / Peter D Kwong /  要旨: Lassa virus (LASV) infection is expanding outside its traditionally endemic areas in West Africa, posing a pandemic biothreat. LASV-neutralizing antibodies, moreover, have proven difficult to elicit. ...Lassa virus (LASV) infection is expanding outside its traditionally endemic areas in West Africa, posing a pandemic biothreat. LASV-neutralizing antibodies, moreover, have proven difficult to elicit. To gain insight into LASV neutralization, here we develop a prefusion-stabilized LASV glycoprotein trimer (GPC), pan it against phage libraries comprising single-domain antibodies (nanobodies) from shark and camel, and identify one, D5, which neutralizes LASV. Cryo-EM analyses reveal D5 to recognize a cleavage-dependent site-of-vulnerability at the trimer apex. The recognized site appears specific to GPC intermediates, with protomers lacking full cleavage between GP1 and GP2 subunits. Guinea pig immunizations with the prefusion-stabilized cleavage-intermediate LASV GPC, first as trimer and then as a nanoparticle, induce neutralizing responses, targeting multiple epitopes including that of D5; we identify a neutralizing antibody (GP23) from the immunized guinea pigs. Collectively, our findings define a prefusion-stabilized GPC trimer, reveal an apex-situated site-of-vulnerability, and demonstrate elicitation of LASV-neutralizing responses by a cleavage-intermediate LASV trimer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
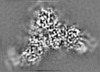
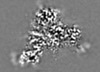
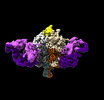
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41048.map.gz emd_41048.map.gz | 97.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41048-v30.xml emd-41048-v30.xml emd-41048.xml emd-41048.xml | 26.3 KB 26.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41048.png emd_41048.png | 72.2 KB | ||
| マスクデータ |  emd_41048_msk_1.map emd_41048_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41048.cif.gz emd-41048.cif.gz | 7.2 KB | ||
| その他 |  emd_41048_additional_1.map.gz emd_41048_additional_1.map.gz emd_41048_additional_2.map.gz emd_41048_additional_2.map.gz emd_41048_half_map_1.map.gz emd_41048_half_map_1.map.gz emd_41048_half_map_2.map.gz emd_41048_half_map_2.map.gz | 51.2 MB 7.1 MB 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41048 http://ftp.pdbj.org/pub/emdb/structures/EMD-41048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41048 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41048 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41048.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41048.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
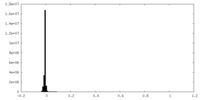
| 注釈 | Sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.076 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41048_msk_1.map emd_41048_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
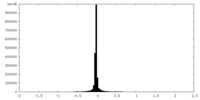
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_41048_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: density modified (resolve)
| ファイル | emd_41048_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density modified (resolve) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_41048_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_41048_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5
| 全体 | 名称: Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5 |
|---|---|
| 要素 |
|
-超分子 #1: Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5
| 超分子 | 名称: Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  Lassa virus Josiah (ウイルス) Lassa virus Josiah (ウイルス) |
-分子 #1: Glycoprotein G1
| 分子 | 名称: Glycoprotein G1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa virus Josiah (ウイルス) Lassa virus Josiah (ウイルス) |
| 分子量 | 理論値: 22.884879 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TSLYKGVYEL QTLELNMETL NMTMPLSCTK NNSHHYIMVG NETGLELTLT NTSIINHKFC NLSDAHKKNL YDHALMSIIS TFHLSIPNF NQYEAMSCDF NGGKISVQYN LSHSYAGDAA NHCGTVANGV LQTFMRMAWG GSYIALDSGG CGNWDCIMTS Y QYLIIQNT ...文字列: TSLYKGVYEL QTLELNMETL NMTMPLSCTK NNSHHYIMVG NETGLELTLT NTSIINHKFC NLSDAHKKNL YDHALMSIIS TFHLSIPNF NQYEAMSCDF NGGKISVQYN LSHSYAGDAA NHCGTVANGV LQTFMRMAWG GSYIALDSGG CGNWDCIMTS Y QYLIIQNT TWEDHCQFSR PSPIGYLGLL SQRTRDIYIS RRRR UniProtKB: Pre-glycoprotein polyprotein GP complex |
-分子 #2: Glycoprotein G2
| 分子 | 名称: Glycoprotein G2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa virus Josiah (ウイルス) Lassa virus Josiah (ウイルス) |
| 分子量 | 理論値: 22.14317 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GTFTWTLSDS EGKDTPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRCKAP AQMSIQLINK AVNALINDQ LIMKNHLRDI MGIPYCNYSK YWYLNHTTTG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE G GGYIPEAP ...文字列: GTFTWTLSDS EGKDTPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRCKAP AQMSIQLINK AVNALINDQ LIMKNHLRDI MGIPYCNYSK YWYLNHTTTG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE G GGYIPEAP RDGQAYVRKD GEWVLLSTFL GGLVPR UniProtKB: Pre-glycoprotein polyprotein GP complex |
-分子 #3: D5 nanobody
| 分子 | 名称: D5 nanobody / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.228544 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AWQLVESGGG SVQPGGSLTL TCQASKSTFS TSGMRWERQA QGKGVEFVAD ISSDSTRKWY SDSVKGRFTI SRSNWWRTVT LQMNDLKPE DTARYYCKDL ESHHLRGQGT QVTVSSSGQA G |
-分子 #4: 8.11G Heavy Chain
| 分子 | 名称: 8.11G Heavy Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.257604 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DQVQLQESGP GLVKPSETLS LTCSISGVST RNYYWSWIRQ SPGKGLEWIG YIFNIGTTNY NPSLKSRLTI SVDTSKNQFS LKITSVTAA DTAVYYCASG FEYGDYTFDY WGQGTPVTVS S |
-分子 #5: 8.11G Light Chain
| 分子 | 名称: 8.11G Light Chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.101508 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DEIVLTQSPA TLSVSPGGRA SLSCRASQSI GDKLSWYQQK PGQAPRLVIY GAYTRATDIS PRFSGSRSGT DFNLTISRMQ SGDFAVYFC QQYENWPRTF GQGTKLEIKR |
-分子 #14: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 14 / コピー数: 23 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 2.0 sec. / 平均電子線量: 51.15 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X