+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Trypanosoma (MDH)4-Pex5, close conformation | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Peroxisomal transport / Trypanosoma / Import / PEX / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報peroxisome matrix targeting signal-1 binding / protein import into peroxisome matrix, docking / malate dehydrogenase / L-malate dehydrogenase (NAD+) activity / carboxylic acid metabolic process / peroxisomal membrane / tricarboxylic acid cycle / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.33 Å | |||||||||
 データ登録者 データ登録者 | Sonani RR / Artur B / Jemiola-Rzeminska M / Lipinski O / Patel SN / Sood T / Dubin G | |||||||||
| 資金援助 |  ポーランド, 2件 ポーランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Biorxiv / 年: 2023 ジャーナル: Biorxiv / 年: 2023タイトル: Noncanonical interactions and conformational dynamics in cargo-Pex5-Pex14 ternary complex for peroxisomal import 著者: Sonani RR / Blat A / Jemiola-Rzeminska M / Lipinski O / Patel SN / Sood T / Dubin G | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40003.map.gz emd_40003.map.gz | 110.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40003-v30.xml emd-40003-v30.xml emd-40003.xml emd-40003.xml | 15 KB 15 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40003.png emd_40003.png | 75.9 KB | ||
| Filedesc metadata |  emd-40003.cif.gz emd-40003.cif.gz | 5.9 KB | ||
| その他 |  emd_40003_half_map_1.map.gz emd_40003_half_map_1.map.gz emd_40003_half_map_2.map.gz emd_40003_half_map_2.map.gz | 200.4 MB 200.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40003 http://ftp.pdbj.org/pub/emdb/structures/EMD-40003 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40003 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40003 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40003_validation.pdf.gz emd_40003_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40003_full_validation.pdf.gz emd_40003_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_40003_validation.xml.gz emd_40003_validation.xml.gz | 15.6 KB | 表示 | |
| CIF形式データ |  emd_40003_validation.cif.gz emd_40003_validation.cif.gz | 18.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40003 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40003 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40003 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40003 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40003.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40003.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_40003_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40003_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of MDH-tetramer and PEX5
| 全体 | 名称: Complex of MDH-tetramer and PEX5 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of MDH-tetramer and PEX5
| 超分子 | 名称: Complex of MDH-tetramer and PEX5 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: malate dehydrogenase
| 分子 | 名称: malate dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.106832 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVNVAVIGAA GGIGQSLSLL LLRELPFGST LSLYDVVGAP GVAADLSHID RAGITVKHAA GKLPPVPRDP ALTELAEGVD VFVIVAGVP RKPGMTRDDL FNVNAGIVMD LVLTCASVSP NACFCIVTNP VNSTTPIAAQ TLRKIGVYNK NKLLGVSLLD G LRATRFIN ...文字列: MVNVAVIGAA GGIGQSLSLL LLRELPFGST LSLYDVVGAP GVAADLSHID RAGITVKHAA GKLPPVPRDP ALTELAEGVD VFVIVAGVP RKPGMTRDDL FNVNAGIVMD LVLTCASVSP NACFCIVTNP VNSTTPIAAQ TLRKIGVYNK NKLLGVSLLD G LRATRFIN NARHPLVVPY VPVVGGHSDV TIVPLYSQIP GPLPDESTLK EIRKRVQVAG TEVVKAKAGR GSATLSMAEA GA RFTMHVV KALMGLDTPM VYAYVDTDGE HECPFLAMPV VLGKNGIERR LPIGPITTVE KEMLEEAVGV VKKNIAKGET FAR SKL UniProtKB: malate dehydrogenase |
-分子 #2: Peroxisome targeting signal 1 receptor
| 分子 | 名称: Peroxisome targeting signal 1 receptor / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 74.268422 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDCSTGAAIG QQFAKDAFHM HGGVGVGPTG NSEHDVLMNE MMMVQTPTGP AGEWTHQFAA YQGQQQQQQQ QHPQELAMRH QQNDAFMLR QQQEMEEAFC TFCTTHPHSH AHSHQPQGLV GPAMMGPQIM PPMMFGPGTG GFMMGAPPMM PYASMKFAGD A AMAAANNT ...文字列: MDCSTGAAIG QQFAKDAFHM HGGVGVGPTG NSEHDVLMNE MMMVQTPTGP AGEWTHQFAA YQGQQQQQQQ QHPQELAMRH QQNDAFMLR QQQEMEEAFC TFCTTHPHSH AHSHQPQGLV GPAMMGPQIM PPMMFGPGTG GFMMGAPPMM PYASMKFAGD A AMAAANNT NMTQGATATS TTSVQQELQQ QSSDNGWVEK LRDAEWAQDY SDAQVFTLEG QSEQTMEEHA KNSEFYQFMD KI RSKELLI DEETGQLVQG PGPDPDAPED AEYLKEWAAA EGLNMPPGFF EHMMQRPQGN NEQAEGRLFD GSNDALMDDG ALD NAADVE EWVREYAEAQ EQLQRVQNET NYPFEPNNPY MYHDKPMEEG IAMLQLANMA EAALAFEAVC QKEPENVEAW RRLG TTQAE NEKDCLAIIA LNHARMLDPK DIAVHAALAV SHTNEHNVGA ALQSLRSWLL SQPQYEHLGL VDLREVAADE GLDEV PEEN YFFAAPSEYR DCCTLLYAAV EMNPNDPQLH ASLGVLHNLS HRFDEAAKNF RRAVELRPDD AHMWNKLGAT LANGNR PQE ALEAYNRALD INPGYVRVMY NMAVSYSNMA QYPLAAKHIT RAIALQAGGT NPQGEGSRIA TRGLWDLLRM TLNLMDR SD LVEASWQQDL TPFLREFGLE EMAV UniProtKB: Peroxisome targeting signal 1 receptor |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 42.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.33 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 25845 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー



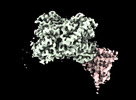















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)