+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of CTR-bound type VII CRISPR-Cas complex at substrate-engaged state I | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | a protein complex / ANTIVIRAL PROTEIN | |||||||||
| 生物種 | metagenome (メタゲノム) | |||||||||
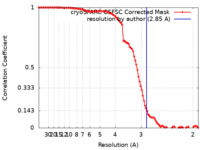
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.85 Å | |||||||||
 データ登録者 データ登録者 | Zhang H / Deng Z / Li X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structural basis for the activity of the type VII CRISPR-Cas system. 著者: Jie Yang / Xuzichao Li / Qiuqiu He / Xiaoshen Wang / Jingjing Tang / Tongyao Wang / Yi Zhang / Feiyang Yu / Shuqin Zhang / Zhikun Liu / Lingling Zhang / Fumeng Liao / Hang Yin / Haiyan Zhao / ...著者: Jie Yang / Xuzichao Li / Qiuqiu He / Xiaoshen Wang / Jingjing Tang / Tongyao Wang / Yi Zhang / Feiyang Yu / Shuqin Zhang / Zhikun Liu / Lingling Zhang / Fumeng Liao / Hang Yin / Haiyan Zhao / Zengqin Deng / Heng Zhang /  要旨: The newly identified type VII CRISPR-Cas candidate system uses a CRISPR RNA-guided ribonucleoprotein complex formed by Cas5 and Cas7 proteins to target RNA. However, the RNA cleavage is executed by a ...The newly identified type VII CRISPR-Cas candidate system uses a CRISPR RNA-guided ribonucleoprotein complex formed by Cas5 and Cas7 proteins to target RNA. However, the RNA cleavage is executed by a dedicated Cas14 nuclease, which is distinct from the effector nucleases of the other CRISPR-Cas systems. Here we report seven cryo-electron microscopy structures of the Cas14-bound interference complex at different functional states. Cas14, a tetrameric protein in solution, is recruited to the Cas5-Cas7 complex in a target RNA-dependent manner. The N-terminal catalytic domain of Cas14 binds a stretch of the substrate RNA for cleavage, whereas the C-terminal domain is primarily responsible for tethering Cas14 to the Cas5-Cas7 complex. The biochemical cleavage assays corroborate the captured functional conformations, revealing that Cas14 binds to different sites on the Cas5-Cas7 complex to execute individual cleavage events. Notably, a plugged-in arginine of Cas7 sandwiched by a C-shaped clamp of C-terminal domain precisely modulates Cas14 binding. More interestingly, target RNA cleavage is altered by a complementary protospacer flanking sequence at the 5' end, but not at the 3' end. Altogether, our study elucidates critical molecular details underlying the assembly of the interference complex and substrate cleavage in the type VII CRISPR-Cas system, which may help rational engineering of the type VII CRISPR-Cas system for biotechnological applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39767.map.gz emd_39767.map.gz | 97.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39767-v30.xml emd-39767-v30.xml emd-39767.xml emd-39767.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_39767_fsc.xml emd_39767_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_39767.png emd_39767.png | 65.4 KB | ||
| Filedesc metadata |  emd-39767.cif.gz emd-39767.cif.gz | 6.1 KB | ||
| その他 |  emd_39767_half_map_1.map.gz emd_39767_half_map_1.map.gz emd_39767_half_map_2.map.gz emd_39767_half_map_2.map.gz | 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39767 http://ftp.pdbj.org/pub/emdb/structures/EMD-39767 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39767 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39767 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_39767_validation.pdf.gz emd_39767_validation.pdf.gz | 971.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_39767_full_validation.pdf.gz emd_39767_full_validation.pdf.gz | 971.4 KB | 表示 | |
| XML形式データ |  emd_39767_validation.xml.gz emd_39767_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_39767_validation.cif.gz emd_39767_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39767 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39767 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39767 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-39767 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39767.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39767.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||||||||||||||||||
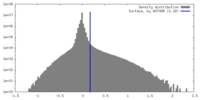
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_39767_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
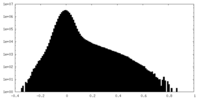
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_39767_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
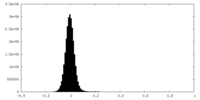
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : a protein
| 全体 | 名称: a protein |
|---|---|
| 要素 |
|
-超分子 #1: a protein
| 超分子 | 名称: a protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
-分子 #1: a protein
| 分子 | 名称: a protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 22.255604 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAKTMKKIYV TMKTLSPLYT GEVRREDKEA AQKRVNFPVR KTATNKVLIP FKGALRSALE IMLKAKGENV CDTGESRARP CGRCVTCSL FGSMGRAGRA SVDFLISNDT KEQIVRESTH LRIERQTKSA SDTFKGEEVI EGATFTATIT ISNPQEKDLS L IQSALKFI ...文字列: MAKTMKKIYV TMKTLSPLYT GEVRREDKEA AQKRVNFPVR KTATNKVLIP FKGALRSALE IMLKAKGENV CDTGESRARP CGRCVTCSL FGSMGRAGRA SVDFLISNDT KEQIVRESTH LRIERQTKSA SDTFKGEEVI EGATFTATIT ISNPQEKDLS L IQSALKFI EENGIGGWLN KGYGRVSFEV KSEDVATDRF LK |
-分子 #2: a protein
| 分子 | 名称: a protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 27.139533 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKEIKGILES ITGFSIPLDN GEYALYPAGR HLRGAIGYIA FNLDLPISSK FLDFDFDDII FRDLLPISKC GKIFYPEKNS NSLKCPSCN EIYGSSVLRN IMARGLSYKE VIEGKKYRLS IIVKDEKYLN EMEAIIRYIL SYGIYLGNKV SKGYGKFKIK E YSIVDILP ...文字列: MKEIKGILES ITGFSIPLDN GEYALYPAGR HLRGAIGYIA FNLDLPISSK FLDFDFDDII FRDLLPISKC GKIFYPEKNS NSLKCPSCN EIYGSSVLRN IMARGLSYKE VIEGKKYRLS IIVKDEKYLN EMEAIIRYIL SYGIYLGNKV SKGYGKFKIK E YSIVDILP VKDSEVLLLS DAIIDNGEKD IVFSKKEISS SKFEIIRKRG KAKGDIIRDN NHNGFYIGKY GGLGFGEIIS LK |
-分子 #3: a protein
| 分子 | 名称: a protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 70.468898 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIKFIGGASK VTGSAFLLET GNAKILIDCG IEQEKGIEKD NNEIIEKKIN EIGKADICIL THAHLAASGL VPLLVKKRKV NKIISTPAT KELCRLLFND FQRIQEENND IPLYSYDDIE SSFEIWDEID DRNTIELFDT KITFYNNSHI IGSVSVFIET H NGNYLFSG ...文字列: MIKFIGGASK VTGSAFLLET GNAKILIDCG IEQEKGIEKD NNEIIEKKIN EIGKADICIL THAHLAASGL VPLLVKKRKV NKIISTPAT KELCRLLFND FQRIQEENND IPLYSYDDIE SSFEIWDEID DRNTIELFDT KITFYNNSHI IGSVSVFIET H NGNYLFSG DIGSKLQQLM DYPPDMPDGN VDYLILESTY GNKSHDSSDR DRLLEIAKTT CENGGKVLIP SFAIGRLQEV LY TFSNYNF NFPVYIDSPM GSKVTNLIKE YNIYLKKKLR RLSITDDLFN NKYIAINTSN QSKELSNSKE PAVIISASGM LEG GRILNH LEQIKNDENS TLIFVGYQAQ NTRGRKILDG EEKVRCRIEK LNSFSAHADQ DELIDYIERL KYTPYKVFLV HGEK EQREI LAKRIISKKI RVELPENYSQ GKEILIEKKV VLNINTDNMC NFASYRLMPF SGFIVEKDDR IEINDKNWFD MIWNE EYNK MRSQIVAEDF STDQNEDSMA LPDMSHDKII ENIEYLFNIK ILSKNRIKEF WEEFCKGQKA AIKYITQVHR KNPNTG RRN WNPPEGDFTD NEIEKLYETA YNTLLSLIKY DKNKVYNILI NFNPKL |
-分子 #4: RNA (49-MER)
| 分子 | 名称: RNA (49-MER) / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 19.142277 KDa |
| 配列 | 文字列: GGUUAAAACU CUUCUCAUGC UGGAUUCGAA AUUAGGUGCG CUUCGCGUUU AAGUCCCAUA |
-分子 #5: RNA (40-MER)
| 分子 | 名称: RNA (40-MER) / タイプ: rna / ID: 5 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: metagenome (メタゲノム) |
| 分子量 | 理論値: 19.354707 KDa |
| 配列 | 文字列: GAACAGAAGA ACACCUAAAC GCGAAGCGCA CCUAAUUUCG AAUCCAGCAU GAGAAGCUAA |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 9 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)