+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3860 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of calcium-bound mTMEM16A chloride channel at 3.75 A resolution | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TMEM16 family / ion channel / membrane protein / cryo-EM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glial cell projection elongation / trachea development / iodide transmembrane transporter activity / iodide transport / mucus secretion / intracellularly calcium-gated chloride channel activity / cellular response to peptide / voltage-gated chloride channel activity / Stimuli-sensing channels / chloride transport ...glial cell projection elongation / trachea development / iodide transmembrane transporter activity / iodide transport / mucus secretion / intracellularly calcium-gated chloride channel activity / cellular response to peptide / voltage-gated chloride channel activity / Stimuli-sensing channels / chloride transport / chloride channel activity / positive regulation of insulin secretion involved in cellular response to glucose stimulus / detection of temperature stimulus involved in sensory perception of pain / chloride channel complex / chloride transmembrane transport / regulation of membrane potential / cell projection / establishment of localization in cell / presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / cellular response to heat / apical plasma membrane / external side of plasma membrane / signaling receptor binding / glutamatergic synapse / protein homodimerization activity / identical protein binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.75 Å | |||||||||
 データ登録者 データ登録者 | Paulino C / Kalienkova V | |||||||||
| 資金援助 |  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Activation mechanism of the calcium-activated chloride channel TMEM16A revealed by cryo-EM. 著者: Cristina Paulino / Valeria Kalienkova / Andy K M Lam / Yvonne Neldner / Raimund Dutzler /  要旨: The calcium-activated chloride channel TMEM16A is a ligand-gated anion channel that opens in response to an increase in intracellular Ca concentration. The protein is broadly expressed and ...The calcium-activated chloride channel TMEM16A is a ligand-gated anion channel that opens in response to an increase in intracellular Ca concentration. The protein is broadly expressed and contributes to diverse physiological processes, including transepithelial chloride transport and the control of electrical signalling in smooth muscles and certain neurons. As a member of the TMEM16 (or anoctamin) family of membrane proteins, TMEM16A is closely related to paralogues that function as scramblases, which facilitate the bidirectional movement of lipids across membranes. The unusual functional diversity of the TMEM16 family and the relationship between two seemingly incompatible transport mechanisms has been the focus of recent investigations. Previous breakthroughs were obtained from the X-ray structure of the lipid scramblase of the fungus Nectria haematococca (nhTMEM16), and from the cryo-electron microscopy structure of mouse TMEM16A at 6.6 Å (ref. 14). Although the latter structure disclosed the architectural differences that distinguish ion channels from lipid scramblases, its low resolution did not permit a detailed molecular description of the protein or provide any insight into its activation by Ca. Here we describe the structures of mouse TMEM16A at high resolution in the presence and absence of Ca. These structures reveal the differences between ligand-bound and ligand-free states of a calcium-activated chloride channel, and when combined with functional experiments suggest a mechanism for gating. During activation, the binding of Ca to a site located within the transmembrane domain, in the vicinity of the pore, alters the electrostatic properties of the ion conduction path and triggers a conformational rearrangement of an α-helix that comes into physical contact with the bound ligand, and thereby directly couples ligand binding and pore opening. Our study describes a process that is unique among channel proteins, but one that is presumably general for both functional branches of the TMEM16 family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3860.map.gz emd_3860.map.gz | 96.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3860-v30.xml emd-3860-v30.xml emd-3860.xml emd-3860.xml | 24.9 KB 24.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3860_fsc.xml emd_3860_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3860.png emd_3860.png | 235 KB | ||
| マスクデータ |  emd_3860_msk_1.map emd_3860_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-3860.cif.gz emd-3860.cif.gz | 7.9 KB | ||
| その他 |  emd_3860_additional.map.gz emd_3860_additional.map.gz emd_3860_half_map_1.map.gz emd_3860_half_map_1.map.gz emd_3860_half_map_2.map.gz emd_3860_half_map_2.map.gz | 96.4 MB 96.2 MB 96.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3860 http://ftp.pdbj.org/pub/emdb/structures/EMD-3860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3860 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3860 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3860_validation.pdf.gz emd_3860_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3860_full_validation.pdf.gz emd_3860_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_3860_validation.xml.gz emd_3860_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_3860_validation.cif.gz emd_3860_validation.cif.gz | 23.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3860 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3860 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3860 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3860.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3860.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.075 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
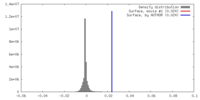
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_3860_msk_1.map emd_3860_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
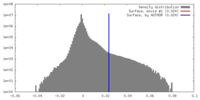
| 密度ヒストグラム |
-追加マップ: None
| ファイル | emd_3860_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
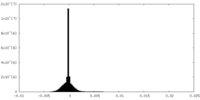
| 密度ヒストグラム |
-ハーフマップ: cryo-EM half map 2 of the ion channel...
| ファイル | emd_3860_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-EM half map 2 of the ion channel TMEM16A from mouse in presence of calcium ions | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
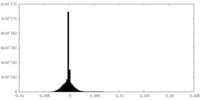
| 密度ヒストグラム |
-ハーフマップ: cryo-EM half map 1 of the ion channel...
| ファイル | emd_3860_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryo-EM half map 1 of the ion channel TMEM16A from mouse in presence of calcium ions | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : mTMEM16A with calcium ions bound
| 全体 | 名称: mTMEM16A with calcium ions bound |
|---|---|
| 要素 |
|
-超分子 #1: mTMEM16A with calcium ions bound
| 超分子 | 名称: mTMEM16A with calcium ions bound / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: calcium-activated chloride channel |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 110.916 KDa |
-分子 #1: Anoctamin-1
| 分子 | 名称: Anoctamin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 111.058992 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MRVPEKYSTL PAEDRSVHIV NICAIEDLGY LPSEGTLLNS LSVDPDAECK YGLYFRDGKR KVDYILVYHH KRASGSRTLA RRGLQNDMV LGTRSVRQDQ PLPGKGSPVD AGSPEVPMDY HEDDKRFRRE EYEGNLLEAG LELENDEDTK IHGVGFVKIH A PWHVLCRE ...文字列: MRVPEKYSTL PAEDRSVHIV NICAIEDLGY LPSEGTLLNS LSVDPDAECK YGLYFRDGKR KVDYILVYHH KRASGSRTLA RRGLQNDMV LGTRSVRQDQ PLPGKGSPVD AGSPEVPMDY HEDDKRFRRE EYEGNLLEAG LELENDEDTK IHGVGFVKIH A PWHVLCRE AEFLKLKMPT KKVYHISETR GLLKTINSVL QKITDPIQPK VAEHRPQTTK RLSYPFSREK QHLFDLTDRD SF FDSKTRS TIVYEILKRT TCTKAKYSMG ITSLLANGVY SAAYPLHDGD YEGDNVEFND RKLLYEEWAS YGVFYKYQPI DLV RKYFGE KVGLYFAWLG AYTQMLIPAS IVGVIVFLYG CATVDENIPS MEMCDQRYNI TMCPLCDKTC SYWKMSSACA TARA SHLFD NPATVFFSVF MALWAATFME HWKRKQMRLN YRWDLTGFEE EEEAVKDHPR AEYEARVLEK SLRKESRNKE TDKVK LTWR DRFPAYFTNL VSIIFMIAVT FAIVLGVIIY RISTAAALAM NSSPSVRSNI RVTVTATAVI INLVVIILLD EVYGCI ARW LTKIEVPKTE KSFEERLTFK AFLLKFVNSY TPIFYVAFFK GRFVGRPGDY VYIFRSFRME ECAPGGCLME LCIQLSI IM LGKQLIQNNL FEIGIPKMKK FIRYLKLRRQ SPSDREEYVK RKQRYEVDFN LEPFAGLTPE YMEMIIQFGF VTLFVASF P LAPLFALLNN IIEIRLDAKK FVTELRRPVA IRAKDIGIWY NILRGVGKLA VIINAFVISF TSDFIPRLVY LYMYSQNGT MHGFVNHTLS SFNVSDFQNG TAPNDPLDLG YEVQICRYKD YREPPWSEHK YDISKDFWAV LAARLAFVIV FQNLVMFMSD FVDWVIPDI PKDISQQIHK EKVLMVELFM REEQGKQQLL DTWMEKEKPR DVPCNNHSPT THPEAGDGSP VPSYEYHGDA L UniProtKB: Anoctamin-1 |
-分子 #2: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 2 / コピー数: 4 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 構成要素 - 濃度: 20.0 mM / 構成要素 - 名称: Hepes / 詳細: 20 mM Hepes 150 mM NaCl 0.5 mM CaCl2 <0.12% digitonin |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 288 K / 装置: FEI VITROBOT MARK IV / 詳細: 2 ul sample volume 2-4 sec blotting time. |
| 詳細 | full-length (wild-type isoform ac) deglycosylated mTMEM16A in presence of 0.5mM CaCl2 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - エネルギー下限: -10 eV エネルギーフィルター - エネルギー上限: +10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7420 pixel / デジタル化 - サイズ - 縦: 7676 pixel / デジタル化 - 画像ごとのフレーム数: 1-80 / 撮影したグリッド数: 3 / 実像数: 4343 / 平均露光時間: 10.0 sec. / 平均電子線量: 80.0 e/Å2 詳細: Data were collected in an automated fashion using SerialEM47 on a K2 Summit detector (Gatan). For the dataset in presence of calcium ions, cryo-EM images were collected at a pixel size of 0. ...詳細: Data were collected in an automated fashion using SerialEM47 on a K2 Summit detector (Gatan). For the dataset in presence of calcium ions, cryo-EM images were collected at a pixel size of 0.5375A in super-resolution mode, a defocus range of -0.5 to -3.0 um, an exposure time of 10 sec and a sub-frame exposure time of 125 ms (80 frames) with an approximate electron dose of 1 e-/A2/frame. An extensive effort was made for this dataset to screen different areas within the grid and only regions that provided an estimated resolution of the CTF fit of better than 4A were selected for data collection. The total accumulated dose on the specimen level was approximately 80 e-/A2. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.5 µm / 倍率(補正後): 46511 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 46511 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-5oyb: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)