+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
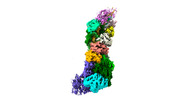
| タイトル | Cryo-EM structure of CRISPR-Csm effector complex from Mycobacterium canettii | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Csm effector complex / Mycobacterium canettii / Cryo-EM structure / Csm1 / Csm2 / Csm3 / Csm4 / Csm5 / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 | : / : / : / : / :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.73 Å | |||||||||
 データ登録者 データ登録者 | Huo Y / Ma X / Jiang T | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Int J Biol Macromol / 年: 2024 ジャーナル: Int J Biol Macromol / 年: 2024タイトル: Type-III-A structure of mycobacteria CRISPR-Csm complexes involving atypical crRNAs. 著者: Hongtai Zhang / Mingmin Shi / Xiaoli Ma / Mengxi Liu / Nenhan Wang / Qiuhua Lu / Zekai Li / Yanfeng Zhao / Hongshen Zhao / Hong Chen / Huizhi Zhang / Tao Jiang / Songying Ouyang / Yangao Huo / Lijun Bi /  要旨: Tuberculosis (TB), a leading cause of mortality globally, is a chronic infectious disease caused by Mycobacterium tuberculosis that primarily infiltrates the lung. The mature crRNAs in M. ...Tuberculosis (TB), a leading cause of mortality globally, is a chronic infectious disease caused by Mycobacterium tuberculosis that primarily infiltrates the lung. The mature crRNAs in M. tuberculosis transcribed from the Clustered Regularly Interspaced Short Palindromic Repeats (CRISPR) locus exhibit an atypical structure featured with 5' and 3' repeat tags at both ends of the intact crRNA, in contrast to typical Type-III-A crRNAs that possess 5' repeat tags and partial crRNA sequences. However, this structural peculiarity particularly concerning the specific binding characteristics of the 3' repeat end within the mature crRNA within the Csm complex, has not been comprehensively elucidated. Here, our Mycobacteria CRISPR-Csm complexes structure represents the largest Csm complex reported to date. It incorporates an atypical Type-III-A CRISPR RNA (crRNA) (46 nt) with 5' 8-nt and 3' 4-nt repeat sequences in the stoichiometry of Mycobacteria Csm12345 The PAM-independent single-stranded RNAs (ssRNAs) are the most suitable substrate for the Csm complex. The 3'-repeat end trimming of mature crRNA was not necessary for its cleavage activity in Type-III-A Csm complex. Our work broadens our understanding of the Type-III-A Csm complex and identifies another mature crRNA processing mechanism in the Type-III-A CRISPR-Cas system based on structural biology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37499.map.gz emd_37499.map.gz | 202.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37499-v30.xml emd-37499-v30.xml emd-37499.xml emd-37499.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_37499.png emd_37499.png | 30.3 KB | ||
| Filedesc metadata |  emd-37499.cif.gz emd-37499.cif.gz | 6.8 KB | ||
| その他 |  emd_37499_half_map_1.map.gz emd_37499_half_map_1.map.gz emd_37499_half_map_2.map.gz emd_37499_half_map_2.map.gz | 171.8 MB 171.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37499 http://ftp.pdbj.org/pub/emdb/structures/EMD-37499 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37499 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37499 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37499_validation.pdf.gz emd_37499_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37499_full_validation.pdf.gz emd_37499_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_37499_validation.xml.gz emd_37499_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_37499_validation.cif.gz emd_37499_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37499 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37499 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37499 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37499 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8wfxMC  8x5dC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37499.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37499.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_37499_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
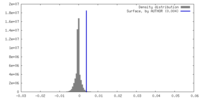
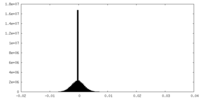
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37499_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
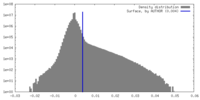
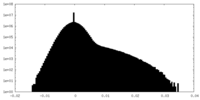
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Csm effector complex
| 全体 | 名称: Csm effector complex |
|---|---|
| 要素 |
|
-超分子 #1: Csm effector complex
| 超分子 | 名称: Csm effector complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
-分子 #1: CRISPR system Cms protein Csm4
| 分子 | 名称: CRISPR system Cms protein Csm4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
| 分子量 | 理論値: 32.26477 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNSRLFRFDV VRTHFGDHGL ESSTIGCPAD TLYSALCVEA LRMGGQQLLD ELVACSTLRL TDLLPYVGPD YLVPKPLKSV RSDSSSLQK KLAKKIGFLP AAQLGSFLDG TADLDDLAAR QTKIGVHAVS AKAAIHNGKK DADPYRVGYF RFEPDAGLWL L ATGSESEL ...文字列: MNSRLFRFDV VRTHFGDHGL ESSTIGCPAD TLYSALCVEA LRMGGQQLLD ELVACSTLRL TDLLPYVGPD YLVPKPLKSV RSDSSSLQK KLAKKIGFLP AAQLGSFLDG TADLDDLAAR QTKIGVHAVS AKAAIHNGKK DADPYRVGYF RFEPDAGLWL L ATGSESEL GLLTRLLKGI SALGGERTSG FGAFIPTESE VPAALTPTTA AASLMTLTTS LPTDDELEQA LAGATYRLVK RS GFVASAT YAETPRRKRD IYKLAAGSVF SRPFQGGILD VSLGGNHPVY SYARPLFLAL PESAA UniProtKB: UNIPROTKB: G0TFC1 |
-分子 #3: CRISPR system single-strand-specific deoxyribonuclease Cas10/Csm1...
| 分子 | 名称: CRISPR system single-strand-specific deoxyribonuclease Cas10/Csm1 (subtype III-A) タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
| 分子量 | 理論値: 91.957586 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIPALIETVI GCLLHDIGKP VQRAALGYRG RHSAIGRAFV KKIWLRDGRN PSEFADEVYE PDIEVFDRRI LDAISYHHSS ALRTAAENG RLAADAPAYV AYIADNIAAG TDRRKADSDD GQGASTWDSD TPLYSVFNRF GADTANLTFA PEMLDDREPM N IPSARRIE ...文字列: MIPALIETVI GCLLHDIGKP VQRAALGYRG RHSAIGRAFV KKIWLRDGRN PSEFADEVYE PDIEVFDRRI LDAISYHHSS ALRTAAENG RLAADAPAYV AYIADNIAAG TDRRKADSDD GQGASTWDSD TPLYSVFNRF GADTANLTFA PEMLDDREPM N IPSARRIE FDKYRYTEIV NKLEAVLVDL ECSDTYLASL LNVLEATLSF VPSSTDASEV VDVSLFDHLK LTGALGACIW HY LQATGQS DFKSALFDKQ DTFYNEKAFL LTTFDVSGIQ DFIYTIHSSG AAKMLRARSF YLEMLTEHLI DELLARVGLS RAN LNYSGG GHAYLLLPNT EFARNSLEEF EREANEWLLE NFATWLFIAT GSVPLAANDL MRRPNESGPQ AHDRALRYSG LYRE LSEQL SAKKLARYSA DQLRELNSSD HDGQKGDREC SVCHTVNRTI KTINDLLLCS LCQALTAASQ QIQSESRRFL LISEE SSDG LPLPFGATLT FCSESGAKQA LQQPQTRRLY AKNKFFAGES LGTGLWVGDY VAQMEFGDYV KRASGIARLG VLRLDV DNL GQAFTHGFMK QGNGKFNTIS RTAAFSRMLS LFFRQHINYV LKHPKLRPLT GDDPERSRRA TIIYSGGDDV FVVGAWD DV IEFGIELRER FHEFTQGKLT VSAGIGMFPD KYPISVMARE VGDLEGAAKS LPGKNGVALF DPEFTFRWDE LLSKVIEE K YRHIADYFRC NEERGMAFIY KLLELLDQRG DRITEARWVY FLMRMRGPTD DTTRFQQFAN RLHQWFQDPS DAKQLKTAL HLYIYRNRKE ESE UniProtKB: UNIPROTKB: G0TFC4 |
-分子 #4: CRISPR system Cms protein Csm5
| 分子 | 名称: CRISPR system Cms protein Csm5 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
| 分子量 | 理論値: 42.167949 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSQYLKPFEL TLRCLGPVFI GSGEKRTPKE YVASTSMVYF PDMERLYADV AAQGKSESFE EFMMNTGKAQ PDERFNEWIA ENGVKVSPK NHGGYGVKIG SIVPGRAHRG RDGQMIQEQR QLNDIHSFIK DVLGNPYVPG SSVKGMLRSI YLQSLVHQRT A QPVRVPGH ...文字列: MSQYLKPFEL TLRCLGPVFI GSGEKRTPKE YVASTSMVYF PDMERLYADV AAQGKSESFE EFMMNTGKAQ PDERFNEWIA ENGVKVSPK NHGGYGVKIG SIVPGRAHRG RDGQMIQEQR QLNDIHSFIK DVLGNPYVPG SSVKGMLRSI YLQSLVHQRT A QPVRVPGH QTREHRQYGE RFERKELRKS GRPNTRPQDA VNDLFQAIRV TDSPGLRTSD LLICQKMDVN VHGKPDGLPL FR ECLAPGT SISLRVVVDT SPTARGGWPA GERFLETLSD TVAFVNKARY AEYAAKYWDD DPQFGPIVYL GGGAGYRSKT FVT QQDDMA KVLDAQFPKI KHVAKTRDLG VSPLVLKLTK IGDKYYEMGQ CELSIRRAE UniProtKB: UNIPROTKB: G0TFC0 |
-分子 #5: CRISPR system Cms protein Csm2
| 分子 | 名称: CRISPR system Cms protein Csm2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
| 分子量 | 理論値: 15.006156 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSVIQDDYVK QAEQVIRGLP KKNGDFELTT TQLRVLLSLT AQLFDEAQLS SDQNLSPALR DKVQYLRVRF VYQAGREKAV RVFVERAGL LDELAQIGDS RDRLLKFCHY MEALVAYKKF LDPKETSKET E UniProtKB: UNIPROTKB: G0TFC3 |
-分子 #6: CRISPR system Cms endoribonuclease Csm3
| 分子 | 名称: CRISPR system Cms endoribonuclease Csm3 / タイプ: protein_or_peptide / ID: 6 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
| 分子量 | 理論値: 25.952537 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MITGYAKIEI TGTITVVTGL HIGAGDGFSA IGAVDKPVVR DPLTKWPMIP GTSLKGKVRT LLSRQYGADT ETFYRKPNDD PAQIRRLFG DTKEYKTGRL VFRDTKLTNK DDLEARGAKT LTEVKFENAI NRVTAKANPR QMERVIPGSE FAFSLVYEVS F GTPDEEQK ...文字列: MITGYAKIEI TGTITVVTGL HIGAGDGFSA IGAVDKPVVR DPLTKWPMIP GTSLKGKVRT LLSRQYGADT ETFYRKPNDD PAQIRRLFG DTKEYKTGRL VFRDTKLTNK DDLEARGAKT LTEVKFENAI NRVTAKANPR QMERVIPGSE FAFSLVYEVS F GTPDEEQK ASLPSSDEII EDFNAIARGL KLLELDYLGG SGTRGYGQVK FSDLKARAAV GTLDRSLLEM LNHELAAV UniProtKB: UNIPROTKB: G0TFC2 |
-分子 #2: RNA (50-MER)
| 分子 | 名称: RNA (50-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium canettii (バクテリア) Mycobacterium canettii (バクテリア) |
| 分子量 | 理論値: 16.001554 KDa |
| 配列 | 文字列: ACGGAAACUU AAAACCGUGU UGCACUGCAA CCCGGAAUUC UUGCACGUCG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.73 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 110023 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)