+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural organization of the palisade layer in isolated vaccinia virus cores | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | poxvirus / assembly / core / palisade layer / A10 / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Poxvirus P4A / Poxvirus P4A protein / virion component / structural molecule activity / Major core protein OPG136 precursor 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Vaccinia virus (strain Western Reserve) (ウイルス) / Vaccinia virus (strain Western Reserve) (ウイルス) /  Vaccinia virus Western Reserve (ウイルス) Vaccinia virus Western Reserve (ウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Qu X / Duan M / Shi X / Liu S / Shi Y / Gao GF | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-ET reveals A10 protein as a major component of the poxvirus palisade layer 著者: Liu Y / Qu X / Duan M / Shi X / Liu S / Shi Y / Gao GF | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_37456.map.gz emd_37456.map.gz | 1016 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-37456-v30.xml emd-37456-v30.xml emd-37456.xml emd-37456.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_37456_fsc.xml emd_37456_fsc.xml | 4.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_37456.png emd_37456.png | 151.8 KB | ||
| マスクデータ |  emd_37456_msk_1.map emd_37456_msk_1.map | 6.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-37456.cif.gz emd-37456.cif.gz | 6.4 KB | ||
| その他 |  emd_37456_half_map_1.map.gz emd_37456_half_map_1.map.gz emd_37456_half_map_2.map.gz emd_37456_half_map_2.map.gz | 3.2 MB 3.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-37456 http://ftp.pdbj.org/pub/emdb/structures/EMD-37456 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37456 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-37456 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_37456_validation.pdf.gz emd_37456_validation.pdf.gz | 691.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_37456_full_validation.pdf.gz emd_37456_full_validation.pdf.gz | 691 KB | 表示 | |
| XML形式データ |  emd_37456_validation.xml.gz emd_37456_validation.xml.gz | 8.2 KB | 表示 | |
| CIF形式データ |  emd_37456_validation.cif.gz emd_37456_validation.cif.gz | 11.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37456 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37456 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37456 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-37456 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8wd7MC  8wdcC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_37456.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_37456.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4 Å | ||||||||||||||||||||||||||||||||||||
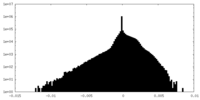
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_37456_msk_1.map emd_37456_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
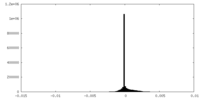
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_37456_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_37456_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vaccinia virus Western Reserve
| 全体 | 名称:  Vaccinia virus Western Reserve (ウイルス) Vaccinia virus Western Reserve (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Vaccinia virus Western Reserve
| 超分子 | 名称: Vaccinia virus Western Reserve / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Vaccinia virus was grown in HeLa cells and purified. The resultant mature virions were treated with DTT and NP40 and then purified to yield viral cores. NCBI-ID: 696871 / 生物種: Vaccinia virus Western Reserve / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Core protein OPG136
| 分子 | 名称: Core protein OPG136 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vaccinia virus (strain Western Reserve) (ウイルス) Vaccinia virus (strain Western Reserve) (ウイルス) |
| 分子量 | 理論値: 71.073477 KDa |
| 配列 | 文字列: MMPIKSIVTL DQLEDSEYLF RIVSTVLPHL CLDYKVCDQL KTTFVHPFDI LLNNSLGSVT KQDELQAAIS KLGINYLIDT TSRELKLFN VTLNAGNIDI INTPINISSE TNPIINTHSF YDLPPFTQHL LNIRLTDTEY RARFIGGYIK PDGSDSMDVL A EKKYPDLN ...文字列: MMPIKSIVTL DQLEDSEYLF RIVSTVLPHL CLDYKVCDQL KTTFVHPFDI LLNNSLGSVT KQDELQAAIS KLGINYLIDT TSRELKLFN VTLNAGNIDI INTPINISSE TNPIINTHSF YDLPPFTQHL LNIRLTDTEY RARFIGGYIK PDGSDSMDVL A EKKYPDLN FDNTYLFNIL YKDVINAPIK EFKAKIVNGV LSRQDFDNLI GVRQYITIQD RPRFDDAYNI ADAARHYGVN LN TLPLPNV DLTTMPTYKH LIMFEQYFIY TYDRVDIYYN GNKMLFDDEI INFTISMRYQ SLIPRLVDFF PDIPVNNNIV LHT RDPQNA AVNVTVALPN VQFVDINRNN KFFINFFNLL AKEQRSTAIK VTKSMFWDGM DYEEYKSKNL QDMMFINSTC YVFG LYNHN NTTYCSILSD IISAEKTPIR VCLLPRVVGG KTVTNLISET LKSISSMTIR EFPRKDKSIM HIGLSETGFM RFFQL LRLM ADKPHETAIK EVVMAYVGIK LGDKGSPYYI RKESYQDFIY LLFASMGFKV TTRRSIMGSN NISIISIRPR VTKQYI VAT LMKTSCSKNE AEKLITSAFD LLNFMVSVSD FRDYQSYRQY RNYCPRYFYA G UniProtKB: Major core protein OPG136 precursor |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 9 / 詳細: 1mM Tris pH 9 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Vaccinia virus was grown in HeLa cells and purified. The resultant mature virions were treated with DTT and NP40 and then purified to yield viral cores. Isolated cores were used as a specimen. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Microscope was equipped with a Cs corrector 色収差補正装置: Microscope was equipped with a Cc corrector エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 40 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 2.9 e/Å2 / 詳細: Dose rate 5.2 e-/pixel/s, EER (241 frames/sec) |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.0 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.6 µm / 倍率(公称値): 53000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8wd7: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)