+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cryo-EM structure of insect gustatory receptor Gr43a from Drosophila melanogaster | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | gustatory receptor / Gr43a / ligand-gated ion channel / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sweet taste receptor activity / ionotropic sweet taste receptor activity / male courtship behavior / regulation of appetite / taste receptor activity / chemosensory behavior / adult feeding behavior / sensory perception of sweet taste / regulation of feeding behavior / cellular response to fructose stimulus ...sweet taste receptor activity / ionotropic sweet taste receptor activity / male courtship behavior / regulation of appetite / taste receptor activity / chemosensory behavior / adult feeding behavior / sensory perception of sweet taste / regulation of feeding behavior / cellular response to fructose stimulus / ligand-gated monoatomic cation channel activity / monoatomic cation transmembrane transport / sensory perception of taste / axon / neuronal cell body / dendrite / signal transduction / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Ma D / Guo J | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Structural basis for sugar perception by gustatory receptors. 著者: Demin Ma / Meiqin Hu / Xiaotong Yang / Qiang Liu / Fan Ye / Weijie Cai / Yong Wang / Ximing Xu / Shenghai Chang / Ruiying Wang / Wei Yang / Sheng Ye / Nannan Su / Minrui Fan / Haoxing Xu / Jiangtao Guo /   要旨: Insects rely on a family of seven transmembrane proteins called gustatory receptors (GRs) to encode different taste modalities, such as sweet and bitter. We report structures of sweet taste ...Insects rely on a family of seven transmembrane proteins called gustatory receptors (GRs) to encode different taste modalities, such as sweet and bitter. We report structures of sweet taste receptors GR43a and GR64a in the apo and sugar-bound states. Both GRs form tetrameric sugar-gated cation channels composed of one central pore domain (PD) and four peripheral ligand-binding domains (LBDs). Whereas GR43a is specifically activated by the monosaccharide fructose that binds to a narrow pocket in LBDs, disaccharides sucrose and maltose selectively activate GR64a by binding to a larger and flatter pocket in LBDs. Sugar binding to LBDs induces local conformational changes, which are subsequently transferred to the PD to cause channel opening. Our studies reveal a structural basis for sugar recognition and activation of GRs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36410.map.gz emd_36410.map.gz | 47.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36410-v30.xml emd-36410-v30.xml emd-36410.xml emd-36410.xml | 14.2 KB 14.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_36410.png emd_36410.png | 64.4 KB | ||
| Filedesc metadata |  emd-36410.cif.gz emd-36410.cif.gz | 5.5 KB | ||
| その他 |  emd_36410_half_map_1.map.gz emd_36410_half_map_1.map.gz emd_36410_half_map_2.map.gz emd_36410_half_map_2.map.gz | 44 MB 44.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36410 http://ftp.pdbj.org/pub/emdb/structures/EMD-36410 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36410 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36410 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36410_validation.pdf.gz emd_36410_validation.pdf.gz | 874.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36410_full_validation.pdf.gz emd_36410_full_validation.pdf.gz | 874.1 KB | 表示 | |
| XML形式データ |  emd_36410_validation.xml.gz emd_36410_validation.xml.gz | 11.7 KB | 表示 | |
| CIF形式データ |  emd_36410_validation.cif.gz emd_36410_validation.cif.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36410 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36410 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36410 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36410 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8jm9MC  8jmaC  8jmeC  8jmhC  8jmiC  8x82C  8x83C  8x84C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36410.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36410.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
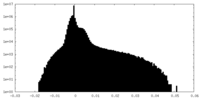
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_36410_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
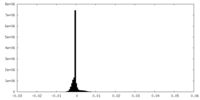
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36410_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
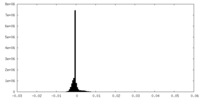
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : gustatory receptor Gr43a
| 全体 | 名称: gustatory receptor Gr43a |
|---|---|
| 要素 |
|
-超分子 #1: gustatory receptor Gr43a
| 超分子 | 名称: gustatory receptor Gr43a / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Gustatory receptor for sugar taste 43a
| 分子 | 名称: Gustatory receptor for sugar taste 43a / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.309391 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEISQPSIGI FYISKVLALA PYATVRNSKG RVEIGRSWLF TVYSATLTVV MVFLTYRGLL FDANSEIPVR MKSATSKVVT ALDVSVVVM AIVSGVYCGL FSLNDTLELN DRLNKIDNTL NAYNNFRRDR WRALGMAAVS LLAISILVGL DVGTWMRIAQ D MNIAQSDT ...文字列: MEISQPSIGI FYISKVLALA PYATVRNSKG RVEIGRSWLF TVYSATLTVV MVFLTYRGLL FDANSEIPVR MKSATSKVVT ALDVSVVVM AIVSGVYCGL FSLNDTLELN DRLNKIDNTL NAYNNFRRDR WRALGMAAVS LLAISILVGL DVGTWMRIAQ D MNIAQSDT ELNVHWYIPF YSLYFILTGL QVNIANTAYG LGRRFGRLNR MLSSSFLAEN NATSAIKPQK VSTVKNVSVN RP AMPSALH ASLTKLNGET LPSEAAAKNK GLLLKSLADS HESLGKCVHL LSNSFGIAVL FILVSCLLHL VATAYFLFLE LLS KRDNGY LWVQMLWICF HFLRLLMVVE PCHLAARESR KTIQIVCEIE RKVHEPILAE AVKKFWQQLL VVDADFSACG LCRV NRTIL TSFASAIATY LVILIQFQRT NGLEGGSSGG WSHPQFEK UniProtKB: Gustatory receptor for sugar taste 43a |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 263979 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)