+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Neck of DT57C bacteriophage in the full state | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Neck / Portal / T5 / VIRUS / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 | Bacteriophage/Gene transfer agent portal protein / Phage portal protein / viral capsid / Head completion protein / Portal protein / Tape measure protein / Tail terminator protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Escherichia phage DT57C (ファージ) / Escherichia phage DT57C (ファージ) /  Tequintavirus DT57C (ウイルス) Tequintavirus DT57C (ウイルス) | |||||||||||||||
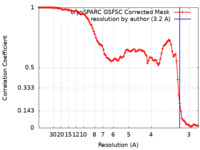
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Ayala R / Moiseenko AV / Chen TH / Kulikov EE / Golomidova AK / Orekhov PS / Street MA / Sokolova OS / Letarov AV / Wolf M | |||||||||||||||
| 資金援助 |  ロシア, ロシア,  日本, 4件 日本, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Nearly complete structure of bacteriophage DT57C reveals architecture of head-to-tail interface and lateral tail fibers. 著者: Rafael Ayala / Andrey V Moiseenko / Ting-Hua Chen / Eugene E Kulikov / Alla K Golomidova / Philipp S Orekhov / Maya A Street / Olga S Sokolova / Andrey V Letarov / Matthias Wolf /     要旨: The T5 family of viruses are tailed bacteriophages characterized by a long non-contractile tail. The bacteriophage DT57C is closely related to the paradigmal T5 phage, though it recognizes a ...The T5 family of viruses are tailed bacteriophages characterized by a long non-contractile tail. The bacteriophage DT57C is closely related to the paradigmal T5 phage, though it recognizes a different receptor (BtuB) and features highly divergent lateral tail fibers (LTF). Considerable portions of T5-like phages remain structurally uncharacterized. Here, we present the structure of DT57C determined by cryo-EM, and an atomic model of the virus, which was further explored using all-atom molecular dynamics simulations. The structure revealed a unique way of LTF attachment assisted by a dodecameric collar protein LtfC, and an unusual composition of the phage neck constructed of three protein rings. The tape measure protein (TMP) is organized within the tail tube in a three-stranded parallel α-helical coiled coil which makes direct contact with the genomic DNA. The presence of the C-terminal fragment of the TMP that remains within the tail tip suggests that the tail tip complex returns to its original state after DNA ejection. Our results provide a complete atomic structure of a T5-like phage, provide insights into the process of DNA ejection as well as a structural basis for the design of engineered phages and future mechanistic studies. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34952.map.gz emd_34952.map.gz | 96.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34952-v30.xml emd-34952-v30.xml emd-34952.xml emd-34952.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34952_fsc.xml emd_34952_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34952.png emd_34952.png | 78.8 KB | ||
| マスクデータ |  emd_34952_msk_1.map emd_34952_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34952.cif.gz emd-34952.cif.gz | 6.6 KB | ||
| その他 |  emd_34952_half_map_1.map.gz emd_34952_half_map_1.map.gz emd_34952_half_map_2.map.gz emd_34952_half_map_2.map.gz | 96.1 MB 96.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34952 http://ftp.pdbj.org/pub/emdb/structures/EMD-34952 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34952 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34952 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34952_validation.pdf.gz emd_34952_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34952_full_validation.pdf.gz emd_34952_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_34952_validation.xml.gz emd_34952_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_34952_validation.cif.gz emd_34952_validation.cif.gz | 22.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34952 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34952 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34952 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34952 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34952.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34952.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34952_msk_1.map emd_34952_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34952_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34952_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tequintavirus DT57C
| 全体 | 名称:  Tequintavirus DT57C (ウイルス) Tequintavirus DT57C (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Tequintavirus DT57C
| 超分子 | 名称: Tequintavirus DT57C / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 1567006 / 生物種: Tequintavirus DT57C / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage DT57C (ファージ) Escherichia phage DT57C (ファージ) |
| 分子量 | 理論値: 45.408574 KDa |
| 配列 | 文字列: MGFKSWITEK LNPGQRIIRD MEPVSHRTNR KPFTTGQAYS KIEILNRTAN MVIDSAAECS YTVGDKYNIV TYANGVKTKT LDTLLNVRP NPFMDISTFR RLVVTDLLFE GCAYIYWDGT SLYHVPAALM QVEADANKFI KKFIFNNQIN YRVDEIIFIK D NSYVCGTN ...文字列: MGFKSWITEK LNPGQRIIRD MEPVSHRTNR KPFTTGQAYS KIEILNRTAN MVIDSAAECS YTVGDKYNIV TYANGVKTKT LDTLLNVRP NPFMDISTFR RLVVTDLLFE GCAYIYWDGT SLYHVPAALM QVEADANKFI KKFIFNNQIN YRVDEIIFIK D NSYVCGTN SQISGQSRVA TVIDSLEKRS KMLNFKEKFL DNGTVIGLIL ETDEILNKKL RERKQEELQL DYNPSTGQSS VL ILDGGMK AKPYSQISSF KDLDFKEDIA GFNKSICLAF GVPQVLIDGG NNANIRPNIE LFYYMTIIPM LNKLTSSLTF FFG YKITPN TKEVAALTPD KEAEAKHLTS LVNNGIMTGN EARLELNLEP LDDEQMNRIR IPANVAGSAT GVSGQEGGRP QGST EGDKE UniProtKB: Portal protein |
-分子 #2: Head completion protein
| 分子 | 名称: Head completion protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage DT57C (ファージ) Escherichia phage DT57C (ファージ) |
| 分子量 | 理論値: 19.2039 KDa |
| 配列 | 文字列: MQIITAEDYR LYGGLKRPEL ESGVEMMITA ANALITSLLG MDDADAVDQL INTKPTRKKY FLSSPSATSV TKMTINDKEI DPEQYKLYS DGVILLKFSP PEGYMDVEYT QGGFNPIPED LKLAACMLVD HWHKQDYRQA KTIGGETVTF NNTKSGIPEH I RTIIEVYR RV UniProtKB: Head completion protein |
-分子 #3: Tail terminator protein
| 分子 | 名称: Tail terminator protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage DT57C (ファージ) Escherichia phage DT57C (ファージ) |
| 分子量 | 理論値: 18.317475 KDa |
| 配列 | 文字列: MDHRTSIAQA LVDRIAQQMD GSQPDEYFNN LYGNVSRQTY KFEEIREFPY VAVHIGTETG QYLPSGQQWM FLELPILVYD KEKTDIQEQ LEKLVADIKT VIDTGGNLEY TVSKPNGSTF PCEATDMSIT SVSTDEGLLA PYGLAEINVT VRYQPPRRSL R R UniProtKB: Tail terminator protein |
-分子 #4: Tape measure protein
| 分子 | 名称: Tape measure protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage DT57C (ファージ) Escherichia phage DT57C (ファージ) |
| 分子量 | 理論値: 132.626406 KDa |
| 配列 | 文字列: MTDKLIRELL IDVKQKGATR TAKSIENVSD ALENAAAASE LTNEQLGKMP KTLYSIERAA DRAAKSLTKM QASRGMAGIT KSIDSIGAK LDDLSIAMIE VADKLEAGFD GVSRSVKVMG NDVAAATEKV QDRLYDTNRV LGGTARGFND TAGAAGRASR A IGNTSGSA ...文字列: MTDKLIRELL IDVKQKGATR TAKSIENVSD ALENAAAASE LTNEQLGKMP KTLYSIERAA DRAAKSLTKM QASRGMAGIT KSIDSIGAK LDDLSIAMIE VADKLEAGFD GVSRSVKVMG NDVAAATEKV QDRLYDTNRV LGGTARGFND TAGAAGRASR A IGNTSGSA RGATRDFAAM AKVGGGLPLL YAAIASNIFV LQSAFEQLKL GDQLNRLEEF GVIVGTQTGT PVQSLARSLQ EA AGYAISF EEAMRQASSA SAYGFDAEQL NKFGLVARRA AAVLGVDMTD ALNRVIKGVS KQEIELLDEL GVTIRLNDAY ADY VKQLNA ANTGITYNIN SLTTFQKQQA YANAVIAEST KRFGYLDEVL RATPWEQFAA NADAALRTIQ QAAAKYLGPV IDAI NTVFY TSQASISAEA ARAQEQTNKQ IDPTNVGAVA LSLSASEEGY NKALDMYKES LDKRNKLKSE FDKRMEQADF YTKLA IRQV GEGIPAGLAT AGASEANKKF VEETAAMGLQ VARLDKEVTD STENLNAWKS AYQAAGAAAA KANPEFQKQI NLQRDT TDP GAVYDFNSTV LKGLTEQQKA YNQTKKTASD LANDIQNVAQ NTDTAAKTSA TLADAIKNIE SLSLGTGKSA DEYVKNL NL GYNTLSEMKT ASQALSEYVK LTGNETKNQL AVQQKIADVY NQTKDKEKAQ EAGRRLELQQ LEEQEAALRR VLQTNQGN K AVEREIEKIQ LEKIKLTNQG MEAQKKVKDY TDKILGVDRE IALLNNRTMT DTQYRLAQLN LELTIEKEKY EWYTKQADK QKEAEQSRRA QAQIEREIWK FHQDQQAEMT SKRQEAFENT LTSMFPLAGE MQKMEMQLDF YTQMKELTKD NANEQMRWNA EIAKTRAQM SALTAQRNAQ MQSSVGSSLG AVYTPTTGLS GEDKKFADMG NQLASYDQAI SKLSELNSEA TAVAQSMGNL A NAMIQFSQ GSLDTTSTIA VGMQTVASMI QYSTSQQVSA IDQAIAAEQK RDGKSEASKA KLKKLEAEKL KIQQDAAKKQ II IQTAVAV MQAATAVPYP FSIPLMIAAG LAGALALAQA SSASGMSSIG DSGADTASYL TLGERQKNID VSMSANAGEL SYV RGDKGI GNANSFVPRA EGGNMYPGVS YQMGEHGTEV VTPMVPMKAT PNDELKNSSN STAGRPIILN ISAMDAASFR EFAS SNSGA LRDAVELALN ENGASLKTLG NS UniProtKB: Tape measure protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 67.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






























 Z
Z Y
Y X
X