+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3445 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-electron microscopy reconstruction of microtubule-bound S.pombe kinesin-5 motor domain in the AMPPNP state | |||||||||
 マップデータ マップデータ | Cryo-electron microscopy of microtubule-bound S. pombe kinesin-5 motor domain in the AMPPNP state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | microtubule-bound S.pombe kinesin-5 / motor domain / AMPPNP bound state / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitotic spindle formation (spindle phase one) / mitotic spindle elongation (spindle phase three) / Kinesins / initial mitotic spindle pole body separation / microtubule plus-end directed mitotic chromosome migration / meiotic spindle assembly / meiotic spindle pole / mitotic spindle midzone / mitotic spindle pole body / mitotic spindle midzone assembly ...mitotic spindle formation (spindle phase one) / mitotic spindle elongation (spindle phase three) / Kinesins / initial mitotic spindle pole body separation / microtubule plus-end directed mitotic chromosome migration / meiotic spindle assembly / meiotic spindle pole / mitotic spindle midzone / mitotic spindle pole body / mitotic spindle midzone assembly / spindle elongation / polar microtubule / plus-end-directed microtubule motor activity / minus-end-directed microtubule motor activity / meiotic spindle / positive regulation of axon guidance / microtubule associated complex / microtubule motor activity / mitotic spindle assembly / microtubule-based process / spindle microtubule / kinetochore / structural constituent of cytoskeleton / microtubule cytoskeleton organization / mitotic spindle / mitotic cell cycle / microtubule cytoskeleton / nervous system development / microtubule binding / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / microtubule / hydrolase activity / protein heterodimerization activity / cell division / GTPase activity / GTP binding / ATP hydrolysis activity / ATP binding / nucleus / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.3 Å | |||||||||
 データ登録者 データ登録者 | Goulet A / Moores CA | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2016 ジャーナル: Proc Natl Acad Sci U S A / 年: 2016タイトル: Schizosaccharomyces pombe kinesin-5 switches direction using a steric blocking mechanism. 著者: Mishan Britto / Adeline Goulet / Syeda Rizvi / Ottilie von Loeffelholz / Carolyn A Moores / Robert A Cross /  要旨: Cut7, the sole kinesin-5 in Schizosaccharomyces pombe, is essential for mitosis. Like other yeast kinesin-5 motors, Cut7 can reverse its stepping direction, by mechanisms that are currently unclear. ...Cut7, the sole kinesin-5 in Schizosaccharomyces pombe, is essential for mitosis. Like other yeast kinesin-5 motors, Cut7 can reverse its stepping direction, by mechanisms that are currently unclear. Here we show that for full-length Cut7, the key determinant of stepping direction is the degree of motor crowding on the microtubule lattice, with greater crowding converting the motor from minus end-directed to plus end-directed stepping. To explain how high Cut7 occupancy causes this reversal, we postulate a simple proximity sensing mechanism that operates via steric blocking. We propose that the minus end-directed stepping action of Cut7 is selectively inhibited by collisions with neighbors under crowded conditions, whereas its plus end-directed action, being less space-hungry, is not. In support of this idea, we show that the direction of Cut7-driven microtubule sliding can be reversed by crowding it with non-Cut7 proteins. Thus, crowding by either dynein microtubule binding domain or Klp2, a kinesin-14, converts Cut7 from net minus end-directed to net plus end-directed stepping. Biochemical assays confirm that the Cut7 N terminus increases Cut7 occupancy by binding directly to microtubules. Direct observation by cryoEM reveals that this occupancy-enhancing N-terminal domain is partially ordered. Overall, our data point to a steric blocking mechanism for directional reversal through which collisions of Cut7 motor domains with their neighbors inhibit their minus end-directed stepping action, but not their plus end-directed stepping action. Our model can potentially reconcile a number of previous, apparently conflicting, observations and proposals for the reversal mechanism of yeast kinesins-5. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3445.map.gz emd_3445.map.gz | 201.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3445-v30.xml emd-3445-v30.xml emd-3445.xml emd-3445.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3445.png emd_3445.png | 192.4 KB | ||
| Filedesc metadata |  emd-3445.cif.gz emd-3445.cif.gz | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3445 http://ftp.pdbj.org/pub/emdb/structures/EMD-3445 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3445 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3445 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3445_validation.pdf.gz emd_3445_validation.pdf.gz | 225.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3445_full_validation.pdf.gz emd_3445_full_validation.pdf.gz | 224.4 KB | 表示 | |
| XML形式データ |  emd_3445_validation.xml.gz emd_3445_validation.xml.gz | 4.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3445 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3445 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3445 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3445 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3445.map.gz / 形式: CCP4 / 大きさ: 489.3 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3445.map.gz / 形式: CCP4 / 大きさ: 489.3 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-electron microscopy of microtubule-bound S. pombe kinesin-5 motor domain in the AMPPNP state | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : microtubule-bound S.pombe kinesin-5 motor domain in the AMPPNP state
| 全体 | 名称: microtubule-bound S.pombe kinesin-5 motor domain in the AMPPNP state |
|---|---|
| 要素 |
|
-超分子 #1: microtubule-bound S.pombe kinesin-5 motor domain in the AMPPNP state
| 超分子 | 名称: microtubule-bound S.pombe kinesin-5 motor domain in the AMPPNP state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 分子量 | 理論値: 140 KDa |
-分子 #1: Tubulin alpha-1D chain
| 分子 | 名称: Tubulin alpha-1D chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 50.107238 KDa |
| 配列 | 文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHP EQLITGKEDA ANNYARGHYT IGKEIIDLVL DRIRKLADQC TGLQGFSVFH SFGGGTGSGF TSLLMERLSV D YGKKSKLE ...文字列: MRECISIHVG QAGVQIGNAC WELYCLEHGI QPDGQMPSDK TIGGGDDSFN TFFSETGAGK HVPRAVFVDL EPTVIDEVRT GTYRQLFHP EQLITGKEDA ANNYARGHYT IGKEIIDLVL DRIRKLADQC TGLQGFSVFH SFGGGTGSGF TSLLMERLSV D YGKKSKLE FSIYPAPQVS TAVVEPYNSI LTTHTTLEHS DCAFMVDNEA IYDICRRNLD IERPTYTNLN RLIGQIVSSI TA SLRFDGA LNVDLTEFQT NLVPYPRGHF PLATYAPVIS AEKAYHEQLS VAEITNACFE PANQMVKCDP RHGKYMACCL LYR GDVVPK DVNAAIATIK TKRTIQFVDW CPTGFKVGIN YEPPTVVPGG DLAKVQRAVC MLSNTTAIAE AWARLDHKFD LMYA KRAFV HWYVGEGMEE GEFSEAREDM AALEKDYEEV GVDSVEGEGE EEGEEY UniProtKB: Tubulin alpha-1D chain |
-分子 #2: Tubulin beta-2B chain
| 分子 | 名称: Tubulin beta-2B chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 49.90777 KDa |
| 配列 | 文字列: MREIVHIQAG QCGNQIGAKF WEVISDEHGI DPTGSYHGDS DLQLERINVY YNEAAGNKYV PRAILVDLEP GTMDSVRSGP FGQIFRPDN FVFGQSGAGN NWAKGHYTEG AELVDSVLDV VRKESESCDC LQGFQLTHSL GGGTGSGMGT LLISKIREEY P DRIMNTFS ...文字列: MREIVHIQAG QCGNQIGAKF WEVISDEHGI DPTGSYHGDS DLQLERINVY YNEAAGNKYV PRAILVDLEP GTMDSVRSGP FGQIFRPDN FVFGQSGAGN NWAKGHYTEG AELVDSVLDV VRKESESCDC LQGFQLTHSL GGGTGSGMGT LLISKIREEY P DRIMNTFS VVPSPKVSDT VVEPYNATLS VHQLVENTDE TYCIDNEALY DICFRTLKLT TPTYGDLNHL VSATMSGVTT CL RFPGQLN ADLRKLAVNM VPFPRLHFFM PGFAPLTSRG SQQYRALTVP ELTQQMFDAK NMMAACDPRH GRYLTVAAVF RGR MSMKEV DEQMLNVQNK NSSYFVEWIP NNVKTAVCDI PPRGLKMSAT FIGNSTAIQE LFKRISEQFT AMFRRKAFLH WYTG EGMDE MEFTEAESNM NDLVSEYQQY QDATADEQGE FEEEEGEDEA UniProtKB: Tubulin beta-2B chain |
-分子 #3: Kinesin-like protein cut7
| 分子 | 名称: Kinesin-like protein cut7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 40.737527 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ALHDENETNI NVVVRVRGRT DQEVRDNSSL AVSTSGAMGA ELAIQSDPSS MLVTKTYAFD KVFGPEADQL MLFENSVAPM LEQVLNGYN CTIFAYGQTG TGKTYTMSGD LSDSDGILSE GAGLIPRALY QLFSSLDNSN QEYAVKCSYY ELYNEEIRDL L VSEELRKP ...文字列: ALHDENETNI NVVVRVRGRT DQEVRDNSSL AVSTSGAMGA ELAIQSDPSS MLVTKTYAFD KVFGPEADQL MLFENSVAPM LEQVLNGYN CTIFAYGQTG TGKTYTMSGD LSDSDGILSE GAGLIPRALY QLFSSLDNSN QEYAVKCSYY ELYNEEIRDL L VSEELRKP ARVFEDTSRR GNVVITGIEE SYIKNAGDGL RLLREGSHRR QVAATKCNDL SSRSHSIFTI TLHRKVSSGM TD ETNSLTI NNNSDDLLRA SKLHMVDLAG SENIGRSGAE NKRARETGMI NQSLLTLGRV INALVEKAHH IPYRESKLTR LLQ DSLGGK TKTSMIVTVS STNTNLEETI STLEYAARAK SIRNKPQNNQ LVF UniProtKB: Kinesin-like protein cut7 |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-分子 #6: GUANOSINE-5'-DIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: GDP |
|---|---|
| 分子量 | 理論値: 443.201 Da |
| Chemical component information |  ChemComp-GDP: |
-分子 #7: TAXOL
| 分子 | 名称: TAXOL / タイプ: ligand / ID: 7 / コピー数: 1 / 式: TA1 |
|---|---|
| 分子量 | 理論値: 853.906 Da |
| Chemical component information | 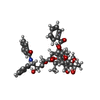 ChemComp-TA1: |
-分子 #8: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 8 / コピー数: 1 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 297 K / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 68000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: GATAN CT3500 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 9.3 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: FREALIGN / 使用した粒子像数: 144300 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: SPIDER |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: FREALIGN |
-原子モデル構築 1
| 詳細 | An initial homology model of S. pombe cut7 kinesin-5 motor domain based on human kinesin-5 structure (PDB 3HQD) was prepared using Modeller. The coordinates of motor bound to an alpha-beta tubulin dimer (PDB 1JFF) were rigidly fitted into the cryo-EM map using Chimera and refined by flexible fitting using Flex-EM. Structural models of loop5 and loop10 were generated using Modeller. The conformation of the neck-linker and the N-terminus were calculated using a conjugate-gradient energy minimization approach. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT 当てはまり具合の基準: Cross-correlation coefficient |
| 得られたモデル |  PDB-5m5i:  PDB-5m5l:  PDB-5m5m:  PDB-5m5n:  PDB-5m5o: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















